【トピックス】
高圧、低温環境に適応する深海由来好圧好冷性微生物のゲノム解析
仲宗根 薫
近大・工
1.はじめに
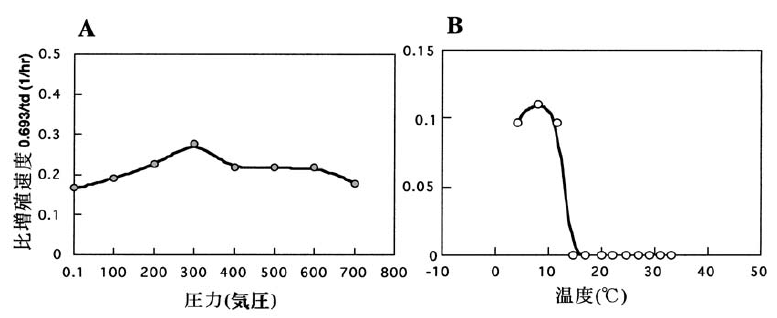
地球表層の70%は海洋でそのほとんどの海域が深度1,000 mを越える「深海環境」である。また深海底の温度は1~3℃であり、恒常的な高水圧下と低温下の環境であるといえる。この様な環境から分離される微生物は、高圧や低温環境に適応した特徴を有するものが多い1)。近年の有人潜水調査船等を利用した調査・研究により、現場環境に適応する深海微生物の多様性、生理学的また生化学的特徴が明らかになってきた2,3)。中でも好圧好冷性微生物は、高圧と低温環境に適応した微生物の典型例である。ここで好圧性とは、大気圧下 (1気圧) よりも高圧下 (200気圧以上) での増殖速度が、また好冷性とは、中温域 (20~40℃) よりも低温域 (0~20℃) での増殖速度が速い性質として定義する。すなわち好圧好冷性微生物は、中温域よりも低温域を、常圧下よりも高圧下を好む微生物の総称である。Shewanella violacea DSS12株は、琉球海溝・深度約5,100 mの底泥から分離された、300気圧および8℃に生育至適を有する、典型的な好圧好冷性細菌である (図1)。本菌株は、大腸菌と近縁な真正細菌であることが明らかにされている4,5)。また大腸菌と系統上非常に近いとはいえ、その好圧性と好冷性は大腸菌と比べ著しく異なっており、これまで本菌株は微生物の高水圧および低温環境への適応機構を研究するためのモデル微生物として扱われてきた。

微生物ゲノムは、病原微生物を中心に解析が進められており、産業上有用な極限環境微生物では、高温環境に適応した (超) 好熱始原菌の解析が活発である。しかしながら、これまで高圧・低温環境に適応する微生物のゲノム解析は皆無であった。本稿では、深海由来好圧好冷性細菌S. violacea DSS12株のこれまでの研究の経緯とその有用性を概観し、本菌株の全ゲノム解析の意義について解説したい。
2.深海微生物研究モデルとしてのS. violacea DSS12株
2.1 細胞分裂機構の解析
圧力および低温は、海洋ではきわめて普遍的物理的因子であり、それを生物がどの様にして認識し、適応しているかを分子レベルで理解することは、基礎研究のみならず、応用研究として深海微生物の遺伝子資源の有効な利用を考える上でも非常に重要である。
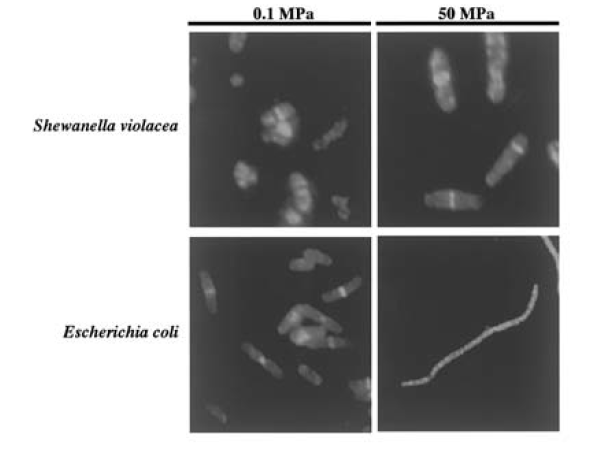
圧力が微生物に与える影響として増殖抑制以外に細胞形態にも変化を与える。図2 は、DSS12株と大腸菌の細胞分裂の様子を、常圧下 (0.1 MPa) と高圧下 (50 MPa) で比較したものである。顕微鏡観察から、大腸菌は高圧力下では、分裂阻害を受け、その形態は長く伸びていく一方、DSS12株では分裂阻害を受けず、その形態に変化はないことが観察される。さらに、細胞分裂時に重要な役割を果たす蛋白質の一つFtsZを免疫蛍光染色法によって顕微鏡観察したところ、中央に集合・配列すべきFtsZは、高圧下では、大腸菌FtsZは規則正しく集合 (リング形成) できないのに対し、DSS12株FtsZは、常圧下と同じように規則正しく配列していることが明らかとなった。この観察結果は、高圧力下での大腸菌の分裂阻害は、FtsZによるリング形成の阻害と密接な関係を有しているものと解釈される6)。すなわち、DSS12株FtsZは、大腸菌FtsZと比べ、高圧力の影響を受けず、相互作用が可能なのである。

2.2 深海微生物の脂肪酸組成の変化
圧力増加と温度低下は、細胞膜の流動性に対し類似の物理的効果を示す。生物の増殖温度の低下、又は増殖圧力の増加に伴い、細胞膜は、流動性を有する状態から流動性のない状態へと可逆的変化を受ける。実際これまでに深海の微生物の高圧および低温環境に対する適応戦略の一つとして、細胞膜内の不飽和脂肪酸の割合を増加させることが観察されている。
Kato & Nogi は、12種類のShewanella 属細菌の脂肪酸組成の変化とその表現型との相関関係について解析を行い、Shewanella属細菌は、Shewanella group1とShewanella group2の亞属 (sub-genus) に分類が可能であることを提唱した7)。この提案は、group1は大量のEPA (20:5 ω3) (全脂質の11~16%) を生産し、group2はEPAを生産しないことから、group1は高圧および低温に適応した細菌であり、一方group2のほとんどが、中温性または圧力感受性であるということを意味している。
脂肪酸組成の圧力変化も、加圧下での特異的な遺伝子発現によって引き起こされたものと解釈され、圧力応答遺伝子の発現調節機構研究の重要性も同時に示唆しているといえよう。
2.3 高圧下における遺伝子発現機構の解析
高圧下における遺伝子発現機構の解析は、80年代後半に初めて報告された8)。米国のBartlettらは深海由来好圧好冷性細菌Photobacterium profundum SS9株より加圧応答する遺伝子ompHをクローニングし、圧力に対する応答現象を解析した8)。また日本においては加藤らが、日本近海深海域より数多くの好圧性細菌を分離し4)、加圧に応答し発現する遺伝子オペロンやプロモーターについて報告している9,10)。
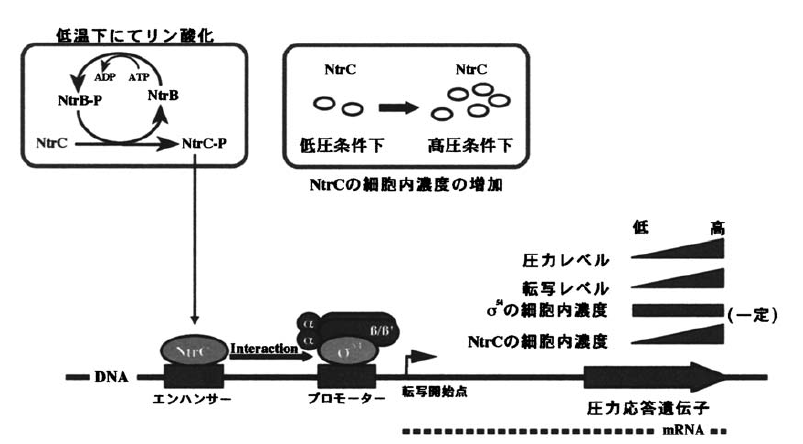
さらに筆者らは、DSS12株より様々な遺伝子をクローニングし、その加圧応答機構の解析を進めてきた。我々はσ因子と転写因子に注目し、σ54因子を活性化する転写因子NtrBおよびNtrCが本菌株の加圧応答の制御に特に重要な役割を担う因子であることを報告してきた11,13)。そこで、σ54因子により制御されていることで知られているglnAオペロン上領域の解析を行ない14)、これら因子群とプロモーター配列の予想される相互作用を加圧応答のモデルとして提案した (図3)15)。

3.深海遺伝子資源確保の戦略的手法としてのゲノム解析
3.1 有用微生物としてのDSS12株
Shewanella 属細菌は、主に水界において温帯域から極域にかけて非常に広範囲に分布しており、嫌気呼吸によってFe (Ⅲ) やMn (Ⅳ) などの金属類を還元するほか,一部の菌種ではCr (Ⅵ) やU (Ⅵ) を還元して無毒化したり,有機塩素化合物の脱ハロゲン化を行う能力を有することから,バイオリメディエーションに有効な細菌として非常に注目を集めている。本菌株はEPAや細胞外プロテアーゼなどを生産することが既に知られており,またShewanella に属することから有毒物質を還元して無毒化する機能を有することも予測される。本菌株は、上記のように、高圧力下および低温下における細胞分裂や遺伝子発現機構など、様々な視点から研究が進められてきており、有用微生物としての側面も持ち合わせている。このような背景から、これまでの遺伝子発現機構に関する知見をゲノム上の分子ネットワークのレベルにまで拡張し、さらに本菌株が生産する有用物質の大量生産法の開発も視野に入れ、複数の研究室によるコンソーシアムにより、本菌株の全ゲノム解析を開始した。
3.2 S. violacea DSS12株の全ゲノム解析
S. violacea DSS12株全ゲノム塩基配列決定に先立ち、第一段階として解析の指標となるゲノムサイズの推定を行った。ゲノムサイズの決定にはパルスフィールドゲル電気泳動を利用した。検出された断片の本数・サイズを解析した後、それらの総和からゲノムサイズを推定したところ、本菌株は約4.8 Mbのゲノムサイズを有することが明らかとなった。また、リボソームRNAオペロン中23SrRNAの塩基配列を認識し切断する制限酵素I-CeuI (5’-TAACTATAACGGTCCTAA/GGTAGCGA-3’) により、本菌株の染色体DNAが12本のDNA断片に分画されたことから、本菌株には12個のリボソームRNAオペロンが存在していることも同時に明らかとなった。原核生物リボソーム合成はrRNAの合成量が律速となっているため、細菌の増殖とrRNAの転写との間には密接な関係があり、増殖速度の増加に伴いリボソーム数も増加する。耐圧菌・好圧菌を含めた深海微生物が高水圧・低温下において良好な増殖速度を維持することは、両環境下においてリボソーム合成が盛んに行われていることを示唆する。深海微生物の現場環境における増殖を理解する上で、今後rRNAやリボソームに関する知見と機能解析は必須なものになるといえよう。
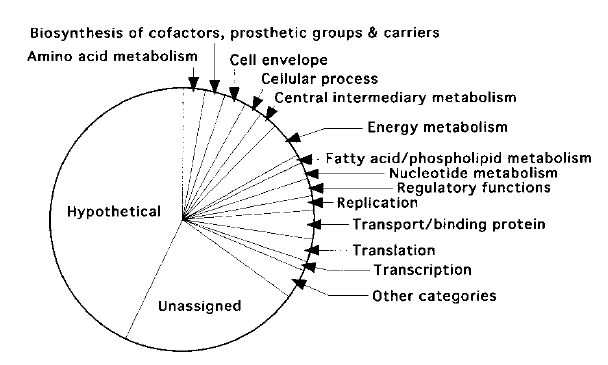
全ゲノムの塩基配列決定には、ホールゲノムショットガン法を採用し、約84,000シークエンスをPhred/Phrap によるアッセンブルと編集を行い約4.8 Mbのコンティグに関して、アノテーションを行った。解析の結果、約4,600のORFの存在が示唆されたが、図4に示すように、系統上近縁な大腸菌遺伝子との構造比較からはその70%近くの機能予測が困難であった。
図4 予測されたORFの分類
本菌株ゲノム中の遺伝子編成に注目すると、リボソーム蛋白質遺伝子およびrRNA オペロン、細胞分裂関連遺伝子群、複製開始起点、等の必須遺伝子群に関しては、基本的にそのオペロン構造の保存性が高いことが確認された。また、ゲノム解析情報の蓄積により水平伝播が多くの微生物の相互作用において普遍的であるということが認識されているが、本菌株においても水平伝播を示す痕跡が幾つか見出されており、現在その情報学的な解析も進めている。
前節で記述した遺伝子発現機構に関してシグマ因子遺伝子の種類と数について検索したところ、本菌株ゲノム中から7種のシグマ因子が見出された。検索された7種のσ因子は、σ70ファミリーrpoD、rpoS、rpoH、σ54ファミリーrpoN、σ70ファミリー/ECF サブファミリーとしてrpoE、新規な2種のECFσ因子 (rpoXおよびrpoYと仮称) であった。これら7種の遺伝子の圧力応答について現在ノーザン解析を行っており、次のステップの解析としてDNAマイクロアレイによって、各々のσ因子が制御する遺伝子群のスクリーニングと圧力応答遺伝子ネットワークの解明が可能になってくるだろう。
有用酵素遺伝子に着目し、相同性検索により本菌株ゲノムを眺めてみると、様々な有用遺伝子の存在が明らかとなった。表1はその一例、バイオリメディエーションに応用可能な酵素遺伝子、工業的に有用な酵素遺伝子、EPAビタミンなど有用な生理活性物質の合成遺伝子など、をまとめたものである。今後は酵素学的な解析とDNAマイクロアレイ、プロテオミクスを組み合わせ、その応用を目指した研究が不可欠である。
表1 相同性解析により明らかとなったDSS12株の持つ有用酵素
4.S. violacea DSS12株のゲノム解析のこれから
今後は、S. violacea DSS12株のゲノム情報を活用し、応用化に向けた機能解析を開始する必要があろう。すなわち1) 本菌株ゲノムのデータベースの構築、2) マイクロアレイ解析およびプロテオーム解析、3) 高圧または低温下において活性至適を有する有用酵素および生理活性物質の探索と大量生産法を目指した基礎的研究、4) Shewanella属のゲノム情報を利用する、低温下におけるバイオリメディエーションの実用化に向けての研究、5) 低温および高圧環境下における微生物の適応機構の解明を目指した学問体系の創造、等が、今後の応用微生物学の発展にとって重要になるのではないだろうか。これは、基礎学問のすそ野を広げることで、より柔軟な応用への道を開くものともいえる。本菌株のゲノム機能解析を行うに当たり、形質転換系は、遺伝子破壊、遺伝子置換、ゲノムの組み込み等を可能にする点で必須なものである。しかしながら、本菌株における形質転換系は未開発であり、現在、エレクトロポレーション法および接合伝達法による形質転換を検討中である。
好冷性を有する微生物ゲノムの解析は、世界で初めての例である。特に、本菌株が生産する低温酵素の利用と今後の本菌株ゲノム情報から発見される多くの低温酵素遺伝子群の情報は、低温性酵素工学の創成を促し得る点で、これまでの微生物ゲノム研究にはなかった視点である。近い将来、低温適応蛋白質の構造プロテオミクスによる、低温適応酵素のもつ立体構造の一般化が可能になる日が来るかも知れない。
謝辞
Shewanella violacea 全ゲノム解析は以下の大学および研究機関のコンソーシアムにより成し遂げられました。解析に参加した多くの方々に、紙面をお借りして深く感謝いたします。コンソーシアム参加機関:慶應義塾大学先端生命科学研究所、奈良先端科学技術大学院大学遺伝子教育センター、海洋科学技術センター海洋生態・環境研究部および極限生物フロンティア、大阪大学遺伝情報実験センター、日本大学文理学部近畿大学工学部 他。
文献
1) 大和田紘一:月刊海洋, 24, 595 (1992).
2) 加藤千明:化学と生物, 34, 129 (1996).
3) 仲宗根薫、加藤千明:月刊海洋/号外, 23, 38 (2000).
4) Kato, C., Sato, T., Horikoshi, K.: Biodiv. Conserv., 4, 1 (1995).
5) Nogi, Y., Kato, C., Horikoshi, K.: Arch. Microbiol., 170, 331 (1998).
6) Ishii, A., Nakasone, K., Sato, T., Wachi, M., Nagai, K., Kato, C.: J. Biochemistry (Tokyo), 132, 183 (2002).
7) Kato, C., Nogi, Y.: FEMS Microbiol. Ecol., 35. 223 (2001).
8) Bartlett, D. H., Wright, M., Yayanos, A. A., Silverman, M.: Nature, 342, 572 (1989).
9) Kato, C., Smorawinska, M., Sato, T., Horikoshi, K.: J. Mar. Biotechnol., 2, 125 (1995).
10) Kato, C., Ikegami, A., Smorawinska, M., Usami, R., Horikoshi, K.: J. Mar. Biotechnol., 5, 210 (1997).
11) Nakasone, K., Ikegami, A., Kato, C., Usami, R., Horikoshi, K.: Extremophiles, 2, 149 (1998).
12) Ikegami, A., Nakasone, K., Fujita, M., Fujii, S., Kato, C., Usami, R., Horikoshi, K.: Biochim. Biophys. Acta., 1491, 315 (2000).
13) Ikegami, A., Nakasone, K., Kato, C., Usami, R., Horikoshi, K.: Biosci. Biotechnol. Biochem., 64, 915 (2000).
14) Ikegami, A., Nakasone, K., Kato, C., Nakamura, Y., Yoshikawa, I., Usami, R., Horikoshi, K.: FEMS Microbiol. Lett., 192, 91 (2000).
15) Nakasone, K., Ikegami, A., Kawano, H., Usami, R., Kato, C., Horikoshi, K.: Extremophiles, 6, 89 (2002).