【トピックス】
サケのミトコンドリアDNAハプロタイプ判別用DNAマイクロアレイの開発
守屋彰悟
日清紡・研究開発センター
1.はじめに
近年、分子生物学的手法の発達と共に遺伝子レベルでサケシロザケOncorhynchus keta) の系群識別を行う研究が進められている1)。系群とは、シロザケなど遡河性魚類の種内において遺伝的に分化した河川あるいは地方ごとの集団をさす。分子生物学的手法の中でもミトコンドリアDNA (mtDNA) の多型を用いた解析が多く行われているが、それらは少数の多型に関してPCR-RFLP法 (Polymerase Chain Reaction-Restriction Fragment Length Polymorphism) を用いた低解像度の解析にとどまっていた2-4)。
北大の阿部らはシロザケのmtDNAコントロール領域の塩基配列解析により多数の多型を検出した5,6)。日本、韓国、ロシア、北米の48の河川から2000を越える個体についてmtDNAコントロール領域5’側前半の塩基配列の解析を行ったところ20箇所の塩基変異サイトとその組み合わせによる30のハプロタイプを見出した。そのハプロタイプの分布は地域により偏りがあることがわかり、さらに海洋のシロザケ混合集団の系群解析を試みたところ従来法と高い相関が得られたことから、このmtDNAハプロタイプ解析は高精度な系群識別法として高い可能性をもつことが示唆された6,7)。しかしながら、シーケンス法による解析には特殊な装置が必要であり、時間と手間、そしてコストもかかるため、系群解析のために必要な多数の個体を短時間で分析することは難しく、シーケンス法に代わる方法が求められている。
多型を検出する際、スラブゲルやキャピラリー電気泳動を用いたシーケンス解析に代わる簡易な方法として、ハイブリダイゼーションを利用したinvader法、TaqMan法、DNAマイクロアレイ法等がある。中でもDNAマイクロアレイ法は一度に多数の多型を検出できる有力な方法として期待されている。
DNAマイクロアレイとは基板上に異なる種類のDNAを整列・固定したものであり、そのようなDNAのうち、標識した検体核酸試料中に含まれる配列と相補的な配列をもつものをハイブリダイゼさせ、水素結合により生じる2本鎖の有無を検出する方法である8-10)。
このように有効な技術として考えられるDNAマイクロアレイ法だが、一枚あたりのコストが高いこと、さらにハイブリダイゼーションによって選択的に得られた2本鎖の検出に蛍光値や電流値の変化を測定するため高価な専用の検出装置が必要であり、シロザケの系群識別を行うことを目的としてDNAマイクロアレイを用いることは不可能と考えられていた。
本稿ではこれらの問題点を解決したDNAマイクロアレイの説明、および、本DNAマイクロアレイを用いたシロザケのmtDNAハプロタイプ判別結果について述べる。
2.DNAマイクロアレイの改良
DNAの固定化方法の開発および判定の目視化によりコストと簡便性の課題をクリアし、実用化のめどをつけたので、まずその内容を以下に示す。
2.1 固定化の改良
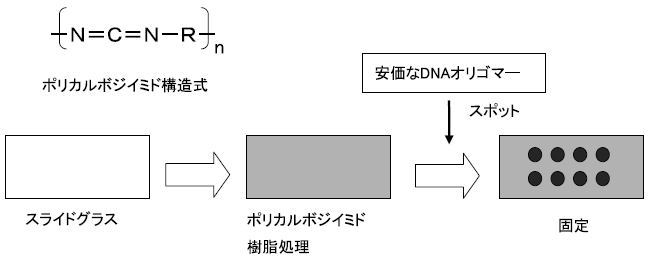
基板上にDNAを固定する場合、いかにしてDNAを安定に固定するかが問題となる。通常基板に樹脂をコートしさらにアミノ化やチオール化等の修飾したDNAを用いて固定するが、この修飾DNAがアレイのコスト高の一因となっていた。そこで、我々はポリカルボジイミド樹脂をコートした基板 (CarboStationR) を開発した。DNAマイクロアレイの製造工程の概略図を図1に示す。ポリカルボジイミド樹脂をコートした基板 (CarboStationR) は高価な修飾無しにDNAを共有結合により固定化でき11)、このため配列や鎖長に関係無くDNAを固定化することが可能となった。
図1 DNAマイクロアレイ製造工程概略
2.2 判定の目視化
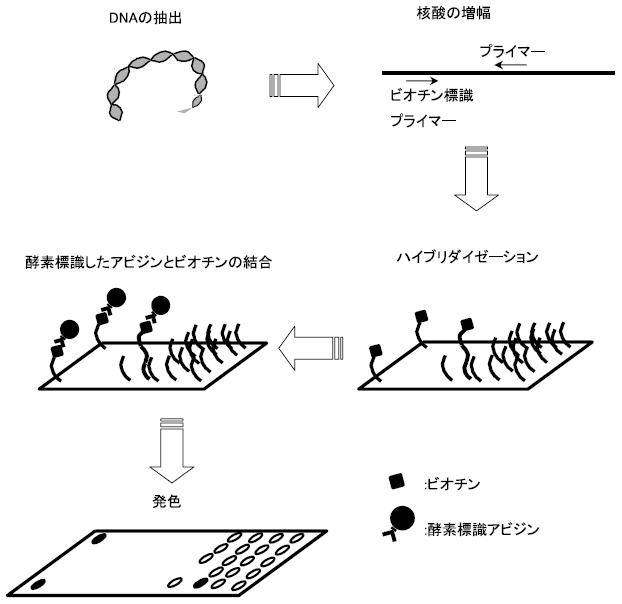
前述したようにハイブリダイゼーションによって得られた2本鎖の検出には専用の検出装置が必要であるが、我々は検出を発色によって行い、専用の検出装置を不要とした。発色方法は組織化学的手法として一般に用いられているABC法 (Avidin-Biotin Complex) と呼ばれるビオチンとアビジンの特異的な結合を利用したものである。この方法を用いた実験の流れを図2に示す。ビオチン標識したプライマーを用いて検体となる核酸を増幅させる。増幅した核酸をDNAマイクロアレイ上でハイブリダイゼーションさせ、相補的な配列を持つ固定化DNAとの2本鎖を形成させる。次にDNAマイクロアレイ上で酵素標識したアビジンとビオチンを結合させる。そこに発色基質を反応させて発色の有無を調べることにより、検体が固定化されたどのDNAスポットと2本鎖を形成したかが目視により判定できる。

このようにして開発されたDNAマイクロアレイは1検体あたり数千円程度で実験を行えるようになった。
3.DNAマイクロアレイを用いたシロザケ (Oncorhynchus keta) のmtDNAハプロタイプ判別
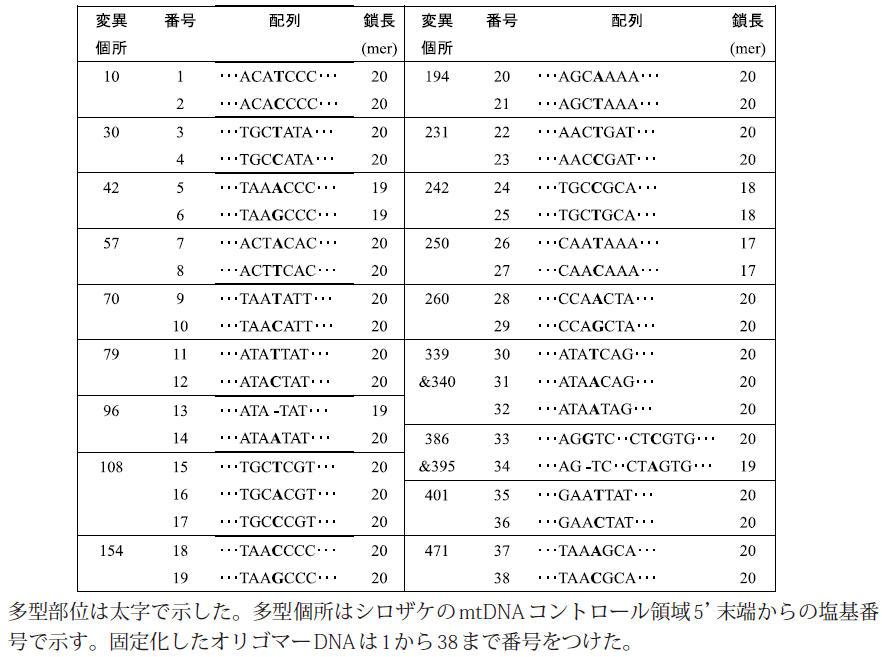
シロザケmtDNAコントロール領域5’側に見出された20の多型についてそれぞれの多型を含むようにオリゴマーDNAを設計した。それぞれの配列を表1に示す。このように設計されたオリゴマーDNA (キャプチャーDNA) 38種類をすべて前述のCarboStationR上に固定してDNAマイクロアレイを作製した。
表1 固定化したオリゴマーDNA一覧
一方、多型個所をすべて含むようプライマーを設計した。5’末端にビオチン標識をしたプライマーを用いてPCR法により対象領域を増幅し、先に作製したDNAマイクロアレイ上に滴下してハイブリダイゼーションを行い、その後、ABC法により発色させてハイブリダイゼーションシグナルを検出した。
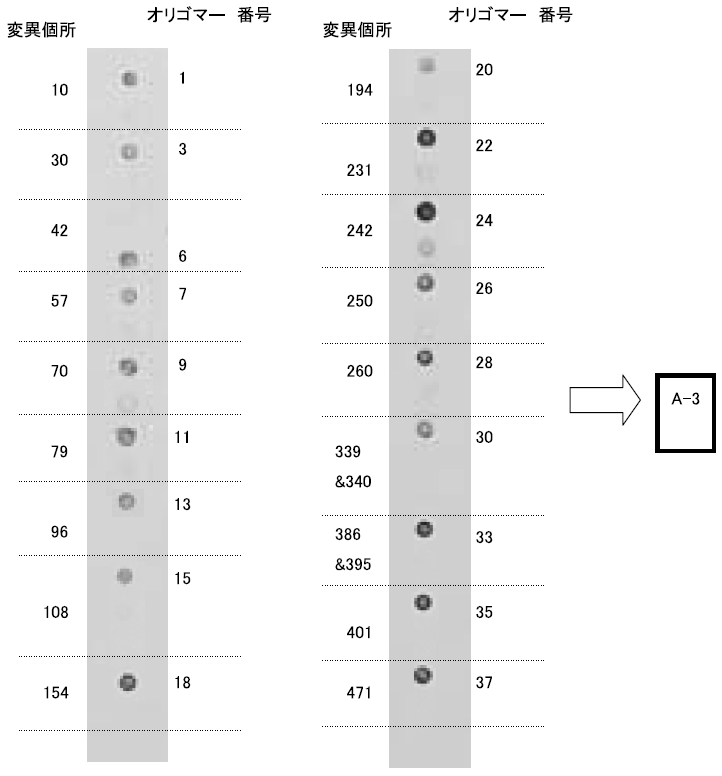
ハイブリダイゼーションシグナル検出例を図3に示す。各多型個所について2から3種類のキャプチャーDNAが固定化されているが、その中で最も濃く発色しているキャプチャーDNAの配列をPCR産物が含むと考えられる。一方、発色が薄いかまたはまったく観察されないキャプチャーDNAはPCR産物がその配列をもたないと考えられる。このように各多型個所について最も発色が強いオリゴマー番号を選び出し、組み合わせることによってハプロタイプを決定する。図3ではオリゴマー番号1, 3, 6, 7, 9, 11, 13, 15, 18, 20, 22, 24, 26, 28, 30, 33, 35, 37がそれぞれ選択され、その組み合わせによってハプロタイプ‘A-3’が決定された。

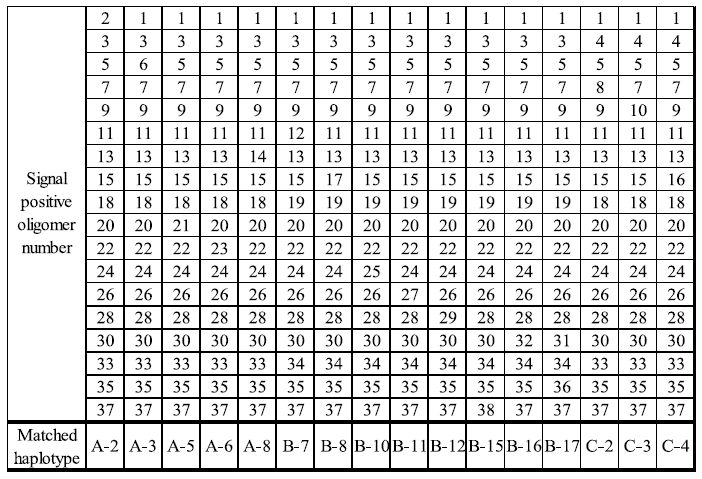
このようにして作製されたDNAマイクロアレイの性能を確認するため、予めシーケンス法によって塩基配列を決定した16サンプルを用い、DNAマイクロアレイにより20の多型個所すべてが検出できるか否か実験した。その結果を表2に示す。すべての多型が検出できており、かつシーケンス法と一致した結果を得た。さらに現在知られている30種類のハプロタイプすべてについて、DNAマイクロアレイを用いて検出を試みたところすべて検出された。このことより、迅速、低価格かつ高精度にシロザケのmtDNAハプロタイプを検出するDNAマイクロアレイを開発できた。
表2 シロザケmtDNAコントロール領域5’側20個所の多型検出結果と決定されたハプロタイプ
開発されたDNAマイクロアレイは、DNA抽出からハプロタイプの決定まで8時間以内で終了する。また、この方法による解析に必要な機器はPCR法に用いるサーマルサイクラーとハイブリダイゼーション用オーブンだけであり、特殊な装置が不要である。そのため、解析は実験室のみならず、調査船上や採卵場等の現場で実験を行うことが可能となった。
実際、2002年9月にベーリング海航海中の水産庁調査船開洋丸上で約1000個体のシロザケのmtDNAハプロタイプ判別を試みた。その結果、すべての個体のハプロタイプが判別でき、抜き取りでシーケンス解析を行った20検体についてDNAマイクロアレイ法と比較したところ一致した結果を得た。
4.終わりに
シロザケのmtDNAハプロタイプ判別を簡便かつ特殊な機器無しに検出可能な方法を確立した。本開発によって得られたDNAマイクロアレイによって大量解析が可能となり海洋調査での採用にもいたった。これらの結果を通して高精度な系群識別が可能となり、シロザケの回遊経路の解明や資源管理等の基礎的知見の蓄積がなされることを願っている。
本DNAマイクロアレイは、塩基の置換・欠失・挿入などを迅速かつ正確に検出できる有効なポストシーケンス遺伝子解析法と考えられる。現在、1枚あたり数千円しているが、数百円レベルを目標とした開発も進めている。低コスト、簡便性の実現によりDNAマイクロアレイを用いた遺伝子検査は医療、食品、環境等幅広い分野へ利用可能となる。これにより遺伝子検査は身近なものとなり、基礎研究はもとより、安全で健康的な生活の実現に貢献できるものと考えている。
文献
1) Ferguson, A., Taggart, J. B., Prodohl, P. A., McMeel, O., Thompson, C., Stone, C., McGinnity, P., Hynes, R. A.: J. Fish. Biol., 47 (SupplA), 103 (1995).
2) Wilson, G. M., Thomas, W. K., Bechenbach, A. T.: Can. J. Fish. Aquat. Sci., 44, 1301 (1987).
3) Cronin, M. A., Spearman, W. J., Wilmot, R. L., Patton, J. C., Biciham, J. W.: Can. J. Fish. Aquat. Sci., 50, 708 (1993).
4) Bickham, J. W., Wood, C. C., Patton, J. C.: J. Hered., 86, 140 (1995).
5) Sato, S., Ando, J., Ando, H., Urawa, S., Urano, A., Abe, S.: Zool. Sci., 18, 99 (2001).
6) Sato, S., Kojima, H., Ando, J., Ando, H., Wilmot, R. L., Seeb, L. W., Efremov, V., LeClair, L., Buchholz, W., Jin, D.-H., Urawa, S., Kaeriyama, M., Urano, A., Abe, S.: Environ. Biol. Fish., in press.
7) Abe, S., Sato, S., Kojima, H., Ando, J., Ando, H., Wilmot, R. L., Seeb, L. W., Efremov, V., LeClair, L., Buchholtz, W., Jin, D.-H., Urawa, S., Kaeriyama, M., Urano, A.: Fish. Sci., 68 (Suppl), 353 (2002).
8) Shene, M., Shalon, D., Davis, R. W., Brown, P. O.: Science, 270, 467 (1995).
9) Hacia, J. G.: Nature Genet., 21, 42 (1999).
10) Lander, E. S.: Nature Genet., 21, 3 (1999).
11) Moriya, S., Urawa, S., Suzuki, O., Urano, A., Abe, S.: Marine Biotechnology, in press.