【トピックス】
フォールディングのためのナノマシン:大腸菌のシャペロニンGroEL
田口英樹
東大院・新領域、JST/さきがけ21
1.はじめに
シャペロニンの話題を工学系、化学系の分野でも聞くことが増えてきた。人工的にシャペロニンを作ってみる1)、とか、半導体ナノ粒子をシャペロニンの空洞内に閉じ込める2)、などを聞いたとき生化学者として非常にびっくりしたが、シャペロニンの機能、構造をずっと調べてきた者として、シャペロニンが各分野に広がっていくのを見るのはとてもうれしいものである。
本稿では、異分野の方にシャペロニンを紹介するつもりで、まずは現在までにわかっている大腸菌のシャペロニンGroELの構造と作用機構を簡単に説明する (詳しくは文献3-6)を参照)。さらには、GroELの作用機構研究の副産物として得られてきた様々なGroEL変異体について、その代表的なものの紹介も行う。本稿が化学・工学などの方々に何かインスピレーションを与えることができれば幸いである。
2.シャペロニンの構造
大腸菌など真正細菌の場合、シャペロニンは2種類のサブユニット、GroELとGroESからなる4,6)。多くの分子シャペロン (DnaKやClpBなど) が生存に必須でなく、ゲノム中に遺伝子が存在しないこともあるのに対し、GroELとGroESはほぼすべてのバクテリアに存在し、また調べられた限り生存に必須の蛋白質である。必須である理由は、生存に必須な別の蛋白質のフォールディングがシャペロニンに完全に依存すればシャペロニンも必須となる、というのが通説だが、実際のところはよくわかっていない。蛋白質のゆりかごとなるシャペロニンの主役はポリペプチドを結合して、ATPの加水分解を行うGroELである。一方、GroESはGroELと結合して「フタ」となり、GroELの働きを制御する。まず構造を説明する。
2.1 GroELはナノサイズのかご状蛋白質である
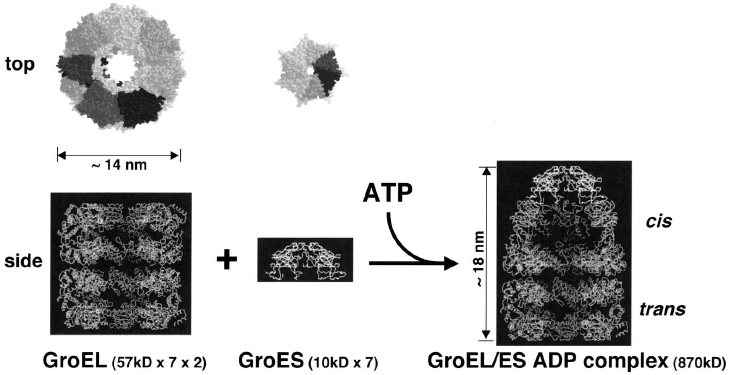
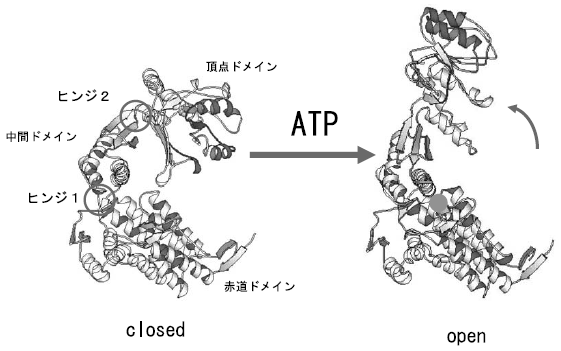
GroELは57 kDのサブユニット7つからなるリングが背中合わせに2つ重なった14量体構造 (総分子量は~800 kD) をとっている7) (図1)。このようにリング構造をとることで内部には直径4~5 nm程度の空洞ができる。GroELのサブユニットは、赤道ドメイン、頂点ドメイン、その両者をつなぐ中間ドメインの3つのドメインからなる。赤道ドメインはATP結合領域を含んだGroEL 構造の土台である (図2)。一方、頂点ドメインには基質となるポリペプチドの結合領域 (と同時にGroES結合領域) がある。中間ドメインは2つのヒンジ部位を含んでいる。
図1 GroEL、GroESおよびGroEL-GroES-ADP複合体の立体構造
図2 GroELサブユニットの立体構造
2.2 GroELとGroESが結合するとフォールディングのための快適空間が形成する
GroELにATPやADPが結合するとGroESが安定に結合できるようになる。ADPを含んだGroEL-GroES複合体は片方のGroELリングの形が上にすぼまり、その上にGroESが乗っかったような「弾丸型」構造となる8)。この立体構造からまずわかることは、GroES、ADPの結合した側のGroELリング (cisリング) の頂点ドメインが大きく立ち上がって開いていることである (この構造をオープン型と呼ぶ)。そのオープン型では、GroELリング内の空洞が体積にして約2倍に拡がる。ここでGroES (10 kD のサブユニットが7つ) はGroELの空洞に「フタ」をすることになり、内部に隔離された空洞が出現する (図1)。これが、シャペロンの「ゆりかご」の実体である。この空洞の中には、57 kD くらいまでの大きさのポリペプチドが入り込んで、フォールディングのわなである凝集体形成の危険から解放され、天然構造までフォールディングできる9)。
2.3 GroELの変性蛋白質結合部位はGroESの結合部位とオーバーラップしている
GroELがフォールディングを助けるためにはまず変性蛋白質を認識して結合しなければならない。GroELの変性蛋白質結合部位は頂点ドメインに全てが集中しており、リングの内側の縁で変性蛋白質を結合する10)。ここで重要なのは、変性蛋白質結合部位はGroESの結合部位とほぼすべてがオーバーラップしていることである10,11)。すなわち、ヌクレオチド存在下でGroELが構造変化を起こしてGroES と結合すると同時に、変性蛋白質は行き場がなくなり、空洞内に閉じ込められる、というのが構造から想定された蛋白質閉じ込め機構である。
また、cisリングでのGroELのオープン構造への変化によって、空洞内は変性蛋白質にとってフォールディングに都合の良い環境になる。GroES が結合していないときには、GroEL のリング内の表面には疎水性アミノ酸残基がたくさん露出しているが、GroESが結合するとその疎水性残基はGroEL-GroESコンタクトなどに隠されてしまう8)。結果としてcisリング内では親水的な残基 (負電荷が中心12)) が主になる。
2.4 変性蛋白質を空洞内に閉じ込めたGroEL-GroES複合体の構造は歪んでいた
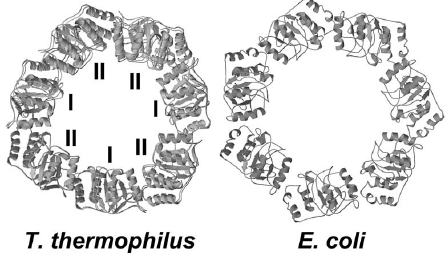
上で述べたGroEL-GroES複合体は空洞内に基質蛋白質を含まない「空」の構造である。では、空洞内に基質蛋白質が含まれているときにシャペロニンの立体構造はどうなるのであろうか?好熱菌Thermus thermophilus のGroEL-GroES複合体は非常に安定なので、細胞内での基質蛋白質を閉じ込めたままの複合体を精製できる13)。すなわち、細胞内で空洞に基質蛋白質を詰め込んだままのシャペロニン複合体が精製できるのである (ちなみに大腸菌のGroEL、GroESはすぐに解離する。結晶構造がわかっているGroELGroES複合体はADP存在下で再構成させたものである8)。この好熱菌シャペロニンの立体構造を決めたところ、興味深いことにGroESが結合しているGroELの頂上ドメインだけが大きく歪んでいて、特徴的な七回対称から大きく崩れていることがわかった12)(図3)。基質となる変性蛋白質がシャペロニンの構造・機能にどのように影響を及ぼすかは今後の大きな課題である。
![]()
以上のようにGroEL/GroESの立体構造はシャペロニンの機能を理解する重要なヒントを与えてくれた。では、上で述べた立体構造が実際の反応サイクルでどのような意味をもつのか次に見てみよう。
3.反応サイクル
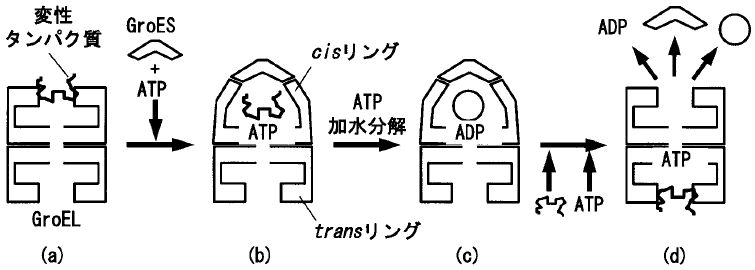
次に、現在信じられているGroELとGroESの反応サイクルの概要を説明しておこう4)(図4)。
図4 シャペロニン (GroEL、GroES) の反応サイクルの概要
(a) GroELに基質となる変性蛋白質が結合する。(b) GroELにATPが結合すると、ATPが結合したリングにGroESが結合する。変性蛋白質が結合しているリングにATPが結合した場合、変性蛋白質はGroESのふたの下にできたGroELの空洞の中に閉じこめられる (cis複合体)。この過程はATPの結合で十分でATPの加水分解は必須ではない。基質蛋白質によっては、この段階でフォールディングが完成する場合もある (後述するが、このステップは2つに分割されると思われる)。(c) cis複合体中のATPが8秒程度で加水分解されてADP型のcis複合体となると、GroESの解離の準備が整う。(d) ADP型のcis複合体のcisと反対側のtransリングにATPが結合すると、GroESが解離する。同時に中に閉じ込められていた基質蛋白質およびADPも放出される。基質蛋白質はフォールディングが完了していなくても、放出される。
ここでATPが結合したtransリングにGroESが結合すると、ステップ (b) に戻る。この一連のサイクルが8秒間隔で進行して、くり返されていく。
我々は、この反応サイクルをさらに詳しく解析するために、1分子蛍光イメージングの手法をシャペロニン研究に応用した。これまでに、 (1) 1分子のレベルでGroELとGroESの相互作用のダイナミクスを解析14)、(2) GroEL/ES 複合体の内部で起こるGFPフォールディングを1分子蛍光イメージングする実験系を構築した15)。
3.1 GroELとGroESの結合と解離の1分子観察
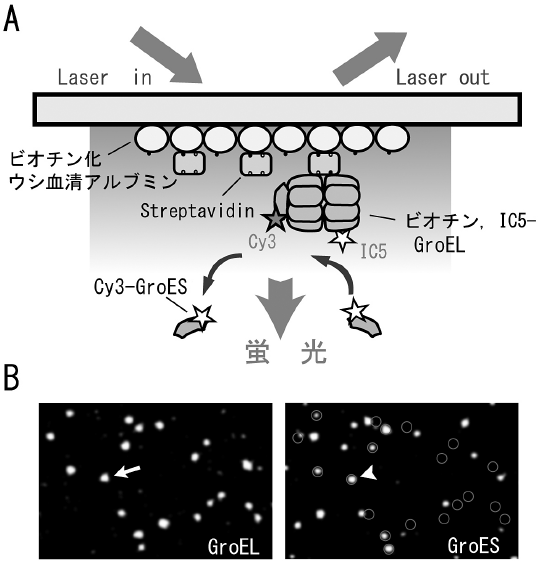
我々は図5Aに示すような方法で蛍光標識したGroELとGroESの結合・解離を観察した14)。1分子の蛍光色素のイメージングには全反射蛍光顕微鏡によるエバネッセント照明を用いた。この顕微鏡は照射面から約150 nmの領域しか励起しないため、背景光が劇的に減るだけでなく、スライドガラス表面に固定した蛍光標識蛋白質を局所励起できるという利点をもつ。まず、蛍光色素 (IC5) で標識したGroELを固定して位置を決定しておく (図5B左)。続いて、別の蛍光色素 (Cy3) で標識したGroESをATP存在下で加え、顕微鏡観察する。GroELに結合していないGroESはブラウン運動が激しいため蛍光スポットとならないが、GroESがGroELに結合すると輝点として観察できる (図5B右)。このような実験系で、単一のGroEL分子にGroESが結合し、そして解離する様子が初めて明らかとなった。

3.2 1 分子蛍光イメージング技術を用いたGFPフォールディングの解析
さらに我々は、シャペロニン1分子内で起こる蛋白質のフォールディングを可視化した15)。基質となる蛋白質としては、緑色蛍光蛋白質 (GFP) を用いた。[変性GFP-GroEL]の複合体を作製してスライドガラスに固定し、その後GroES、caged ATPで観察セルを満たした。顕微鏡上で、この観察セルに紫外線を照射してcaged ATPからATPを生成すれば、GFP-GroEL複合体にGroESが結合してGFPのフォールディングが起こり、その蛍光が観察できる。実験の結果、紫外線照射前には存在しなかったGFPの蛍光が、紫外線照射後にGroELの位置に次々と現れた。このような実験系で、蛋白質フォールディングを1分子レベルで捉えることに初めて成功した。
3.3 1分子研究から産まれた2タイマーモデル
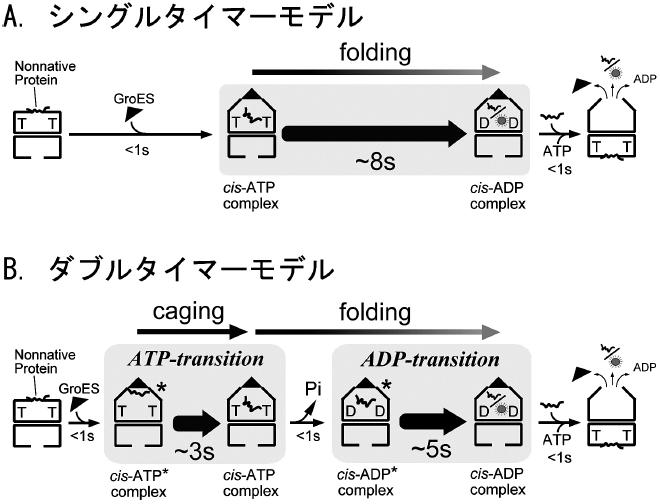
このようにGroELの働きを1分子レベルでイメージングできたわけであるが、1分子実験の本領発揮は統計的な解析にある。多分子の平均からはなかなか得られない情報が1分子研究から抽出できることが多いのである。実際、シャペロニンの1分子研究からも多分子では引き出せない情報が得られた。一番重要なのは、GroEL-GroES複合体の寿命解析の統計処理の結果である。従来の生化学研究からは、この複合体の寿命はGroELのATP加水分解反応 (約8秒) で規定されていることになっている (図6A)。その場合、律速反応が一つ、つまりGroESは結合後直ちに解離し始めるので結合時間のヒストグラムは単一の指数関数でフィッティングできる右下がりの形になり、その時定数が8秒くらいになると予想した。しかし、意外なことにヒストグラムは5秒付近にピークをもつ山型の分布となった14)。これは、GroESはGroELに結合後、ある中間体を経てから解離することを意味する。この山型のヒストグラムを2段階の反応でフィットしたところ、3秒と5秒の2つの時定数からなることがわかった。従来、GroELはATPの加水分解に必要な8秒をタイマー代わりに用いる、と考えられてきたが、実は、そのタイマーは3秒と5秒に分けられることが1分子実験から明らかとなった。
さらに1分子研究に付随して慎重に生化学実験をやり直した結果もタイマーが二つという上記の結論を支持したことから我々は2タイマーモデルを提案している15)(図6B) (詳細は省くが、従来の実験ではGroELのATP加水分解などの速度論を調べるのに基質蛋白質を入れていなかったが、変性蛋白質があると大きく速度論が異なることがわかった)。このモデルのポイントは、従来説では省略されていた変性蛋白質の取り込みを重要なステップとして位置づけたことである。従来の1つのタイマー説では、シャペロニンの空洞内でのフォールディングは説明できても、どのように変性蛋白質を取り込むか、という、ある意味ではシャペロニンの機能の一番重要な点が説明されていない。しかし、2タイマーモデルでは、変性蛋白質の取り込みには従来想定されていなかった中間複合体が存在することを示した。従来モデルではGroESと変性蛋白質は同じ結合部位を単に競合する。そのために、GroESは変性蛋白質を「はじき飛ばす」というイメージであったのに対し、2タイマー説では、GroESが結合したときにはまだ変性蛋白質はGroELのどこかに結合したまま、と考える (後述するように、生化学実験からもそのような中間体が示唆されてきている)。
では、タイマーが2つに分かれることの意義は何であろうか。「タイマーが2つ」=「2段階反応」ということは、凝集しやすい変性蛋白質がシャペロニン内部に取り込まれてから最初のタイマーの間は外に放出されないことを意味する。シャペロニンの最大の任務は変性蛋白質の凝集防止にあるが、2つのタイマー機構で一定時間はフォールディングのための空間を保持できるのは都合がよいと考察している。
4.GroEL変異体ギャラリー
工学系、化学系の方々とGroELについてディスカッションしているとGroELを改変してより有用な (おもしろい) ものにしてみたい、という話になることがままある。その際に、「こんなのもあります、あんなのもあります」と、さみだれ式に説明しているのが常である。よい機会なので本稿では代表的なGroEL 変異体をその性質とともに紹介したい。作用機構解明の途上で産まれたものばかりだが、なかなかどうしてGroELというのは「いじりがい」がある蛋白質ということがわかってもらえるだろう。
4.1 シングルリングGroEL変異体 (SR1)
GroELのリング間の界面で相互作用しているアミノ酸3カ所に変異を入れるとダブルリング構造を取れない16)。が、このシングルリングGroEL (SR1と呼ばれる) は、GroESによる蛋白質の空洞内への取り込みをしてフォールディングを助ける。ただ、反対側のGroELリングがないために、一回GroESを結合するとGroESがなかなか外れなくなってしまう (このことはGroELがダブルリングである説明にも使われる)。シングルターンオーバー変異体、GroESのトラップとして使われることがある16)。
4.2 GroEL/ES複合体の寿命が非常に長くなるGroEL変異体 (D398A)
ATPのγリン酸を引き抜く際に重要なカルボキシル基を提供する重要なAsp398に変異を入れるとATP加水分解活性が野生型の2%以下になる8,17)。このD398AはATP結合に伴う構造変化は野生型と同様に起こすので、GroESの結合や変性蛋白質の空洞への取り込みは通常通りである17)。ただ、ATPが加水分解しないのでサイクルが次に回らず、GroESが30分以上はずれない。空洞を長時間保持したいときに使われる。
4.3 変性蛋白質が結合しなくなるGroEL変異体
GroELの変性蛋白質の結合領域は疎水残基が主である。GroELのH、Iヘリックスと200番付近のループ付近の疎水残基に変異を入れると、変性蛋白質との結合が非常に弱くなる (例えば、Y203E, L237Qなど)10)。
4.4 GroESが結合しないが変性蛋白質はより強く結合するGroEL変異体 (N265A)
4.3の変性蛋白質結合部位の中のAsn265はGroESとの結合に重要でこの残基をAlaに置換したGroELはGroESと結合しなくなるが10)、変性蛋白質との結合は野生型GroELよりも強くなる11)。
4.5 構造変化の開閉をS-S結合で制御可能なGroEL変異体
GroELの頂点ドメインと赤道ドメインが最も近づくAsp83とLys327を共にCysに変えたGroELは空気酸化によりジスルフィド架橋されて、ATPを加えてもオープン型への構造変化を起こさない18)。その後、ジスルフィド結合を還元すると元に戻る。なお、野生型GroELには3つCysがあるので、あらかじめすべてのCysをSerに置換している (その後、野生型のCysはAlaに置換する方がよりよいことがわかった19))。
4.6 基板固定用GroEL (D490C)
1分子レベルでの研究のためにはGroELを活性を保ったままガラス板に固定することが必須である。野生型GroELの場合、ガラス板に非特異的な吸着で固定すると、活性を失ってしまう。そこでGroELの赤道ドメインの表面にCysを導入したところ、そのCysにビオチンや蛍光色素を導入してもGroELの機能に影響なく、アクティブなままGroELを基板に固定することができた14)。
4.7 低温において反応サイクル途中でストップするGroEL変異体 (C138W)
中間ドメインのヒンジのそばに位置するCys138にTrpを導入すると37℃では野生型と機能が変わらないのに25℃だとATPaseサイクルがストップすることがわかった20)。この止まった状態ではGroESが結合しているにもかかわらず変性蛋白質は空洞内に閉じ込められていない。すなわち、図6Bの2タイマーモデルの最初の中間体と目されている。
4.8 一筆書きGroEL21)
GroELリング内で各GroELサブユニットのC末端は隣のN末端と近い位置に来る7)。そこで7つのGroELサブユニットを遺伝子操作で数珠繋ぎにつなげて7つのサブユニットを一本にしてしまう、という離れ業が成し遂げられている21)。この一筆書きGroELを用いるとリング内の特定のサブユニットだけに変異を導入するというようなことが可能である21)。
4.9 リング間ハイブリッドGroEL (二つのリングが別の種類)
ATPとカリウム存在下で大腸菌GroELと好熱菌GroELを共存させると大腸菌と好熱菌それぞれのリングからなるハイブリッドGroELが得られる22)。ハイブリッド形成の機構はよくわかっていないが、ATPとK+存在下でリング間のコンタクトがなくなる、もしくは弱くなるからだと推測されている。同じ大腸菌同士でもできる割合は少ないもののやはりハイブリッドGroELができる23) (mix ring とも呼ばれる)。
4.10 リング内ハイブリッドGroEL (リング内に野生型と変異型を混ぜる)24)
野生型GroELと変異体GroELを尿素で変性させたのちに、リング構造に再構成させるとリング内に変異体が混じったGroELが得られる。野生型とオープン型になり得ない変異型GroEL (上記の4.5) を混ぜた実験からリング内に一個でもオープン型にならないGroELがあると、フォールディングを助ける能力が大きく落ちることがわかった。
4.11 GroESが両方に結合するフットボール型25)
変異体ではないが、リン酸アナログとして知られるフッ化ベリリウムはGroELの反応サイクルを途中でストップさせ、さらには両側のリングにGroESが結合することがわかった。このときGroELのダブルリングの両方に空洞ができ、一つのGroELあたり2分子の基質蛋白質を取り込んでフォールディングを助ける。
4.12 単量体GroEL (ミニシャペロン)26-28)
GroELのN末端を除去すると14量体構造にならない。このような単量体GroELでも一部シャペロンとしての働きをもつことが知られている。ただ、変性蛋白質との親和性は非常に弱く、ATPに依存しない。
なお、GroEL、GroESともにTrpは含まれていない。大腸菌GroELは60℃くらいまで安定である (GroESは90℃でも失活しない)。
5.おわりに
以上、後半はかなり好き勝手に書かせていただいた。実際、筆者は10数年来GroELにのめり込んでいるが、まだまだ楽しい蛋白質である。今後はより詳細に作用機構を調べてGroELの働きをアニメーションで再現できるようになれば、と夢想している。また、さまざまな分野の研究者がシャペロニンGroELを使うようになって、シャペロニンが他の分野でも「必須」の蛋白質になれば、と思う。
文献
1) Nomura, Y., Ikeda, M., Yamaguchi, N., Aoyama, Y., Akiyoshi, K.: FEBS Lett, 553, 271 (2003).
2) Ishii, D., Kinbara, K., Ishida, Y., Ishii, N., Okochi, M., Yohda, M., Aida, T.: Nature, 423, 628 (2003).
3) 田口英樹:蛋白質核酸酵素, 47, 1196 (2002).
4) Bukau, B., Horwich, A. L.: Cell, 92, 351 (1998).
5) Sigler, P. B., Xu, Z., Rye, H. S., Burston, S. G., Fenton, W. A., Horwich, A. L.: Annu. Rev. Biochem., 67, 581 (1998).
6) Hartl, F. U., Hayer-Hartl, M.: Science, 295, 1852 (2002).
7) Braig, K., Otwinowski, Z., Hegde, R., Boisvert, D. C., Joachimiak, A., Horwich, A. L., Sigler, P. B.: Nature, 371, 578 (1994).
8) Xu, Z., Horwich, A. L., Sigler, P. B.: Nature, 388, 741 (1997).
9) Sakikawa, C., Taguchi, H., Makino, Y., Yoshida, M.: J. Biol. Chem., 274, 21251 (1999).
10) Fenton, W. A., Kashi, Y., Furtak, K., Horwich, A. L.: Nature, 371, 614 (1994).
11) Motojima, F., Makio, T., Aoki, K., Makino, Y., Kuwajima, K., Yoshida, M.: Biochem. Biophys. Res. Commun., 267, 842 (2000).
12) Shimamura, T., Koike-Takeshita, A., Yokoyama, K., Masui, R., Murai, N., Yoshida, M., Taguchi, H., Iwata, S.: Structure (Camb), 12, 1471 (2004).
13) Taguchi, H., Konishi, J., Ishii, N., Yoshida, M.: J. Biol. Chem., 266, 22411 (1991).
14) Taguchi, H., Ueno, T., Tadakuma, H., Yoshida, M., Funatsu, T.: Nat. Biotechnol., 19, 861 (2001).
15) Ueno, T., Taguchi, H., Tadakuma, H., Yoshida, M., Funatsu, T.: Mol. Cell, 14, 423 (2004).
16) Weissman, J. S., Hohl, C. M., Kovalenko, O., Kashi, Y., Chen, S., Braig, K., Saibil, H. R., Fenton, W. A., Horwich, A. L.: Cell, 83, 577 (1995).
17) Rye, H. S., Burston, S. G., Fenton, W. A., Beechem, J. M., Xu, Z., Sigler, P. B., Horwich, A. L.: Nature, 388, 792 (1997).
18) Murai, N., Makino, Y., Yoshida, M.: J. Biol. Chem., 271, 28229 (1996).
19) Rye, H. S., Roseman, A. M., Chen, S., Furtak, K., Fenton, W. A. Saibil, H. R., Horwich, A. L.: Cell, 97, 325 (1999).
20) Kawata, Y., Kawagoe, M., Hongo, K., Miyazaki, T., Higurashi, T., Mizobata, T., Nagai, J.: Biochemistry, 38, 15731 (1999).
21) Farr, G. W., Furtak, K., Rowland, M. B., Ranson, N. A., Saibil, H. R., Kirchhausen, T., Horwich, A. L.: Cell, 100, 561(2000).
22) Taguchi, H., Amada, K., Murai, N., Yamakoshi, M., Yoshida, M.: J. Biol. Chem., 272, 18155 (1997).
23) Burston, S. G., Weissman, J. S., Farr, G. W., Fenton, W. A., Horwich, A. L.: Nature, 383, 96 (1996).
24) Shiseki, K., Murai, N., Motojima, F., Hisabori, T., Yoshida, M., Taguchi, H.: J. Biol. Chem., 276, 11335 (2001).
25) Taguchi, H., Tsukuda, K., Motojima, F., Koike-Takeshita, A., Yoshida, M.: J. Biol. Chem., in press (2004).
26) Taguchi, H., Makino, Y., Yoshida, M.: J. Biol. Chem., 269, 8529 (1994).
27) Makino, Y., Taguchi, H., Yoshida, M.: FEBS Lett., 336, 363 (1993).
28) Zahn, R., Buckle, A. M., Perrett, S., Johnson, C. M., Corrales, F. J., Golbik, R., Fersht, A. R.: Proc. Natl. Acad. Sci. USA, 93, 15024 (1996).