【トピックス】
超好熱性古細菌の分子シャペロン
養王田正文、飯塚 怜、座古 保、吉田尊雄
東農工大院・共生科学技術研究部
1.はじめに
超好熱性菌は、至適生育温度が80℃以上の微生物であり、その多くは古細菌に分類される。超好熱性菌が水の沸点に近い高温で生育が可能なのは、その生体分子、特にタンパク質が物理的に高い安定性を有しているからであり、タンパク質の耐熱性機構の解明や耐熱性酵素の工業的利用などで注目されている。そのため、1996年にメタン生成古細菌Methanococcus jannaschiiのゲノムが解読1)されて以来、多くの超好熱性菌のゲノムが解読されている。これらのゲノムにコードされているタンパク質の多くは高い耐熱性を示し、大腸菌で発現させた場合、加熱処理だけで容易に精製が可能である。この優れた特性から、超好熱性菌は、ゲノムにコードされているタンパク質の網羅的構造・機能解析のターゲットとして活用されている。
もちろん、タンパク質には個性があり、超好熱性菌の全てのタンパク質が一様に安定ということはなく、比較的耐熱性の低いタンパク質も存在する。また、超好熱性菌にも生育至適温度があり、熱ショック応答もある。Jonathan Trent教授らは、好酸好熱性古細菌Sulfolobus shibatae*1の熱ショック誘導タンパク質を解析し、分子量55kDaの熱ショックタンパク質TF55を発見した2)。このTF55はタンパク質の立体構造形成に関与する分子シャペロンのなかでも重要な大腸菌のシャペロニンGroELとアミノ酸配列や構造上類似していると同時に、真核生物のTCP-1と呼ばれるタンパク質と高い相同性があることから脚光を浴びた。それまで、真核生物の細胞質にGroELに相当するシャペロニンが存在しないことが大きな謎であったが、この結果からGroELとは異なるタイプのシャペロニンが真核生物や古細菌に存在することが明らかになった。これは、古細菌に真正細菌が共生して進化することにより真核生物が生まれたと考える共生説と対応している。現在、真正細菌やオルガネラに存在するGroELと類似のシャペロニンを1型シャペロニン、古細菌のシャペロニンや真核生物のシャペロニンCCT (Cytosolic Chaperonin containing TCP-1) を2型シャペロニンとして分類している3)。
*1 厳密な意味では超好熱性ではないが、ここでは超好熱性古細菌として取り扱う。
S. shibataeの熱ショック応答タンパクで誘導されるのは主にシャペロニンだけであり、他の生物と比較して極端に熱応答タンパク質の種類が少ない。また、Sulfolobusや他の超好熱性古細菌のゲノム解析の結果からも分子シャペロンなどのタンパク質構造形成に関与するタンパク質としてはシャペロニン以外では、sHsp (small heat shock protein)、PPIase (peptidyl-prolyl cistrans isomerase) とプレフォルディン (後述) の遺伝子が存在するのみである。常温性の古細菌がHsp70システムなどを有することと比較すると、超好熱性古細菌は非常にシンプルな分子シャペロンシステムを有している。古細菌特有の新しい分子シャペロンの存在も示唆されている4)が、超好熱性古細菌のタンパク質の多くは分子シャペロンに依存しないで構造形成が可能であり、一部の比較的安定性の低いタンパク質のために少数の分子シャペロンが機能していると考えるのが妥当だろう。
本稿では、超好熱性古細菌の分子シャペロンシステムにおいて主要な役割を担っている2型シャペロニンを中心に、超好熱性古細菌の分子シャペロンシステムと熱ショック応答について紹介する。
2.超好熱性古細菌Thermococcus sp. KS-1の由来シャペロニンと熱ショック応答
シャペロニンは2層の環状構造をしており、その中の空洞に変性タンパク質を捉え、ATP依存的にそのフォールディングを促進する。大腸菌のシャペロニンGroELを用いた研究により、1型シャペロニンの詳細な反応機構の解明が進んでいる。GroES (Hsp10) の7量体がこの空洞のふたの役割をしており、GroES (Hsp10) の結合と解離に伴うふたの開閉がシャペロニンの機能に重要な役割を担っていることが分かっている。それに対して、2型シャペロニンにはGroESが存在しないことが反応機構の大きな謎であった。
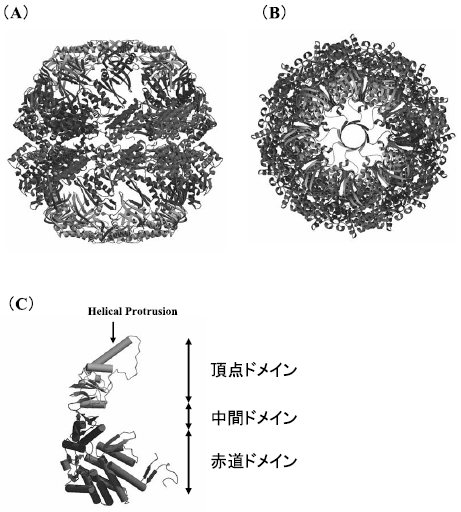
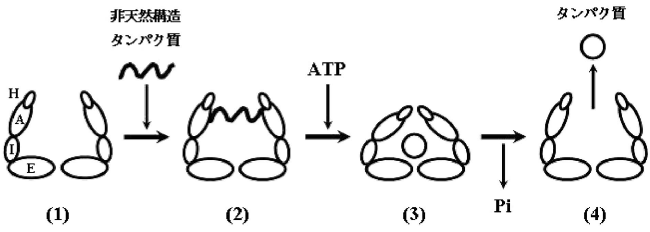
2型シャペロニンの研究における最も大きな進歩は、好酸好熱性古細菌Thermoplasma acidophilum 由来シャペロニンのX線結晶構造解析の結果により得られた5)。GroELとの配列の比較から、2型シャペロニンの頂点ドメインにGroELには存在しない配列が挿入されているが、X線結晶構造解析から、その挿入された領域が空洞の“ふた”を形成していることが明らかになった (図1)。この領域はその構造上の特徴からHelical Protrusionと命名されている。この結果から、2型シャペロニンには“ビルトイン構造の“ふた” (Built-in Lid) ”があり、ふたが開いて変性タンパク質を空洞に捕獲し、閉じてフォールディングを促進し、最後にまたふたが開いて構造形成したタンパク質をリリースするというものである。

残された問題は、この開閉機構とタンパク質フォールディング機構の関連の証明である。ところが、困ったことに、in vitroでタンパク質フォールディング活性を示す古細菌のシャペロニンはほとんどないという問題があった。J. Trent教授や私たちはSulfolobusのシャペロニンを用いてフォールディング活性の研究を行っていたが、それらはヌクレオチド依存的なフォールディング活性は示さなかった6,7)。最も研究が進んでいたT. acidophilumのシャペロニンについてもATP依存的フォールディング活性は確認されていない。Sulfolobusシャペロニンの研究を執拗に続けているJ. Trent 教授らは、古細菌シャペロニンがタンパク質フォールディングではなく細胞骨格や細胞膜の要素として機能すると主張するに至っている8,9)。幸い、私たちは、超好熱性古細菌Thermococcus sp. strain KS-1 (T. KS-1) 由来シャペロニンがin vitroで高いタンパク質フォールディング活性を示すことを発見し10)、このシャペロニンを用いて反応機構の解明の手がかりを得ることに成功した。これまで、様々な古細菌のシャペロニンが研究されているが、これほど優れた特性を示すシャペロニンは見つかっておらず、古細菌シャペロニン研究の理想的なモデルシステムであると言ってよい。T. KS-1は共同研究者の丸山正博士が鹿児島県子宝島で採取した超好熱性古細菌であり、京都大学の今中忠行教授たちが同じ場所で採取したThermococcus kodakaraensisと近縁の種類だと考えられる。実際に、T. kodakaraensisのシャペロニンはT. KS-1のシャペロニンとほぼ同じ配列である11)。
多くの古細菌ではシャペロニンの2種類のサブユニット (αとβ) があり、それらの相同性は60%程度である。T. acidophilumでは、これらが1対1のヘテロオリゴマーを形成する。PyrococcusやM. jannaschiiには遺伝子が1つだけ存在するのでホモオリゴマーとして機能する。Sulfolobusには2種類のサブユニットが存在すると考えられていたが、ゲノム解析により3番目のサブユニットが存在することが明らかになった。最近、この3番目のサブユニットγは低温で誘導されることが分かった12)。
T. KS-1には、2つのシャペロニン遺伝子が存在する10)。これらは著しく高い相同性 (81%) があり、これらのサブユニットは相互にランダムな複合体を形成する可能性を示唆していた。またβサブユニットが1種類のサブユニットを有するPyrococcusと高い相同性を有することから、ホモオリゴマーとして機能することも示唆されていた。実際に、それぞれのサブユニットは単独で環状構造を形成し、ATP依存的なタンパク質フォールディング活性を有していた10)。
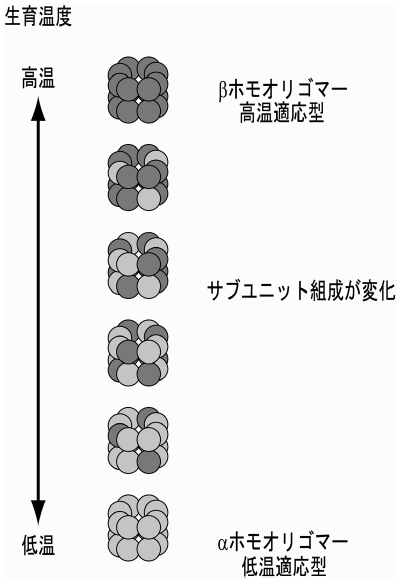
種々の温度で培養したT. KS-1におけるα、βサブユニットの量をサブユニットに特異的抗体を用いて解析したところ、β/α比は80℃では0.45であったが、90℃では8.5になっていた。αの量はほぼ一定なのに対して、βの量は温度上昇に伴い著しく上昇していた13)。また、βを多く含むシャペロニンは熱安定性に優れていることから、αは低温適合型、βは高温適合型のサブユニットであり、T. KS-1は温度に対応してシャペロニンの組成を変化させ、高いフォールディング活性を維持していると考えられる (図2)14)。

T. KS-1では高温においてsHspの発現も顕著に誘導されることから15)、T. KS-1はシャペロニンβサブユニット及びsHsp を発現することにより熱ストレスに対応していると考えて良い。シャペロニンが常に発現しているのに対して、sHspは高温のみで生産されていることから、シャペロニンは熱ショック応答以外の通常のタンパク質構造形成にも重要なのに対し、sHspは熱ショック時においてのみ機能すると考えられる。しかし、シャペロニンやsHspの基質となるタンパク質は明らかになっていないので、in vivoでの機能の詳細は明らかではない。
3.超好熱性古細菌由来シャペロニンのタンパク質フォールディング機構
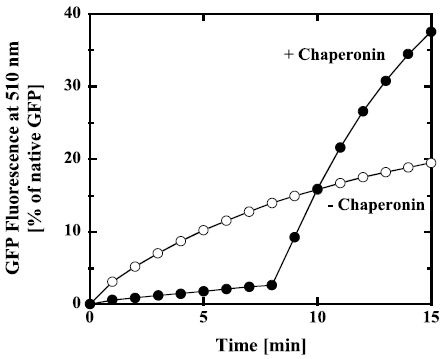
タンパク質フォールディング活性は、主にGFPを基質として用いて解析している。酸で変性したGFPを希釈により中和すると自発的に巻き戻り、蛍光を発するようになる。シャペロニンが存在すると、この自発的フォールディング阻害され、蛍光を発しなくなる。この状態をArrest状態と呼ぶ。ここでATPを加えるとATP依存的にフォールディングが推進されることによりフォールディング活性の有無を調べている。とはいえ、相手は超好熱性シャペロニンなので、それほど簡単ではない。当然だが、シャペロニンが元気な高温 (50℃以上) ではGFPのフォールディングは不可能であり、GFPが好む常温では、シャペロニンは全く機能しない。そこで、耐熱性GFP変異体16)を用いて60℃前後で実験を行うことにより、GFPを用いたフォールディング活性の測定が可能になった (図3)17)。

特に興味深いことは、非加水分解性ATPアナログであるAMP-PNPにより、βのホモオリゴマーは空洞に閉じ込めたままの状態でGFPをフォールディングすることである。この結果から、ATPの結合によりBuilt-in Lidが閉じ、内部でタンパク質のフォールディングが起きることが示唆された17)。
一方、他のグループはフォールディング機構の代わりにBuilt-in Lidの開閉に焦点を当てて研究を行っていた。Gutsche博士らは、T. acidophilum由来シャペロニンを中性子小角散乱法により解析し、ATPまたはAMP-PNP存在下において分子長の短い成分が増大することから、ATP結合により“ふた”が閉じた構造へと転換すると報告した18)。しかし、Meyer博士らは、リン酸アナログを用いたX線小角散乱・プロテアーゼによる限定分解実験から、CCTの“ふた”を閉じるにはATP加水分解が必要であることを報告した19)。彼らは、従来の実験結果がAMP-PNPに混入しているATPによるものであり、グループ2型シャペロニンの“ふた”の開閉はATPの結合ではなく、加水分解で制御されていると結論付けた。
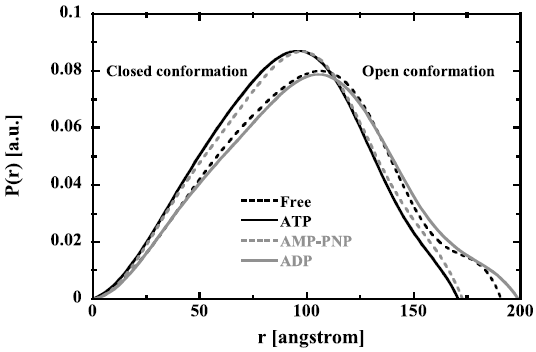
我々も、T. KS-1のシャペロニンを用いて同様の解析を行った。AMP-PNPを含む様々なアデニンヌクレオチドによるプロテアーゼ切断パターンとHelical rotrusion に導入したトリプトファンの蛍光の変化から、ATPの結合により構造変化が起こることを明らかにした20)。さらに、X線小角散乱による解析を行い、ATP結合型はヌクレオチドフリー型やADP結合型に比べ、よりコンパクトな構造をしており、T. KS-1シャペロニンは、ATP結合により“ふた”が閉じるという結論に達した (図4)21)。これらの実験結果は、ATPを完全に除去したAMP-PNPを用いても変わらなかったことから、真核型と古細菌型との間には、構造変化の引き金となるのがATP結合もしくは結合後のATP加水分解と相違があると考えた方が正しいだろう。

我々は、Helical Protrusionの機能についても解析を行い、この領域がないとOpen型からClosed型への構造変化ができなくなること、変性タンパク質との結合能はあるが、ATP依存的フォールディング活性が無くなることを明らかにした21)。我々が提唱している古細菌型シャペロニンの反応モデルを図5に示したが、まだ不完全なものであり、Helical Protrusionの構造変化における役割やもう一方のリングの機能など解明するべき課題は多く残っている。特に、“ふた”の開閉機構の解明には“ふた”が開いたOpen型構造の解明が不可欠である。T. KS-1 由来シャペロニンに関しても、“ふた”を閉じることができない変異体など様々な結晶の解析を行っているが、全て“ふた”が閉じたClosed 型構造であった22)。また、他のグループも成功したという報告はない。結晶化に用いる沈殿剤により、Open型からClosed型への構造変化が誘導されることが原因のようである。

4.プレフォルディンとの協同機構
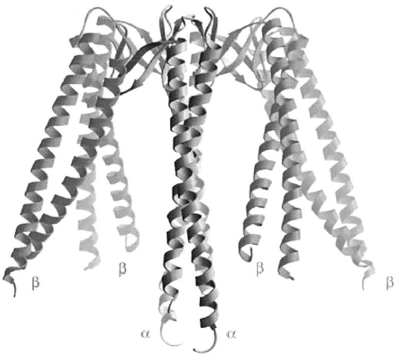
プレフォルディンは、酵母のサイトゾルにおいて細胞骨格の形成に必要な遺伝子GIM (Genes Involved in Microtuble Biogenesis) として同定され23)、変性βアクチンに結合し真核生物の2型シャペロニンCCTに受け渡す機能を有することからプレフォルディンと命名された24)。真核生物のプレフォルディンは2つのαタイプと、4つのβタイプの互いに配列の異なる6つのサブユニットで構成される。一方、古細菌プレフォルディンはαとβの2種類のサブユニットからなるα2 β4という比較的シンプルなサブユニット構造をしている。2000年に解明されたメタン生成古細菌Methanobacterium thermoautotrophicum由来プレフォルディン (MtGimC) のX線結晶構造によると、プレフォルディンはクラゲのような形をしており、2つのバレル構造から6つの触手のようなコイルドコイルが突き出している (図6)25)。基質タンパク質やシャペロニンとの結合はコイルドコイルの先端部分で起こっていると考えられている。2002年には、哺乳類由来のプレフォルディンと基質複合体の電子顕微鏡像が得られた。構造はMtGimCと同様であり、基質はクラゲの足の溝の部分に相当するコイルドコイル間に結合していた26)。

我々は超好熱性古細菌Pyrococcus horikoshii由来プレフォルディンを用いて、プレフォルディン、シャペロニン間の協調機構や結合のキネティックスに関する研究を進めている。
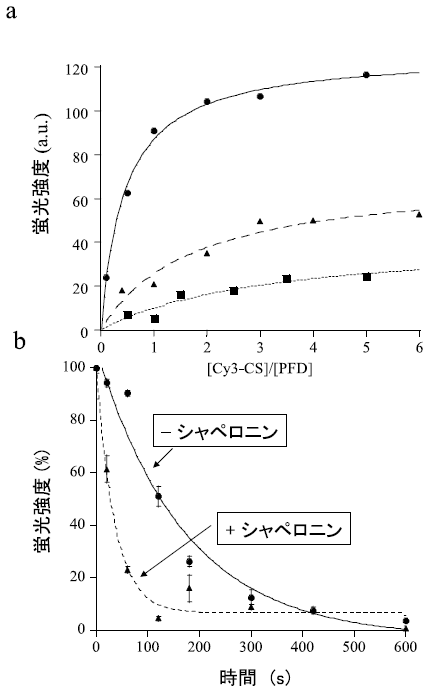
超好熱性古細菌Pyrococcus horikoshii由来プレフォルディンは、ブタ心臓由来クエン酸合成酵素 (CS) の熱変凝集を阻害し、酸変性したGFPの自発的凝集を阻害する。また、GFPと結合したプレフォルディンに超好熱性古細菌由来のシャペロニンを共存させ、ATPを加えるとGFPのフォールディングが促進される27)。以上のことから、プレフォルディンが変性中間状態のタンパク質を捉え、シャペロニンに受け渡すことが機能的に示唆された。
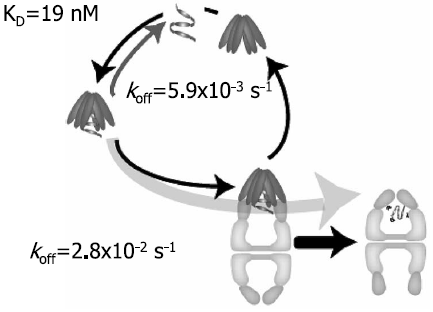
一方、実際にプレフォルディンとシャペロニンが物理的に相互作用していることは、免疫沈降、SPRセンサーによる解析、1分子イメージングなどにより証明している28)。さらに、シャペロニンとの協同機構解明のために、蛍光顕微鏡を用いたプレフォルディンと基質タンパク質の相互作用の解析を行った。プレフォルディンがガラスに強く結合することを利用し、蛍光標識した基質タンパク質の結合をシャペロニン存在、非存在下で蛍光顕微鏡により測定したところ、シャペロニン存在下では解離速度が4倍程度速くなった (論文投稿中) (図7)。この結果から、プレフォルディンとシャペロニンが協調してタンパク質のフォールディングを促進していることが証明された (図8)。


5.まとめ
GroELなどの1型シャペロニンと比較して2型シャペロニンの研究は著しく遅れていたが、優れたモデル系であるT. KS-1由来シャペロニンの発見により、反応機構の解明に近づくことができた。しかし、上述したように未解明の問題が多く残されている。特に、in vivoにおける機能はほとんど手付かずの状態である。また、真核生物の2型シャペロニンCCTは細胞骨格などの形成に重要な機能を担っているが、非常に複雑な構造をしているため、詳細な分子機構の解明は非常に困難である。古細菌の2型シャペロニンの構造や機能の解析がCCTの反応機構の解明に大きく寄与することも期待できる。
謝辞
今回紹介した我々の研究成果は、海洋科学技術センター丸山正博士、産業総合研究所石井則行研究員、京都大学三木邦夫教授、庄村康人博士、東京大学桑島邦弘教授、早稲田大学船津高志教授、東京農工大学大河内美奈博士らとの共同で行ったものであり、改めて感謝の意を表します。
文献
1) Bult, C. J., White, O., Olsen, G. J., Zhou, L., Fleischmann, R. D., Sutton, G. G., Blake, J. A., FitzGerald, L. M., Clayton, R. A., Gocayne, J. D., Kerlavage, A. R., Dougherty, B. A., Tomb, J. F., Adams, M. D., Reich, C. I., Overbeek, R., Kirkness, E. F., Weinstock, K. G., Merrick, J. M., Glodek, A., Scott, J. L., Geoghagen, N. S., Venter, J. C.: Science, 273, 1058 (1996).
2) Trent, J. D., Nimmesgern, E., Wall, J. S., Hartl, F. U., Horwich, A. L.: Nature, 354, 434 (1991).
3) Kim, S., Willison, K. R., Horwich, A. L.: Trends Biochem. Sci., 19, 543 (1994).
4) Guagliardi, A., Cerchia, L., Moracci, M., Rossi, M.: J. Biol. Chem., 275, 31813 (2000).
5) Ditzel, L., Lowe, J., Stock, D., Stetter, K. O., Huber, H., Huber, R., Steinbacher, S.: Cell, 93, 125 (1998).
6) Kagawa, H. K., Osipiuk, J., Maltsev, N., Overbeek, R., Quaite-Randall, E., Joachimiak, A., Trent, J. D.: J. Mol. Biol., 253, 712 (1995).
7) Yoshida, T., Yohda, M., Suzuki, M., Yazaki, K., Miura, K., Endo, I.: Biochem. Biophys. Res. Commun., 242, 640 (1998).
8) Trent, J. D., Kagawa, H. K., Yaoi, T., Olle, E., Zaluzec, N. J.: Proc. Natl. Acad. Sci. USA, 94, 5383 (1997).
9) Trent, J. D., Kagawa, H. K., Paavola, C. D., McMillan, R. A., Howard, J., Jahnke, L., Lavin, C., Embaye, T., Henze, C. E.: Proc. Natl. Acad. Sci. USA, 100, 15589 (2003).
10) Yoshida, T., Yohda, M., Iida, T., Maruyama, T., Taguchi, H., Yazaki, K., Ohta, T., Odaka, M., Endo, I., Kagawa, Y.: J. Mol. Biol., 273, 635 (1997).
11) Izumi, M., Fujiwara, S., Takagi, M., Kanaya, S., Imanaka, T.: Appl Environ Microbiol., 65, 1801 (1999).
12) Kagawa, H. K., Yaoi, T., Brocchieri, L., McMillan, R. A., Alton, T., Trent, J. D.: Mol. Microbiol., 48, 143 (2003).
13) Yoshida, T., Ideno, A., Hiyamuta, S., Yohda, M., Maruyama, T.: Mol. Microbiol., 39, 1406 (2001).
14) Yoshida, T., Ideno, A., Suzuki, R., Yohda, M., Maruyama, T.: Mol. Microbiol., 44, 761 (2002).
15) Usui, K., Yoshida, T., Maruyama, T., Yohda, M: J. Biosci. Bioeng., 92, 161 (2001).
16) Sakikawa, C., Taguchi, H., Makino, Y., Yoshida, M.: J. Biol. Chem., 274, 21251 (1999).
17) Yoshida, T., Kawaguchi, R., Taguchi, H., Yoshida, M., Yasunaga, T., Wakabayashi, T., Yohda, M., Maruyama, T.: J. Mol. Biol., 315, 73 (2002).
18) Gutsche, I., Holzinger, J., Rauh, N., Baumeister, W., May, R. P.: J. Struct. Biol., 135, 139 (2001).
19) Meyer, A. S., Gillespie, J. R., Walther, D., Millet, I. S., Doniach, S., Frydman, J.: Cell, 113, 369 (2003).
20) Iizuka, R., Yoshida, T., Shomura, Y., Miki, K., Maruyama, T., Odaka, M., Yohda, M.: J. Biol. Chem., 278, 44959 (2003).
21) Iizuka, R., So, S., Inobe, T., Yoshida, T., Zako, T., Kuwajima, K, Yohda, M.: J. Biol. Chem., 279, 18834 (2004).
22) Shomura, Y., Yoshida, T., Iizuka, R., Maruyama, T., Yohda, M., Miki, K.: J. Mol. Biol., 335, 1265 (2004).
23) Geissler, S., Siegers, K., Schiebel, E.: EMBO J., 17, 952 (1998).
24) Vainberg, I. E., Lewis, S. A., Rommelaere, H., Ampe, C., V ekerckhove, J., Klein, H. L., Cowan, N. J.: Cell, 93, 863 (1998).
25) Siegert, R., Leroux, M. R., Scheufler, C., Hartl, F. U., Moarefi, I.: Cell, 103, 621 (2000).
26) Martin-Benito, J., Boskovic, J., Gomez-Puertas, P., Carrascosa, J. L., Simons, C. T., Lewis, S. A., Bartolini, F., Cowan, N. J., Valpuesta, J. M.: EMBO J., 21, 6377 (2002).
27) Okochi, M., Yoshida, T., Maruyama, T., Kawarabayasi, Y., Kikuchi, H., Yohda, M.: Biochem. Biophys. Res. Commun., 29, 769 (2002).
28) Okochi. M., Nomura, T., Zako, T., Arakawa, T., Iizuka, R., Ueda, H., Funatsu, T., Leroux, M., Yohda, M.: J. Biol. Chem., 279, 31788 (2004).