【トピックス】
柱状構造高感度DNAチップの開発と酵母研究への応用
信正 均、秋山英雄
東レ㈱
1.はじめに
バイオ関連の基礎研究に一般的な分析手段となりつつあるDNAチップは、多数の遺伝子情報を一斉に解析できる遺伝子関連研究支援ツールとして、特にゲノム関連の基礎研究に大いに寄与している1-9)。その市場は近年急速に成長し、現在、数百億円規模と言われている。そのため、種々の分野の研究機関やメーカーで、DNAチップが開発され、製品化されている。特に、テーラーメイド医療や創薬開発、環境検査、食品検査・工程管理などで市場が飛躍的に拡大すると期待されている10-14)。しかし、現状では、上記の基礎研究用途が主であり、その他の応用領域ではほとんど実用に至っていない。
その理由の一つとして、既存のDNAチップでは、感度や再現性、定量性が必ずしも十分ではないことが挙げられる。特に、初期のころ、スポットされたプローブが一部はがれてスポット形状が安定しなかったり、スポットの並びが揃わなかったり、ノイズ成分が高い、というような課題があった。また、感度が十分でないため、例えば、種々の遺伝子情報を一斉に検出できるアレイを用いているにもかかわらず、高発現遺伝子の発現変動の解析結果しか得られず、低発現遺伝子の発現変動を議論することができない、というような課題もあった。また、医療現場において検査や診断に用いられることを考えた場合、患者に負担が少ない低侵襲な微量検体による解析が難しく、さらに、極僅かな遺伝子の存在に関する精度の高い解析についても検出感度が十分とは言い難い状況であった。
このような課題に関し、一般ユーザーにとってブラックボックス的な統計処理によりデータの精度を高めようとする方法や検体の増幅を繰り返すことにより検出できるように工夫する方法などが試みられているが、解析アルゴリズムの複雑化やバイアスの課題等が懸念されている。
ここでは、現在主流となっている蛍光検出技術をベースに、これまでのデータや主なプロトコール、さらにはシステム面の資産を活かすことができ、チップを替えるだけで従来チップ技術の最高で100倍の高感度が実現できる新しいDNAチップ技術を開発したので、その概要と高感度チップを用いた酵母発現解析の例を記す。
2.高感度チップの特徴
今回開発した高感度化DNAチップは次の3つの特徴を有している。
(1) チップ形状・材質による検出スポット形状の安定化とノイズ低減
(2) チップに固定するDNA (プローブDNA) の密度制御
(3) ターゲットDNAとの反応性向上
具体的には、DNAチップ基板をこれまでの平坦な形状から凹凸形状にする画期的なアイデアと、基板表面をナノレベルの特殊加工で活性化する技術および解析のための反応 (ハイブリダイゼーション) を強制的に促進させる技術の開発により、従来のDNA チップに比べ、最高で100倍の検出感度が達成できる技術の開発に成功した15-18)。
このことは、それまで不可能であった低発現遺伝子の発現変動の議論や極微量の検体からの解析を可能とすることを示唆する。例えば、このような技術を用いて網羅的な研究用DNAチップが開発できれば、(特に) 低発現域の遺伝子発現情報を高精度に検出できることから、基礎研究において新しい発見が期待されるだけでなく、上述した極僅かな遺伝子の存在を、網羅的かつ高精度に検出できる。また、応用面では、内視鏡技術で微量の検体を採取するだけで、迅速かつ正確な検査結果が得られる可能性が期待され、患者QOL (Quality of Life) の改善、医療費削減の観点から、早急な展開が期待される。
上記 (1)~(3) の高感度DNAチップの特徴について、その詳細を次に記す。
2-1 チップ形状・材質による検出スポット形状の安定化とノイズ低減
従来、DNAチップの基板としては平坦なガラス基板を用いるのが一般的である。スポット形状安定化と検出システムとの関係から、より平坦な基板を開発する方向にある。平坦なガラス基板上にプローブDNAをスポットし静電結合や共有結合により基板への固定化を向上させている。
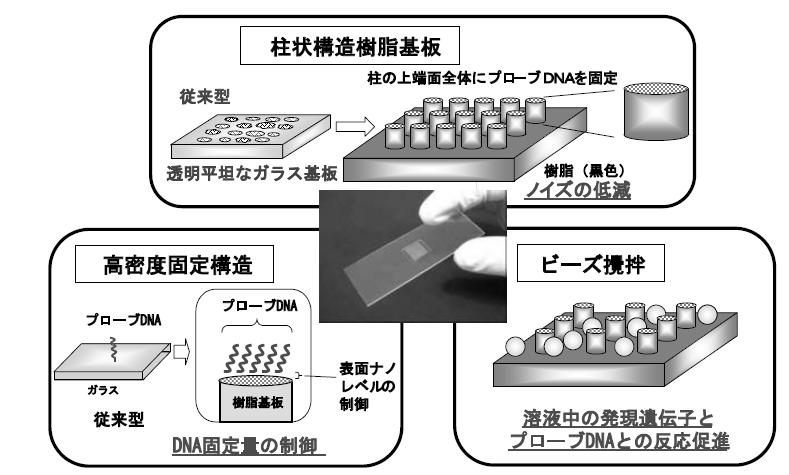
しかし、東レでは、平坦な基板ではなく、検出部に凹凸構造を持たせた革新的な柱状構造チップを考案した。この柱の上端面にプローブDNAを固定することにより、スポットの形状と整列の安定化が期待される。このような微細構造を有するチップを開発するにあたり、その基材としては、加工が容易で、かつ材料自体が有する自家蛍光が小さな合成樹脂を用いた。図1には、このようにして作製した約250のスポットの柱状構造チップの写真を示す。チップの基板表面に、直径、高さとも数十~数百μmの凹凸構造が施されている。図2に様々な種類のプローブDNAを凹凸構造の上端面にスポットし、ターゲットDNAと反応させた後のスポット形状の検出イメージを従来型チップの場合と比較して示す。検出には、(一部を除く) 市販のDNAチップ検出装置を用いることができる。このイメージから、柱状構造DNAチップでは安定したスポット形状でシグナルを観察できることが確認され、柱の上端面全体にDNAが固定、スポット内の分布の均一性が優れていることがわかる。一方、平坦なガラス基板にスポットした場合、スポット形状にバラツキが観察される。また、合成樹脂からなる柱状構造チップでは、材料自体の自家蛍光を制御でき、市販の従来型DNAチップ (平坦なガラス基板) の場合と比較して、スポットおよびスポット周辺のノイズが数分の一に減少できることが観察された。このように、柱状構造チップを用いることにより、スポットの形状が安定化できるばかりでなく、ノイズも低減できることが確認され、精度の高い検出が可能になると期待される。
図1 柱状構造高感度DNAチップの形状
図2 検出結果の比較
なお、樹脂製基板を、精度高く微細加工するためには多くのノウハウを必要とする。検査・診断用で想定される数百程度までのスポットのチップだけではなく、研究用の網羅的な遺伝子解析に適用できる数万のスポットのチップの作製技術も開発できている。この網羅的なDNAチップにより、高感度性能を実証し、実績を蓄積して、今後、検査・診断用チップとして用いられていくことが期待される。
2-2 チップに固定するDNA (プローブDNA) の密度制御
上述のとおり、今回開発した合成樹脂からなる柱状構造チップでは、表面加工・修飾の自由度が高く、柱状構造の上端面のナノレベルの設計を行うことによりDNAとの相互作用を制御する技術が開発できる。その結果、ターゲットDNAとのハイブリダイゼーション効率が最も良好な密度でプローブDNAを固定させることに成功している。また、DNAとの結合様式を種々設計できることを利用して、非特異なDNAの吸着抑制に有効な表面を実現している。これらの表面設計技術は、シグナルを向上させるとともにノイズレベルを低減させ、検出感度向上に有効である。
2-3 ターゲットDNAとの反応性向上
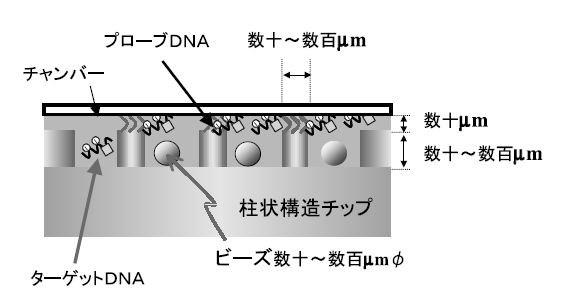
一般的にDNAチップでは、反応時にターゲット溶液中のDNA の拡散が遅く (~200μm19))、希薄な溶液を用いた場合や低発現の遺伝子を検出しようとした場合、十分な反応効率が期待できない。そこで、反応時にターゲットDNAの拡散を物理的に加速するコンセプトで検討を進めた。その結果、ビーズによる攪拌を考案し、上述の柱状構造、表面のナノレベルの設計と組み合わせることにより、プローブDNAには何等影響を与えることなく効果的に反応を促進できる技術を開発した (図3)。
図3 柱状構造高感度DNAチップの特徴
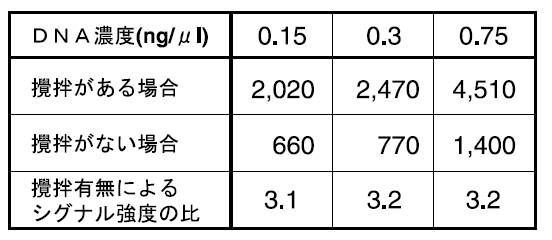
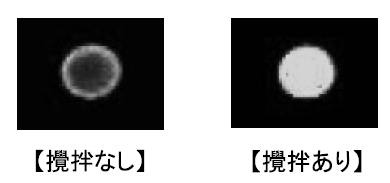
すなわち、図4に示すように、ハイブリダイゼーション時にターゲットDNAと一緒に直径数十~数百μmの大きさのビーズをチャンバー内に封入してチップ全体を振とうし、ビーズを動かしてハイブリダイゼーション液の攪拌を行う。このようにハイブリダイゼーション液を攪拌することにより、静置時に比べて効率的にターゲットDNAをプローブDNAと会合でき、ハイブリダイゼーションを格段に加速し、シグナル強度を大幅に向上できる (表1)。また、スポット間の反応均一性およびスポット内の反応均一性も格段に向上させることに成功した (図5)。この時、プローブDNAがスポットされている柱状構造の上端面とカバーグラスの間にビーズが侵入しないような設計を行うことにより、プローブDNAが傷つけられることはない。
図4 柱状構造とビーズ攪拌 (断面図)
表1 攪拌によるシグナル強度の向上
図5 柱状構造とビーズ攪拌 (断面図)
この反応性向上手法は、検査・診断用で想定される数百程度までのスポットのチップの場合だけでなく、基礎研究などに用いられる数万のスポットが形成された網羅的なチップの場合でも、柱状構造の形状とレイアウト、および攪拌プロトコールを工夫することにより、同様の効果が確認できている。
3.柱状構造DNAチップの性能評価
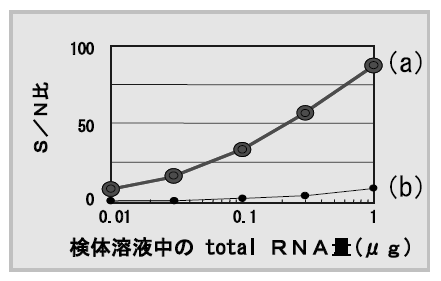
上記3つの技術をベースに作製したDNAチップの性能を以下に記す。図6にターゲットとして用いるtotal RNAの濃度を変化させた場合のシグナル/ノイズ比 (S/N比) を示す。また、図7に、0.01~1.0μg相当のtotal RNAを用いた場合の従来型チップと今回開発した柱状構造DNAチップの検出イメージを示す。従来の平坦なガラス製DNAチップと比較して、顕著なシグナルの上昇およびノイズ低減が観察された。従来型DNAチップでは0.01μg相当以下ではシグナル検出が困難であるにもかかわらず、今回開発した柱状構造DNAチップでは検出が可能であり、従来の平坦なガラス基板によるDNAチップと比較して、約1/100 のターゲットDNA量でもシグナルが検出できることが確認された。

![]()
この結果から、検査・診断応用を考えた場合、耳掻き1杯程の組織検体や極微量の血液からでも遺伝子の発現変動が検出できることが確認された。また、検出感度が大幅に向上できたことから、再現性や低発現遺伝子の検出における定量性も従来のチップに比べ飛躍的に優れた性能が得られている。
4.酵母全遺伝子型DNAチップを用いた遺伝子発現解析
柱状構造からなる酵母全遺伝子型チップ“3D-Gene Yeast Oligo chip 6k”を用いた遺伝子発現解析の例を記す。本チップにはSaccharomyces Genome Database (SGD) に登録された5,888遺伝子に対するプローブが搭載され、出芽酵母Saccharomyces cerevisiae の遺伝子発現を網羅的に解析できる。
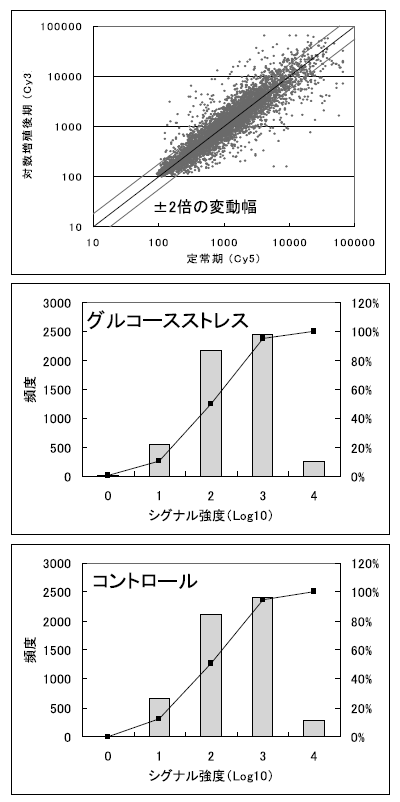
まず、酵母株を5 mlのYPAD培地で一晩、30℃で振とう培養 (前培養) した後、OD660=0.1になるように150 mlのYPAD培地に植菌、30℃で振とう培養した (本培養)。OD660が0.6にまで増殖したところで、本培養液を10 mlづつ分配して集菌、滅菌水で洗浄した後、各々集菌した細胞に10 mlのグルコースを含まない培地で細胞を再懸濁し、30℃で15~30分間穏やかに振とうした。コントロールとして、YPAD培地で細胞を再懸濁し、同様に振とうした。この再懸濁から集菌し得られた細胞を発現解析用酵母細胞とし、Schmittらの方法20)にしたがいtotal RNAを調製、競合ハイブリダイゼーションによる発現解析を行った。図8に結果をまとめるが、本DNAチップは低シグナルから高シグナルまで4桁近くのダイナミックレンジがあること、特にガラス平板の従来型DNAチップではノイズにより検出困難な低発現な遺伝子 (約20%) が本DNAチップにより解析対象となることが確認された。
図8 酵母遺伝子の発現ヒストグラム
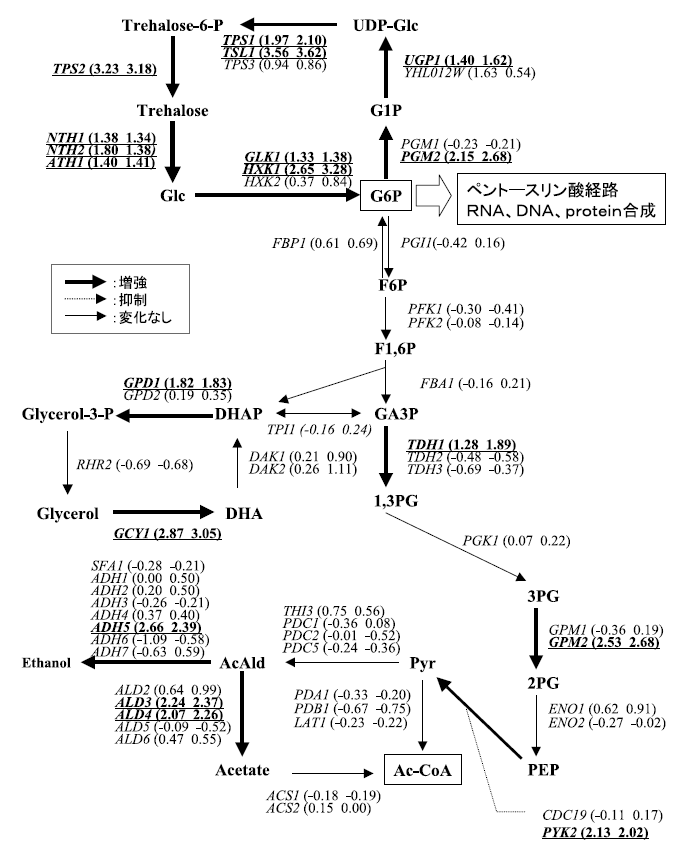
今回、対数増殖期においてグルコースストレスにより発現が特異的に誘導、もしくは抑制される遺伝子を中心に絞り込みを試みた。好気的条件下では、グルコースは解糖系、TCAサイクル (クエン酸回路)、電子伝達系等の代謝系によって二酸化炭素および水にまで分解され、ATPが発生する。当然、これらの代謝系に関与する酵素をコードする遺伝子の多くは高発現となる。一方グルコースが枯渇すると、グルコースキナーゼ (HXK1, HXK2, GLK1) やペントースリン酸経路に関与する遺伝子群、グルタミン酸脱水素酵素 (GDH1, GDH3) 等の発現が強く誘導されることが知られる21)。図9に糖代謝に関連する酵素の発現変化を網羅的に解析した結果を示すが、グルコースストレスによりグルコースキナーゼ遺伝子のうち低親和性グルコーストランスポーターHXK1が特異的に強く誘導されることがわかる。またPGMやADHに代表される相同性の高いタンパク質が存在するファミリー酵素についても特異的に機能する遺伝子が特定でき、プローブの高い特異性 (選択性) が確認された。さらにグルコース以外のストレス、例えばリン酸ストレスで発現誘導されることが知られるPHO関連遺伝子はYPAD条件で極低発現なため、従来型DNAチップでは検出困難とされるが、今回、数種類の遺伝子についてリン酸ストレスと同様に著しく発現誘導されることが確認できた。
図9 グルコースストレスで変動する遺伝子〔 ( ) 内は重複実験を示す〕
酵母は最も古くから研究されている生物の一つであり、個々の因子に関する知識が豊富で、またそれらを記述したデーターベースがよく整備されている。今後、高い特異性をもつプローブと100倍の高感度を実現した新しいDNAチップ技術の組み合わせで、極低発現な遺伝子の機能を網羅的に解明、データベースを充実化させることにより、真核細胞の「モデル生物」として世界中でより広範な分野で研究が行われると期待される。
5.今後の展開
現在、DNAチップの用途は多数のプローブDNA (1,000~50,000遺伝子) を搭載した網羅的解析用が主流である。高感度なDNAチップを用いることにより、これまでのDNAチップでは検出できなかった低発現な遺伝子、あるいは微量の遺伝子に関する情報が確度高く得られるようになり、遺伝子関連の研究に革新をもたらすものと期待される。既に、研究用の網羅的チップとして酵母の全遺伝子を搭載したチップが製品化されており、さらに、ヒトの全遺伝子を搭載したチップ、特定の疾患領域に関係する遺伝子を搭載したチップの製品化が進められている。また、各種カスタムチップの試作や解析も進められており、基礎研究領域だけでなく臨床研究、産業用途における実績も蓄積されつつある (ホームページURL[http://www.3d-gene.com])。
5.まとめ
以上のように、従来の技術の方向性 (平坦な形状) とは逆の斬新な形状 (柱状構造) とナノレベルの表面修飾、新規な反応促進技術を開発することにより、超高感度化技術を開発することに成功した。今後、このDNAチップ技術に様々な遺伝子情報を搭載し、このチップの特長を生かした領域で実績を蓄積し、非臨床および臨床分野で検査・診断ツールとして実用されることが期待される。
文献
1) Fordor, S. P. A., Read, J. L., Pirrung, M. C., Stryer, L., Lu, A. T., Solas, D.: Science, 251, 767 (1991).
2) DeRisi, J. L., Iyer, V. R., Brown, P. O.: Science, 278, 680 (1997).
3) Landegren, U., Nilsson, M., Kwok, P.-Y.: Genome Res., 8, 769 (1998).
4) Thiel, A. J.: Anal. Chem., 69, 4948 (1997).
5) Flaim, C. J., Chien, S., Bhatia, S. N.: Nat. Methods, 2, 119 (2005).
6) Maekawa, M., Yamamoto, T., Tanoue, T., Yuasa, Y., Chisaka, O., Nishida, E.: Development, 132, 1773 (2005).
7) Soronen, P., Laiti, M., Törn, S., Härkönen, P., Patrikainen, L., Li, Y., Pulkka, A., Kurkela, R., Herrala, A., Kaija, H., Isomaa, V., Vihko, P.: Bichem. Mol. Biol., 92, 281 (2004).
8) Jamshidi-Parsian, A., Dong, Y., Zheng, X., Zhou, H. S., Zacharias, W., McMasters, K. M.: Gene, 344, 67 (2005).
9) Daniell, H., Kumar, S., Dufourmantel, N.: Nature. Biotechnol., 23, 238 (2005).
10) Katsuma, S., Nishi, K., Tanigawara, K., Ishikawa, H., Shiojima, S., Takagaki, K., Kaminishi, Y., Suzuki, Y., Hirasawa, A., Ohgi, T., Yano, J., Murakami, Y., Tsujimoto, G.: Biochem. Biophys. Res. Commun., 288, 747 (2001).
11) Yamada, M., Katsuma, S., Adachi, T., Hirasawa, A., Shiojima, S., Kadowaki, T., Okuno, Y., Koshimizu, TA., Fujii, S., Sekiya, Y., Miyamoto, Y., Tamura, M., Yumura, W., Nihei, H., Kobayashi, M., Tsujimoto, G.: Proc. Natl. Acad. Sci. USA, 102, 7736 (2005).
12) van’t Veer, L. J., Dai, H., van de Vijver, M. J., He, Y. D., Hart, A. A., Mao, M., Peterse, H. L., van der Kooy, K., Marton, M. J., Witteveen, A. T., Schreiber, G. J., Kerkhoven, R. M., Roberts, C., Linsley, P. S., Bernards, R., Friend, S. H.: Nature, 415, 530 (2002).
13) Gerhold, D. L., Jensen, R. V., Gullans, S. R.: Nature Genetics, 32, 547 (2002).
14) Margalit, O., Somech, R., Amariglio, N., Rechavi, G.: Blood Rev., 19, 223 (2005).
15) 2004年9月17日付, 日本経済新聞朝刊.
16) 2004年9月24日付, 日本経済新聞朝刊.
17) BOSS 11月22日号, ㈱経営塾発行, 71 (2004).
18) Nagino, K., Nomura, O., Takii, Y.: The Journal of Biochemistry, 139, 697 (2006).
19) Politz, J. C., Browne, E. S., Wolf, D. E., Pederson, T.: Proc. Natl. Acad. Sci. USA, 95, 6043 (1998).
20) Schmitt, M. E., Brown, T. A., Trumpower, B. L.: Nucl. Acids Res., 18, 3091 (1990).