【トピックス】
酵素触媒機構データベースEzCatDB
長野希美
産総研・生命情報
1.はじめに
従来の酵素の分類であるEC 番号は、主に基質・産物の化学構造や触媒反応に関わる補酵素などに基づいて分類が行われており1)、触媒機構において重要である蛋白質の配列情報や立体構造に関する情報が全く考慮されていない。例えば、異なるドメイン構造やスーパーファミリーに属する酵素でも、類似の反応を触媒することもあるのに対して、同じ配列ファミリーに属する酵素でも異なる機構で触媒機能を担うこともある。EC番号では、そのような詳細な触媒機構を反映することは難しい。また、酵素によっては1種類の反応だけでなく複数の基本反応 (加水分解反応、転移反応、解離反応など) を組み合わせた複雑な反応を触媒することもあり、そのような場合も一つのEC番号だけで触媒機構を表現するのは困難である。
他方で、酵素蛋白質の立体構造のデータは、現在Protein Data Bank (PDB)2)に1万8000エントリー以上登録されているが、PDBの酵素立体構造データには、蛋白質のみならず、基質・産物・補酵素あるいは反応中間体そのもの、あるいはそのアナログ化合物や遷移状態のアナログなど重要なリガンド情報も多数含まれており、PDBそのものが創薬を行う上でリード化合物候補の宝庫となっていると考えられる。それにも関わらず、PDBを含めて従来の立体構造関連のデータベースでは、酵素蛋白質とリガンドとの関係がアノテーションされていない。
こうした点を踏まえて、著者らは酵素触媒機構データベースEzCatDB を構築し、酵素蛋白質の触媒部位やリガンドとの対応関係などをアノテーションし、酵素触媒反応分類などを行っている (URL: http://mbs.cbrc.jp/EzCatDB/)3,4)。本稿では、特に酵素触媒反応分類について紹介する。
2.触媒反応分類・RLCP
EzCatDB では、酵素とリガンドの反応部位に特に注目し、酵素触媒機構の系統的な分類であるRLCP を開発しており、一般公開している (URL:http://mbs.cbrc.jp/EzCatDB/)3,4)。
この分類データベースでは、 (1) 基本反応[R]、 (2) リガンドの反応部位の構造[L]、 (3) 触媒機構の種類[C]、 (4) 酵素側の触媒残基、補酵素の種類[P]、というようにRLCP という4項目がEzCatDB 独自の分類基準となる。この分類では、スーパーファミリーを超えて類似する反応を同じグループに分類していることも特徴である。例として、プロリン・イミノペプチダーゼという酵素の反応は基本反応 (Reaction) は「加水分解 (Hydrolysis) 」、リガンド (Ligand) の反応部位の構造は「アミド結合 (amide bond) 」、触媒機構 (Catalysis) の種類は「Trypsin型触媒機構」、酵素活性部位 (Protein) の種類は「Ser/His/Asp + mainchain amide」というように分類されている。
3.加水分解、転移反応の触媒機構の分類
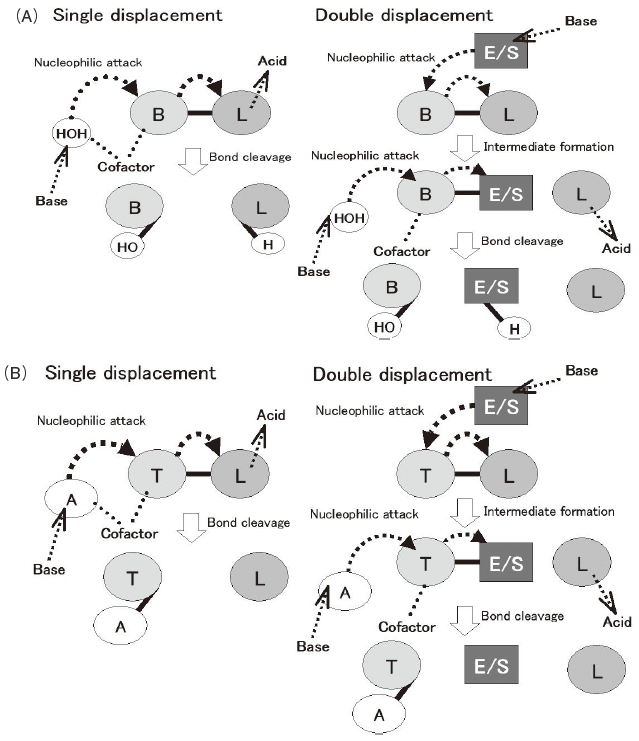
本稿では、特に加水分解反応、転移反応の触媒機構の分類について紹介する。図1に示すように、加水分解反応、転移反応は定義できる。加水分解反応は、「共有結合」を水分子で切断する反応であり、転移反応は、「脱離基」から「受容基」へ「転移基」を移す反応である。

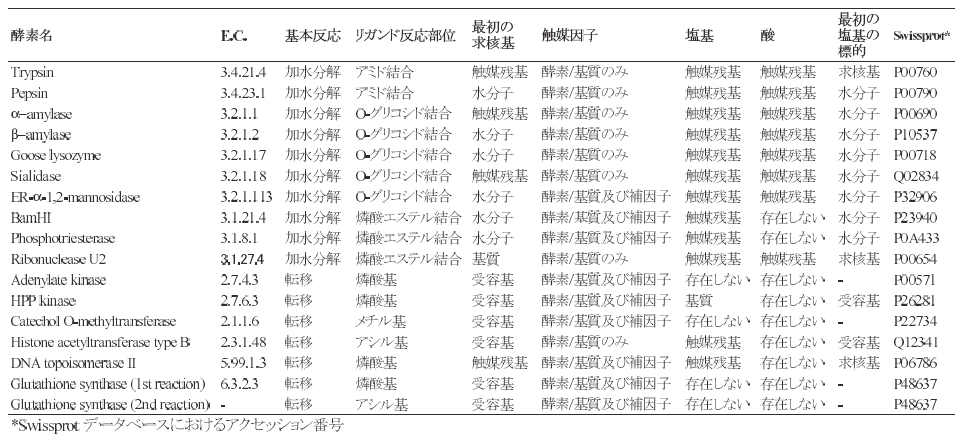
図1に示すように、加水分解反応、転移反応は、いずれも置換反応の組み合せであり、必ず1つ以 上の求核基が存在し、加水分解の場合、そのうちの一つは、水分子である。加水分解反応、転移反応は、いずれも次の2種類に大別できる。求核基が1つしかないsingle displacement 機構と2つの求核基が存在するdouble displacement機構である。加水分解反応の場合、single displacement機構では、水分子が直接、標的となる共有結合を求核攻撃し、反応が終結する。ペプシン5)、β-アミラーゼ6)などの触媒機構が、single displacement 機構に分類できる。それに対して、double displacement 機構では、まず、触媒残基か基質のある部位が最初の求核基として標的の共有結合を求核攻撃し、新しい共有結合をもつ中間体を形成する。次のステップで、水分子がこの新しい結合を求核攻撃することで加水分解反応が終結する。トリプシン7)、α-アミラーゼ8)などの触媒機構が、double displacement機構に分類できる。更に、図1に示すように、これらの触媒機構を更に多様化させる触媒因子 (塩基、酸、補酵素など) が多数存在しており、触媒反応を促進している。ここで、触媒機構を、触媒因子や基質の間の分子内、分子間ネットワークとして捉えることができることが分かる。こうした触媒因子やそれらの標的因子をアノテーションすることによって加水分解反応の触媒機構は記述でき、相互に比較することができる (表1)。
表1 主要な酵素に対する触媒因子とそれらの関係について
転移反応は、加水分解反応と類似している。水分子の代わりに基質の受容基が求核基として働き、転移基に求核攻撃をすることによって新しい共有結合を形成する。したがって、転移反応の触媒機構は加水分解反応の場合と同様の方法で記述できる。
上記のように、触媒因子は互いに相互作用するが、RLCPでは、Bartlett 等の定義9)に更に修正を加えた定義で分類を行っている3,4)。表1に代表的な酵素とその触媒因子の概要を示している。
4.頻繁に観られる酵素反応の特徴
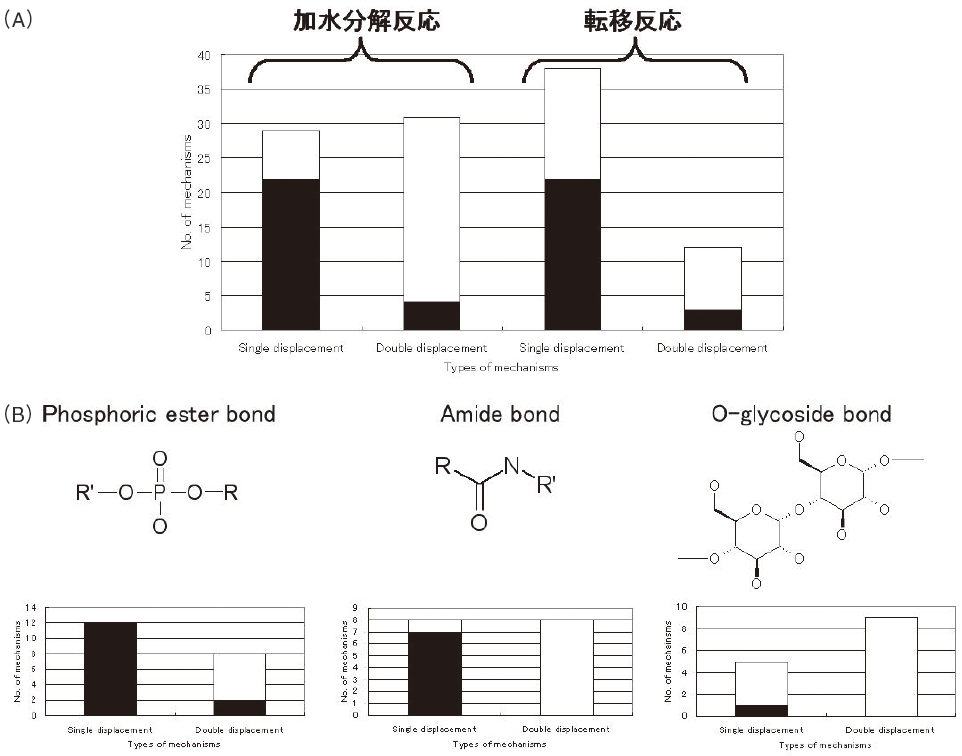
270種類の酵素に関して上記のような触媒機構の分類を行ったところ、これまでに、加水分解反応に関しては60種類の触媒機構、転移反応に関しては50種類の触媒機構と、合計110種類の触媒機構が見つかってきている4)。転移反応に関しては、図2A に示すように、single displacement機構の方が、double displacement機構よりも多く観られ、加水分解反応では、いずれの機構も同程度観られる。更に、加水分解、転移反応の両方の反応において、マグネシウムイオンや亜鉛イオンのような無機系の補因子が、double displacement機構よりもsingle displacement 機構で頻繁に使われることも判明した (図2A参照)4)。

加水分解反応では、リン酸エステル結合、アミド結合、O-グリコシド結合が主要な標的共有結合となっている。したがって、こうした結合に対しては、触媒機構の種類も数多く存在している。図2Bに示すように、リン酸エステル結合とアミド結合の加水分解では、類似の傾向を示すのに対して、O-グリコシド結合の加水分解反応は、全く異なる挙動を示している。例えば、リン酸エステル結合とアミド結合の加水分解では、single displacement 機構では殆どの場合マグネシウムイオンや亜鉛イオンのような補因子が使われるのに対し、double displacement機構では殆ど使われない。それに対して、O-グリコシド結合の加水分解反応では、single displacement機構でも、殆どそのような補因子が使われない。こうした傾向の違いは、リン酸エステル結合とアミド結合がエステル結合であるのに対して、O-グリコシド結合は、非エステル結合であることを反映している可能性がある4)。
このように、多くの触媒反応を比較、解析することによって、触媒機構の傾向が見えてくると考えられる4)。
5.基本反応から複雑な反応へ
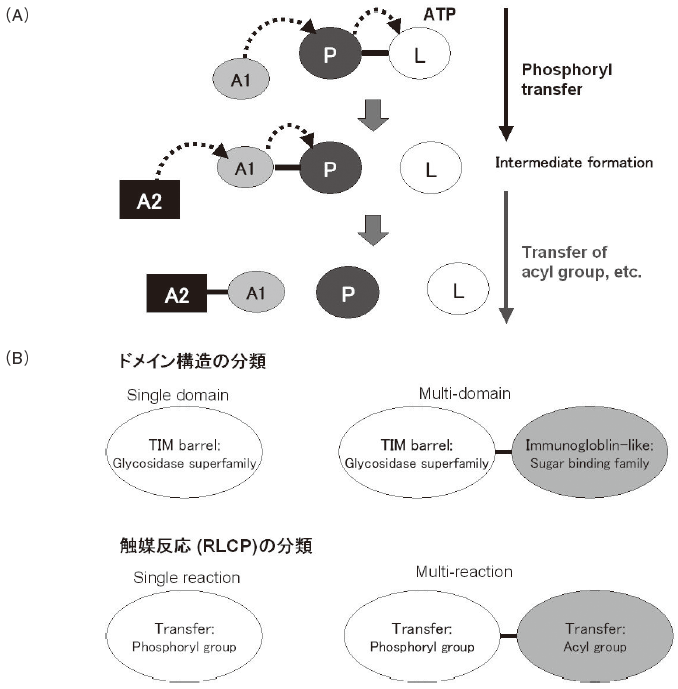
殆どの加水分解反応や転移反応は、それぞれ加水分解酵素 (E.C. 3.-.-.-) や転移酵素 (E.C. 2.-.-.-) に触媒されるが、リガーゼ (E.C. 6.-.-.-) など他の酵素類が、こうした反応を触媒することもある。図3Aに示すように、リガーゼ類の多くは、2つの転移反応を次々と触媒している。例えば、グルタチオン合成酵素 (E.C. 6.3.2.3) の場合、ATP からアシル基にリン酸基を転移し、そのアシル基をリン酸基から別の受容基に転移する10,11)。この酵素の活性部位の構造を観ると、リン酸転移酵素の一つであるHPP キナーゼ (E.C. 2.7.6.3) の活性部位12)とアシル転移酵素の一つであるヒストン・アセチル転移酵素 (E.C. 2.3.1.48) の活性部位の構造13)の特徴を併せ持つことが分かる4)。上記のRLCP分類では、グルタチオン合成酵素のような酵素には、2種類の反応分類のクラスが割り当てられている3,4)。
それに対して、加水分解酵素、転移酵素に分類される酵素でも、必ずしも、それぞれ加水分解反応、転移反応を触媒するとは限らない。例えば、アデノシン・デアミナーゼ (E.C. 3.5.4.4) のような酵素は、加水分解酵素に分類されているが、実際には水の付加反応と脱離反応を触媒し、水酸基ではなくカルボニル基を生成している14)。したがって、RLCP分類では、これらの酵素反応は、加水分解反応のカテゴリーからは除かれている4)。
こうした反応分類、RLCP 分類は、SCOP、CATHといった蛋白質のドメイン構造の階層分類と類似していると考えられる4)。ドメイン構造分類では、「シングル・ドメイン」が分類の単位となっている。図3Bに示すように、複数のドメインから構成される「マルチ・ドメイン」蛋白質に対しては、ドメイン毎にクラスが割り当てられている。RLCP分類でも複雑な反応はいくつかの基本反応に分割されて、それぞれ分類される。したがって、多くの酵素は「シングル・リアクション」を触媒するが、「マルチ・リアクション」を触媒する酵素も存在する (図3B参照)4)。

6.おわりに
系統的な酵素触媒反応分類を行うことによって、酵素の活性部位の構造と触媒反応の相関関係を見出せると考えられる4)。
また、EzCatDB は、現在は酵素触媒反応分類に重点を置いているが、酵素触媒機構を三次元的に再現したモデルの構築や中間体化合物などの解析も包括し、将来的には酵素の触媒機構から創薬を支援するデータベースに発展させていく予定である。
謝辞
本研究を推進するにあたり、酵素データのアノテーションにご協力頂いた長谷川祐子氏、中川善一氏、野中啓太郎氏に感謝します。
また、本研究は、科学技術振興機構・さきがけ (PRESTO)、独立行政法人日本学術振興会・科学研究費補助金・研究成果公開促進費、科学技術振興機構・バイオインフォマティクス推進事業 (BIRD) を受けて行われました。心より感謝の意を表します。
文献
1) Webb, E. C.: Enzyme Nomenclature 1992, Academic Press Inc. (1992).
2) Berman, H. M., Westbrook, J., Feng, Z., Gilliland, G., Bhat, T. N., Weissig, H., Shindyalov, I. N., Bourne, P. E.: Nucleic Acids Res., 28, 235 (2000).
3) Nagano, N.: Nucleic Acids Res., 33, D407 (2005).
4) Nagano, N., Noguchi, T., Akiyama, Y.: PROTEINS: Structure, Function, and Bioinformatics., 66, 147 (2007).
5) Suguna, K., Padlan, E. A., Smith, C. W., Carlson, W. D., Davies, D. R.: Proc. Natl. Acad. Sci. USA, 84, 7009 (1987).
6) Miyake, H., Otsuka, C., Nishimura, S., Nitta, Y.: J. Biochem. (Tokyo) , 131, 587 (2002).
7) Sprang, S., Standing, T., Fletterick, R. J., Stroud, R. M., Finer-Moore, J., Xuong, N. H., Hamlin, R., Rutter, W. J., Craik, C. S.: Science, 237, 905 (1987).
8) Aghajari, N., Roth, M., Haser, R.: Biochemistry, 41, 4273 (2002).
9) Bartlett, G. J., Porter, C. T., Borkakoti, N., Thornton, J. M.: J. Mol. Biol., 324, 105 (2002).
10) Polekhina, G., Board, P. G., Gali, R. R., Rossjohn, J., Parker, M. W.: EMBO J., 18, 3204 (1999).
11) Dinescu, A., Cundari, T. R., Bhansali, V. S., Luo, J. L., Anderson, M. E.: J. Biol. Chem., 279, 22412 (2004).
12) Blaszczyk, J., Shi, G., Yan, H., Ji, X.: Structure Fold Des., 8, 1049 (2000).
13) Dutnall, R. N., Tafrov, S. T., Sternglanz, R., Ramakrishnan, V.: Cell, 94, 427 (1998).
14) Wilson, D. K., Rudolph, F. B., Quiocho, F. A.: Science, 252, 1278 (1991).