【トピックス】
FT-ICR/MS を利用した酵素の基質探索
古屋俊樹、木野邦器
早大・理工学術院
1.はじめに
DNAシーケンスの高速化、低コスト化が進み、米国ではヒトゲノムを1,000ドルで解読する1,000ドルゲノムプロジェクトが進められており、微生物に関してもすでに800を越える株数のゲノムが解読されている。今後、ますます加速する勢いで様々な生物のゲノムが解読されていくものと思われるが、ゲノム配列上の多くの遺伝子は機能未知であるため、その機能を解明するポストゲノム研究に重点が移行しつつある。酵素工学の観点からは、多くの機能未知の酵素遺伝子を含むゲノム配列は新規有用酵素の探索源として非常に魅力的である。探索方法として、ゲノム配列から既知酵素と配列相同性の高い酵素をin silicoで検索し、その遺伝子をクローニングして大腸菌等で発現させ、既知酵素の基質と類似する化合物に対して活性を評価して新規な性質 (基質選択性、位置・立体選択性、耐熱性等) を有する酵素を探索するというのが一般的である。しかしながら、多くの機能未知酵素は既知酵素と配列相同性が低いかないため、基質を予測するのは容易ではなく、結果として活性評価が困難である。
我々は、フーリエ変換イオンサイクロトロン共鳴質量分析装置 (FT-ICR/MS) を利用した機能未知酵素の基質探索法を考案した1)。FT-ICR/MSはエレクトロスプレーイオン化法等により発生させた化合物のイオンを高磁場中でサイクロトロン運動させ、質量電荷比 (m/z) に依存した波形をフーリエ変換して質量を測定する装置で、質量分解能と質量精度において格段に優れている2)。本手法では酵素を複雑な化合物混合溶液と反応させることにより基質となる化合物を探索するが、反応液の一斉解析にFT-ICR/MSが威力を発揮する。本稿では、FT-ICR/MSを利用した基質探索法によりP450モノオキシゲナーゼの基質を見出した例について紹介する。あわせて本手法の有効性および応用の可能性について言及したい。
2.本手法の戦略1)
FT-ICR/MSは質量分解能と質量精度において格段に優れている。質量分解能は100,000に達し、m/z 500では質量数差わずか0.005のマスピークも分離できることになる。これにより、質量分析装置の前段階で液体クロマトグラフィーにより成分を分離しなくてもマスピーク同士が重なる確率は低いため、質量分析装置へのダイレクトインジェクションによる分析が可能となる。また、質量精度は1 ppm以下に達し、m/z 500では0.0005より高い質量精度が得られることになり、装置の磁場強度にもよるが小数点以下第3~5位までの精密質量数を決定することができる。これにより、精密質量数に基づいて既知成分の同定、さらには未知成分の組成式の決定が可能となる。このような分析特性によりFT-ICR/MSでは複雑な多成分溶液の一斉解析を短時間かつ高い同定能で実現することができる。バイオの分野ではプロテオーム解析3)やメタボローム解析4)にも利用されている。
FT-ICR/MSを利用した酵素の基質探索法は、酵素を複雑な化合物混合溶液と反応させて反応液をFT-ICR/MSに直接導入し、一斉解析して基質となる化合物を探索するという非常にシンプルな方法である。図1にモノオキシゲナーゼをターゲットとした場合について示す。化合物混合溶液に対してモノオキシゲナーゼを含む反応液と含まない反応液を用意してFT-ICR/MS分析を行う。化合物Aが酸化されたとするとAのマスピークは酸素の質量数分だけシフトするので、酸化生成物を精密質量数に基づいて同定することができる。さらに、酸素同位体18O2存在下で反応を行うと、酸化生成物のマスピークは18Oと16Oの精密質量数差分だけシフトするため、これにより同定能を高めることができる。
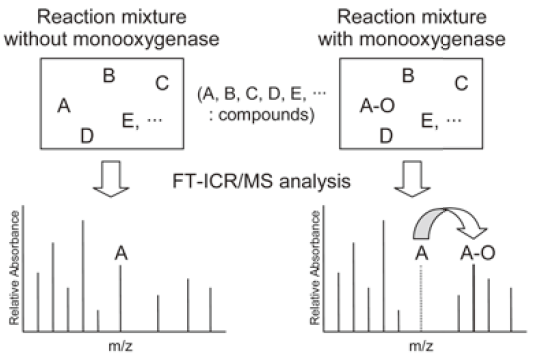
図1 FT-ICR/MSを利用したモノオキシゲナーゼの基質探索
3.P450モノオキシゲナーゼの基質探索1)
モノオキシゲナーゼは温和な条件下で位置・立体選択的に化合物に酸素を導入することができるため、触媒としての利用に期待が寄せられている5)。とくにP450モノオキシゲナーゼファミリーは動物、植物から微生物に至るまで多くの生物に遍在しており、機能においても多様性を示す有用な酵素群である。ここでは、Bacillus属のP450
をモデルとして基質探索に供した。B. subtilisおよびB. cereusのゲノムからその配列情報をもとにPCRを用いてそれぞれ8種類と5種類のP450遺伝子をクローニングし、大腸菌内で発現させた。さらに、無細胞抽出液の一酸化炭素結合スペクトルを測定し、13種類全てのP450に対して活性型P450に特徴的な450 nm付近の吸収を確認した。
一方、テルペノイド類を中心とする市販化合物を収集し、30化合物 (トリテルペン24化合物、ジテルペン2化合物、セスキテルペン2化合物、ポリケチド2化合物) のモデルライブラリーを作製して基質の探索源とした。その混合溶液 (各濃度3.3µM) を調製し、FT-ICR/MS分析を行った。使用した装置はFinnigan LTQ FT (Thermo社製、磁場強度7T) で、エレクトロスプレーイオン化法、ポジティブイオンモードにより分析を行った。その結果、30化合物のH+付加イオンおよびNa+付加イオンの精密質量数に相当するマスピークを検出することができた (図2)。構造異性体のピークを分離することはできないが、それ以外のピークの分離を確認した。例えば、酢酸メドロキシプロゲステロンのNa+付加イオン (m/z409.235) とコンパクチンのH+付加イオン (m/z409.259) は0.024の質量数差しかないが、ピークは分離していることがわかる (図2)。各成分のピーク強度の違いは主にイオン化効率の違いに起因する。また、アダクトイオン形成や溶媒、装置等由来のコンタミネーションに起因すると思われる未知ピークも検出されたが、これらを含むほとんど全てのピークを分離することができ、精密質量数に基づいて判別することが可能であった。
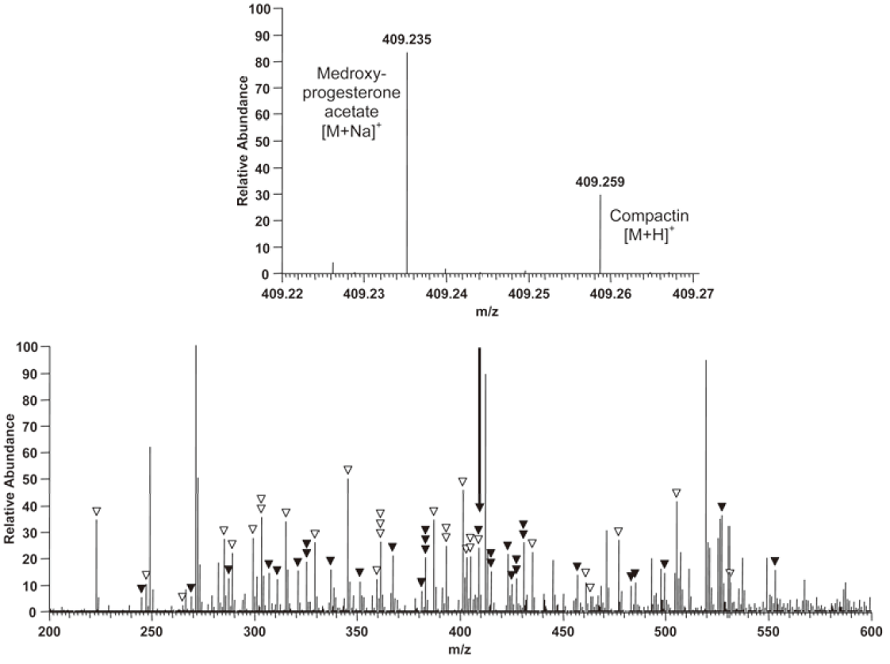
![]()
そこで、各P450酵素を化合物混合溶液と反応させ、基質探索を実施した。P450酵素としては組換え大腸菌の無細胞抽出液を用い、必要に応じて電子伝達系タンパク質コンポーネントを相補させた。反応後、酢酸エチルで抽出し、メタノール-ギ酸溶液に置換して分析サンプルとした。基質の同定は、 (i) 酸化生成物の精密質量数に相当するピークの検出、 (ii) 18O同位体標識による酸化生成物のピークシフトの検出、 (iii) 候補化合物に対する単一化合物での確認、の3段階を経て入念に行った。データ解析はソフトウェア (ジナリス社製) を用いて行った (詳細は省略)。
まず、コンパクチンを基質とすることが報告されているB. subtilis由来CYP109B1を用いて、本手法により化合物混合溶液中のコンパクチンに対する活性を検出可能か検討した。CYP109B1を含む反応液と含まない反応液を比較すると、含む反応液でコンパクチン酸化生成物のナトリウム付加イオンの精密質量数に相当するピークが検出された (図3)。さらに、当該活性を確認する方法として酸素同位体18O2下で反応させたところ、18Oと16Oの精密質量数差分だけピークがシフトした (図3)。これにより、本手法がモノオキシゲナーゼの基質探索に有効なことを確認した。そこで、B. subtilis由来の8種類のP450に対して基質探索を実施したところ、CYP109B1はコンパクチンのみならずいくつかのトリテルペンに対しても活性が検出され、これまでに活性が報告されていないCYP107J1、CYP134A1に対してもそれぞれ1化合物、1化合物を基質として見出すことに成功した (図4)。同様に、B. cereus由来の3種類のP450に対しても基質が見出された (図4)。
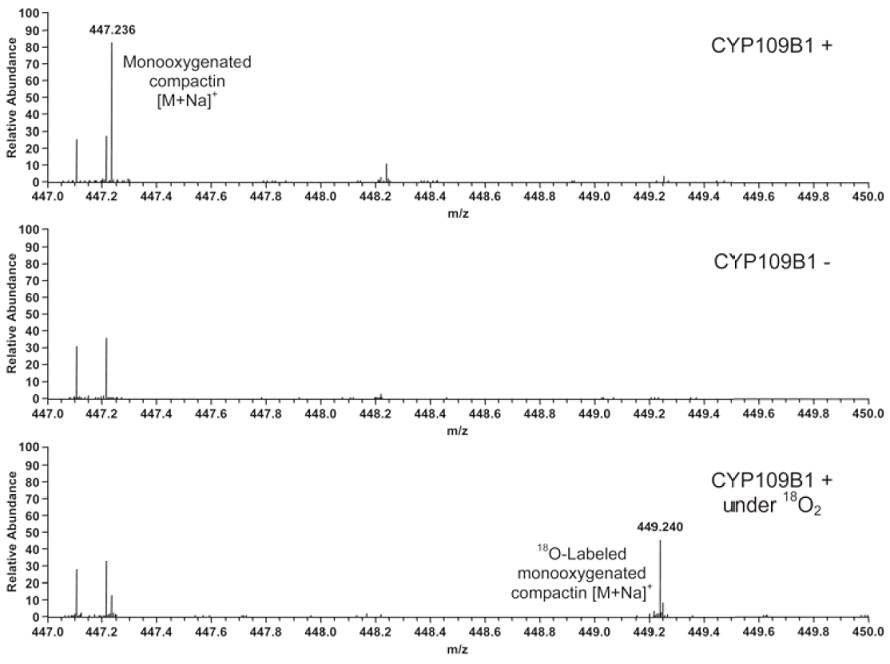
図3 CYP109B1による化合物混合溶液中のコンパクチン酸化のFT-ICR/MS分析
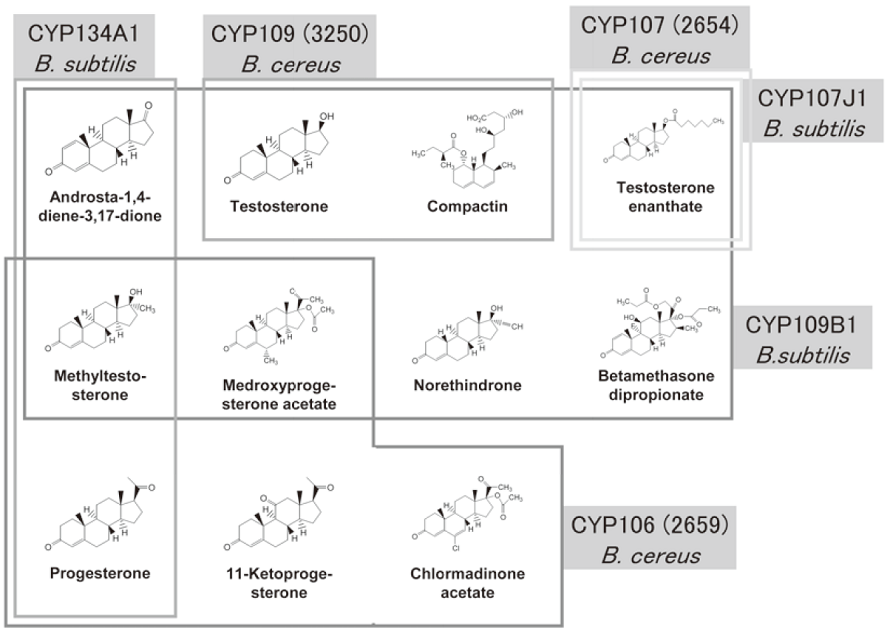
図4 FT-ICR/MSを利用した探索法により見出されたBacillus由来P450の基質
4.おわりに
FT-ICR/MSを利用した酵素の基質探索法は非常にシンプルな方法であり、本稿ではP450モノオキシゲナーゼの基質探索に有効なことを示した。本手法はサンプル導入からデータ解析終了まで1サンプル当たり10分も要さず、多数の化合物に対する活性の有無を短時間かつ高い同定能で評価可能な点を特長とする。一方、構造異性体を区別することはできないので、一次評価、つまり“基質探索”として有効であり、詳細な構造解析はMS/MS分析、NMR分析等で行うことになる。
また、Bacillus由来の6種類のP450に対してトリテルペン類を基質として同定したが、これらのP450は配列系統樹上でいくつかのファミリーに偏って存在していることを確認しており、本手法は配列情報に基質情報を付与するツールとして有効なことが示唆された。化合物ライブラリーの種類を増やすことにより酵素遺伝子に基質情報のアノテーションを付与したデータベースを構築することも可能であり、データを蓄積することで配列と基質の間に相関関係を見出せるかもしれない。一方、本手法はマスピークのシフトを巧みに利用したものであるため、質量変化を伴う反応、現象に広く応用可能である。FT-ICR/MSの装置自体は高価なこともありまださほど普及していないが、質量分析装置の分解能、質量精度は向上の一途を辿っており、本手法の概念が様々な分野で応用されることを期待したい。
謝辞
本研究は㈶かずさDNA研究所の柴田大輔博士らとの共同研究であり、お世話になった方々に深く感謝いたします。
文献
1) Furuya, T., Nishi, T., Shibata, D., Suzuki, H., Ohta, D., Kino, K.: Chem. Biol., 15, 6 (2008).
2) Marshall, A. G., Hendrickson, C. L., Jackson, G. S.: Mass Spectrom. Rev., 17, 1 (1998).
3) Qian, W. J., Camp, D. G. 2nd, Smith, R. D.: Expert Rev. Proteomics, 1, 87 (2004).
4) Oikawa, A., Nakamura, Y., Ogura, T., Kimura, A., Suzuki, H., Sakurai, N., Shinbo, Y., Shibata, D., Kanaya, S., Ohta, D.: Plant. Physiol., 142, 98 (2006).
5) Chefson, A., Auclair, K.: Mol. Biosyst., 2, 462 (2006).