【トピックス】
GroEL/ES シャペロンによる酵素進化の促進
徳力伸彦
ワイツマン研究所
1.酵素進化における安定性の重要性
タンパク質は進化によって多様な機能や構造を獲得してきた。進化のメカニズム (遺伝子の多様化と環境による選択) を模倣し、タンパク質を実験室内で人工的に進化させる進化工学は、酵素の安定化や機能の向上、新規抗体の生産などを成功させ、産業界や医学界でも広く応用されており一定の成果を収めてきたと言える1-4)。しかし、全く新しい反応を行う酵素や、天然の酵素のように非常に高い触媒能を持つ酵素を生産することはほとんど成功していない。多くの酵素の進化工学を行った実験では、数倍から数百倍程度の新規機能の向上の後、それ以上進化しないという現象が観察されている。これは、我々の進化のダイナミクスに対する知見が未だに成熟していないためであると考えられる。
このような背景の中、10年ほど前からタンパク質の進化のダイナミクスをさらに理解しようという研究が盛んに行われてきている5-8)。その中での最も重要な知見に「タンパク質は安定性の低下が進化のボトルネックになっている」がある5-8)。タンパク質の天然構造は、天然構造と変性構造のわずかな安定性の差によって保たれている。多くの研究で示されているように、わずか1アミノ酸変異によって、タンパク質の安定性が低下し凝集や分解が誘導され、結果的にタンパク質を不活性化させる。また、アミノ酸変異のほとんどはタンパク質の安定性を低下することがわかってきた9)。つまり、タンパク質はアミノ酸変異による安定性の低下に常にさらされながらも、凝集や分解などによる不活性化を回避しながら進化しなければならないのである。このことは、酵素の機能進化を考える際にも非常に重要である5,10)。実際、酵素機能の向上に寄与するアミノ酸変異の多くがタンパク質の安定性を低下させることが我々の研究によって明らかにされた11)。酵素が生体内で不活性化してしまえば、あるアミノ酸変異が酵素の機能向上に寄与しても、その変異体は進化の過程で選択されてくることはない。すなわち、あるアミノ酸変異のタンパク質の安定化および不安定化 (凝集や分解) に対する影響が、タンパク質の進化速度を規定しているといえる。タンパク質の進化が円滑に進むためには、安定性の低下による凝集や分解を効率的に回避するメカニズムが重要なキーポイントとなる。例えば、タンパク質を安定化する変異が、機能を向上させる変異の安定性の低下を補償することで、進化において非常に重要な役割を果たしてきていることが近年の研究であきらかにされてきた5,10-12)。本稿では、安定性の低下を緩衝するメカニズムの一つとして、シャペロンが酵素の進化速度を速めることを実験的に示した我々の研究を紹介する13)。
2.シャペロンの生体進化における重要性
シャペロンは生体内の他のタンパク質のフォールディングを補助するタンパク質として知られている。特に熱などのストレスによって生体内で発現量が増加することからHeat shock protein (HSP) とも呼ばれている。従来、熱などの環境変化によるタンパク質のミスフォールディングに対応するために、シャペロンが進化してきたと考えられてきた。一方で、シャペロンが他のタンパク質の進化に重要な役割を果す可能性についても議論されてきた9)。1989年にLaRossaのグループは温度感受性の高い遺伝子を持つ大腸菌の変異体が、GroEL/ES大量発現下では熱感受性が抑制されることを報告した17)。さらに、1998年にRutherfordとLindquistは、ショウジョウバエ集団のHSP90の機能を低下させたところ様々な奇形が観察されることを示した18)。このことは、HSP90によりフォールディングを補助されていた多くのタンパク質が、HSP90の機能低下によりミスフォールディングし、機能を発揮できないために発生の過程で様々な奇形として現れたと考えられている。これらの研究が試金石となり、シャペロンは変異により不安定化したタンパク質のフォールディンを助けることで、「遺伝的緩衝剤」または「遺伝的コンデンサ」として働くという概念が提案された14-16)。また、Barrioのグループは、GroEL/ES大量発現下で、大腸菌を変異源物質を含んだ培地で3240世代継代した。すると、継代後の大腸菌はGroEL/ES大量発現がない状態では増殖速度が著しく低下した19)。これは、上述したように、GroEL/ESが突然変異により不安定化した変異体タンパク質のフォールディングを助けることで、大腸菌内のタンパク質は進化してきたため、GroEL/ESの発現量が低下するとそれらの変異体はフォールディングできなくなり、大腸菌の増殖速度の低下をもたらしたのだと考えられている。これらの研究は、シャペロンの進化における重要性を実験的に見事に証明しているが、分子レベルのメカニズム、さらにシャペロンが変異体の不安定化をどの程度緩衝できるかなど明確に示されていなかった。
3. GroEL/ESシャペロンの酵素の中立進化における影響
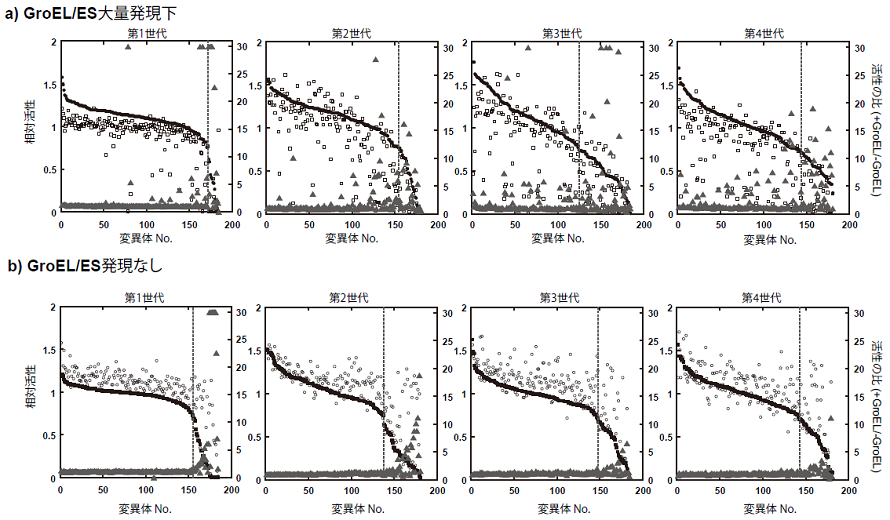
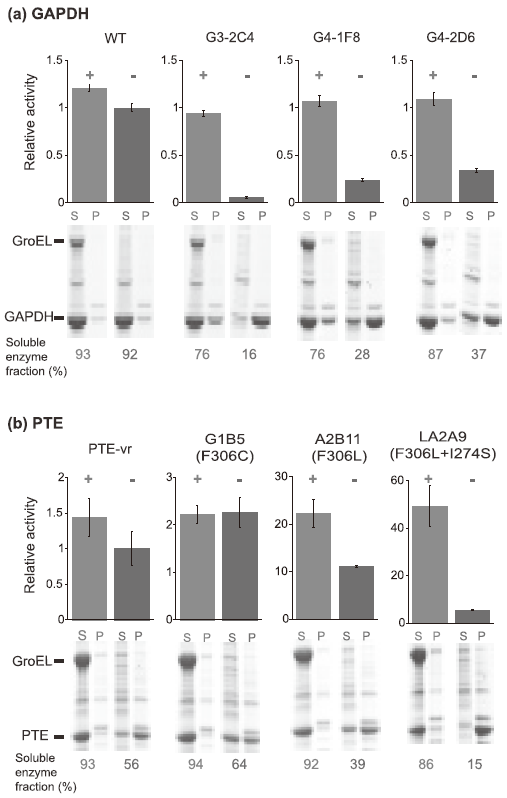
我々は、どの程度シャペロンが変異による安定性低下の影響を緩衝するかを観測するため、大腸菌GroEL/ESの大量発現下で、酵素の実験室内進化実験を行った。roEL/ESの発現をアラビノースプロモーターで制御し、酵素の発現をT7プロモーターで制御することで、GroEL/ESと酵素の共発現系を確立した。単純な実験のモデル図を図1に示す。まず、酵素の中立進化実験 (変異と浄化選択の繰り返し:平均1アミノ酸変異/遺伝子) を行い、どの程度GroEL/ESが不安定化する変異を緩衝できるのかを観察した。GAPDH (Glyceraldehyde dehydrogenase) における中立進化の結果を図2に示した。各世代190変異体をランダムに抽出し、GroEL/ES大量発現下 (●)、もしくは非発現下 (□) での大腸菌ライセートにおける酵素活性量を測定した。それらの活性の比 (▲:+GroEL/-GroEL) を求め、それをGroEL依存度の指標とした。浄化選択は、天然型の70%以上の活性を持つ変異体 (点線より左の変異体) を回収することにより行った。回収された変異体ライブラリーにさらに変異を加え、浄化選択を4世代ほど繰り返した。GroEL/ES大量発現下では、数世代でGroEL依存発現する変異体 (▲の値の高い点) が増殖した (図2上)。一方で、GroEL/ES非存在下ではそのような変異体は蓄積されなかった (図2下)。いくつかの変異体の可溶性発現をSDS-PAGEによって調べたところ、GroEL/ES非発現下における触媒活性の大きな減少は酵素の凝集化によるものであることが確認された (図3)。3、4世代目で得られた変異体のアミノ酸配列を解析すると、GroEL依存性となった変異体 (+GroEL/-GroEL>2は、GroEL非依存性の変異体と比較して、2倍以上 (3.5 vs1.6 アミノ酸変異/遺伝子) のアミノ酸変異が蓄積していることがわかった。さらに、GroEL非依存性の変異体ではほとんどの変異がタンパク質表面に分布していたにもかかわらず、GroEL依存性の変異体では、多くのアミノ酸変異はタンパク質のコアにも広く分布していた。計算機プログラム (FoldX) でそれらの変異による熱力学的安定性への影響 (ΔG) を解析すると GroEL 依存性変異体では遺伝子あたり3.5 kcal/mol安定性が低下していたのに対し、GroEL非依存性の変異体ではの進化では1 kcal/molほどしか低下していなかった。つまり、GroEL/ESは、タンパク質の安定性の低下を2.5 kcal/mol程度緩衝することで、タンパク質に蓄積する変異の数、種類に大きな影響を及ぼしたことが、我々の実験室内進化実験により示された。他の2つの異なる酵素、PTE (phosphotriesterase)、CAII (carbonic anhydorase) を用いて同様に中立進化実験を行ったところ、ほぼ同じ結果が得られた。これらのことから、GroEL/ESの大量発現は、アミノ酸変異による安定性の低下によって引き起こされる酵素の凝集による不活性化を防ぎ、進化の過程でより多くの変異の蓄積を可能とすることで、酵素の進化速度を促進していることが示された。
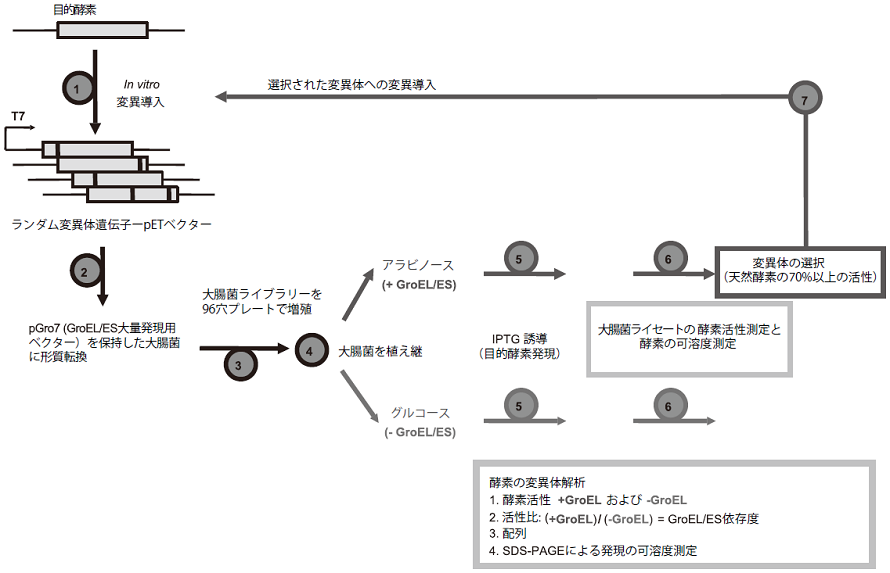
図1 進化実験の概要


4. GroEL/ESシャペロンが酵素の機能進化を補助する
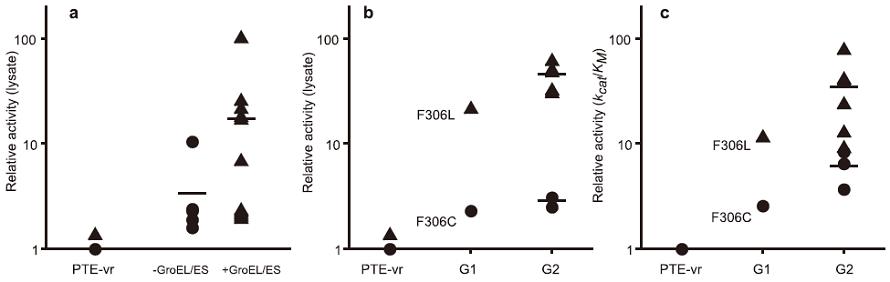
GroEL/ESは酵素の機能進化の側面においても、進化速度を促進する役割を果たすのであろうか。この疑問に対し、我々は、フォスフォトリエステラーゼ (paraoxon) であるPTEを、エステラーゼ (2-naphtylhexanoate) に進化させるモデル実験を、GroEL/ESの大量発現と非発現下の両条件下で行った。すると、GroEL/ES大量発現下では、機能が向上した変異体が非発現下と比較して2倍程度得られた。また、それらの触媒活性は10倍以上向上していることが確認された (図4)。得られた変異体の安定性を調べてみると、GroEL/ES大量発現下で得られた変異体の安定性は軒並み低下しており、GroEL/ES非存在下で変異体を発現させてみると、それらの変異体は凝集する傾向にあることがわかった。これらのことは、GroEL/ESが機能を向上させるが安定性を低下させるアミノ酸変異によるPTE変異体の凝集を防ぎ、機能向上を担保していることを示している。

5. GroEL/ESの緩衝能の限界と、安定性を向上するアミノ酸変異
上記の実験室内中立進化実験および機能進化実験は、GroEL/ESシャペロンがアミノ酸変異による安定性の低下を緩衝し、タンパク質進化において大きな影響を与えることを示している。しかし、GroEL/ESの緩衝能には明らかに限界がある。中立進化実験ではGroEL/ESによって緩衝された安定性の低下は2~3 kcal/mol程度であり、それ以上の安定性の低下はGroEL/ES大量発現下であろうと酵素の凝集を引き起こす。そこで、重要な役割を果たすのがタンパク質の安定性を向上させる別のアミノ酸変異である。実際、GroEL/ES大量発現下で選択されたPTEの変異体 (Phe306Leu) を、GroEL/ESなしの条件下でさらにエステラーゼに対する進化実験を行ったところ、安定化に寄与するアミノ酸変異 (Ile274Ser) が選択されてきた。その変異体酵素を精製し触媒活性を調べたところ、Ile274Serはエステラーゼ活性を向上させておらず、安定性を向上させるアミノ酸変異であることが確認された。つまり、GroEL/ESは安定性の低下を緩衝し数アミノ酸変異の蓄積を補助することで、酵素の進化速度を一時的に向上させるが、長い酵素進化においては、安定性を向上させるアミノ酸変異の蓄積が必要不可欠である。
6.まとめ
タンパク質は安定性を保ちながら、凝集、分解などの不活性化を回避しながら進化しなければならない。近年の研究から、タンパク質の安定性に対する進化のプレッシャーが非常に大きいことがわかってきた。アミノ酸変異による安定性の低下、不活性化を回避、緩衝するメカニズムはタンパク質進化を理解するために重要な知見となる。我々の研究は、シャペロンは、変異による安定性の低下を緩衝し、タンパク質の進化能を向上させるメカニズムとして働くことを明確に示した13)。このメカニズムは天然進化においても重要な役割を果たしてきたと予想される。シャペロンの大量発現は、元来熱ストレスによって誘導されるとされたが、現在では殆ど全てのストレスによって誘導されることがわかっている。また同様に様々なストレスによってゲノムの変異率の上昇も誘導されることがわかってきている。生物はストレスに面した時、変異率を上げるだけでなく、シャペロンを大量発現することで、変異導入されたタンパク質の安定性の低下を緩衝し、多様な機能性タンパク質の発現を促進するといった、ストレスに対応した進化を遂げるメカニズムを作ってきたのかもしれない。
また、シャペロンによる安定性の低下の緩衝および安定性を向上させるアミノ酸変異は、新規タンパク質を生産する進化工学においても非常に有用である。これまでの新規機能の向上を目指した進化工学実験の多くは、目的の機能レベルに達することなく頭打ちになってしまっていた。そのような状況の変異体は非常に不安定で凝集しやすくなっているようである。シャペロンを用いたり、安定性を向上させるアミノ酸変異を加えることで、さらに高い機能に実験室内で進化できるようになると考えられる20-23)。事実、我々の実験では、明らかにシャペロン非存在下では蓄積されなかった変異が、シャペロン大量発現下では蓄積された。タンパク質の機能を進化工学、計算機デザインまたはその組み合わせにより研究を進めている方々は、ぜひここで提案した方法を取り入れていただきたい。
文献
1) Turner, N. J.: Trends Biotechnol., 21, 474 (2003).
2) Griffiths, A. D., Tawfik, D. S.: Curr. Opin. Biotechnol., 11, 338 (2000).
3) Tao, H., Cornish, V. W.: Curr. Opin. Chem. Biol., 6, 858 (2002).
4) Jaeger, K. E., Eggert, T.: Curr. Opin. Biotechnol., 15, 305 (2004).
5) DePristo, M. A., Weinreich, D. M., Hartl, D. L.: Nat. Rev. Genet., 6, 678 (2005).
6) Pal, C., Papp, B., Lercher, M. J.: Nat. Rev. Genet., 7, 337 (2006).
7) Camps, M., Herman, A., Loh, E., Loeb, L. A.: Crit. Rev. Biochem. Mol. Biol., 42, 313 (2007).
8) Tokuriki, N., Tawfik, D. S.: Science, 324, 203 (2009).
9) Tokuriki, N., Stricher, F., Schymkowitz, J., Serrano, L., Tawfik, D. S.: J. Mol. Biol., 369, 1318 (2007).
10) Wang, X., Minasov, G., Shoichet, B. K.: J. Mol. Biol., 320, 85 (2002).
11) Tokuriki, N., Stricher, F., Serrano, L., Tawfik, D. S.: PLoS Comput. Biol., 4, e1000002 (2008).
12) Andersson, D. I.: Curr. Opin. Microbiol., 9, 461 (2006).
13) Tokuriki, N., Tawfik, D. S.: Nature, 459, 668 (2009).
14) Rutherford, S. L.: Nat. Rev. Genet., 4, 263 (2003).
15) Fares, M. A., Moya, A., Barrio, E.: Trends Genet, 20, 413 (2004).
16) Rutherford, S., Hirate, Y., Swalla, B. J.: Crit. Rev. Biochem. Mol. Bio., 42, 355 (2007).
17) Van Dyk, T. K., Gatenby, A. A., LaRossa, R. A.: Nature, 342, 451 (1989).
18) Rutherford, S. L., Lindquist, S.: Nature, 396, 336 (1998).
19) Fares, M. A., Ruiz-González, M. X., Moya, A., Elena, S. F., Barrio, E.: Nature, 417, 398 (2002).
20) Bershtein, S., Goldin, K., Tawfik, D. S.: J. Mol. Bio., 379, 1029 (2008).
21) Bloom, J. D., Labthavikul, S. T., Otey, C. R., Arnold, F. H.: Proc. Natl. Acad. Sci. USA, 103, 5869 (2006).
22) Bloom, J. D., Meyer, M. M., Meinhol, P., Otey, C. R., MacMillan, D., Arnold, F. H.: Curr. Opin. Struct. Biol., 15, 447 (2005).
23) Bershtein, S., Tawfik, D. S.: Curr. Opin. Chem. Biol., 12, 151 (2008).