【トピックス】
アリールマロン酸脱炭酸酵素の機能改変
宮本憲二
慶大・理工
1.はじめに
近年、進化工学的手法による酵素の機能改変が盛んに行われている。様々な手法が開発されているが、「Rational Design」と「Random Mutation」に大別することができる。前者は、構造情報を参考とした合理的なデザインをもとに改変を行う。特定の部位に変異を導入するのでスクリーニングする変異体は少なくてよいが、詳細な構造情報が不可欠である。後者は、遺伝子に対してランダムに変異を導入し、その中から目的の性質を持つ変異酵素を探す方法である。したがって、構造情報は必要ないがライブラリーサイズは大きくなるので、効率の良いスクリーニング系が必須となる。
脱炭酸酵素は、カルボン酸から二酸化炭素を脱離する反応を触媒し、[EC 4. 1. 1. -]に分類される。脱炭酸酵素の中には逆反応であるカルボキシル化を触媒するものも存在し、様々な芳香族ヒドロキシカルボン酸の合成に適用されている1) (図1–式1)。一方、正反応である脱炭酸は、物質生産に利用されることはあまりなかった。本稿で取り上げる脱炭酸酵素は我々が独自に見出し、補酵素なしで直接マロン酸の脱炭酸を行うユニークな酵素である2)。本酵素は、非天然型基質であるアリールマロン酸に作用し、光学的に純粋なアリール酢酸誘導体へと変換する活性を持つため、物質生産の観点からも有用性が高い (図1-式2)。著者らは、本酵素を自然界から見いだし、その酵素学的諸性質や反応メカニズムを明らかにした。更に、配列や構造情報に基づいて最小限の変異を導入することで、本来酵素が持っていない様々な機能を付与することに成功した。また最近、X線結晶構造解析にも成功し、その興味深い反応メカニズムの全体像が見えて来たので紹介したい。
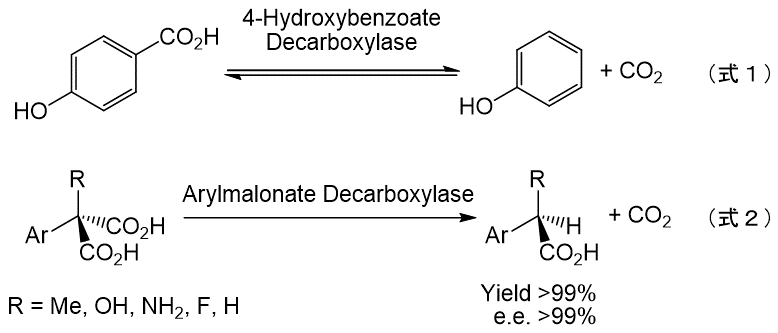
図1 脱炭酸酵素の例
2.アリールマロン酸脱炭酸酵素
全国各地の土壌から、フェニルマロン酸を単一炭素源として集積培養を行い、アリールマロン酸に対する脱炭酸酵素活性を持つ細菌Alcaligenes bronchisepticusを分離した3)。培養条件の最適化を行ったところ、本酵素はフェニルマロン酸によってのみ誘導された。本酵素を各種クロマトグラフィーにより均一に精製したところ、分子量24 kDa (SDS-PAGE) のモノマー酵素であった。活性の発現には既知の脱炭酸酵素に必要な補酵素を要求しない、全く新しいタイプの酵素であった4)。基質特異性を調べたところ、アリール基が直接結合したマロン酸には作用するが、アルキルマロン酸には活性を示さなかった。また、二置換マロン酸にも作用して、高い光学純度のアリール酢酸誘導体を与えた。また、二置換より一置換のアリールマロン酸の方が高い反応性を示したことから、アリールマロン酸脱炭酸酵素 (以下AMDase) と命名した。現在では、[EC 4. 1. 1. 76]に分類されている。また、この株以外からも性質の異なるAMDaseの取得に成功5,6)しており、ライブラリーを構築している。
A. bronchisepticusのゲノムDNAからショットガンクローニング法により、AMDaseと推定される遺伝子を取得した。720 bpのORFがコードするタンパク質の分子量は24,737 Daであり、精製したAMDaseのSDS-PAGEの結果 (24 kDa) とよく一致した。また、ORFより推定されるタンパク質のN末端配列は、精製酵素から決定した配列と完全に一致した7)。以上の結果から、本ORFがAMDaseをコードしていると判断した。また、大腸菌において全タンパク質の30%以上にも達する過剰発現にも成功し、野生株では発現量の少なかった酵素を大量に得ることが可能となった。
阻害実験からシステインが活性の発現に重要な残基であることが分かっていた。そこで、4つ存在するシステインをそれぞれアラニンに置換した変異体を作成し活性を測定したところ、188番目が活性システイン残基であることがわかった8)。更に、反応の詳細なメカニズムを明らかにするために、13Cでラベルした光学活性な基質を合成し反応を試みた。その結果、脱炭酸反応は完全に立体反転により進行していた9)(図2)。メチルマロニル-CoA脱炭酸酵素などの既知酵素では、反応は全て立体保持で進行することが知られており、本酵素は非常に特異な反応メカニズムであることが推定された。立体反転が起こるためには、一旦プロキラルなエノール型の中間体が生成し、これに対してエナンチオ面選択的なプロトン付加が起こっていることが予想された。
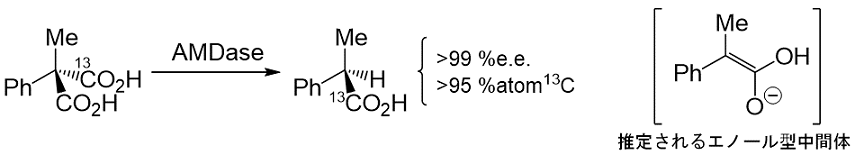
図2 脱炭酸反応のメカニズム
3 .脱炭酸酵素によるアルドール反応 (Catalytic Promiscuity)
ここまでの検討から脱炭酸反応の過程でエノール型中間体の存在が強く示唆された。そこで、エノール中間体の近傍に求電子剤であるアルデヒドが存在すると、アルドール反応が進行するのではないかと考えた。そこで、反応系にアセトアルデヒド等のアクセプター分子を共存させてみたが、アルドール生成物を確認することができなかった。そこで、中間体の近傍にアルデヒド基が確実に位置するように、フェニルマロン酸の分子内にアルデヒド基を導入した基質をデザインした (図3)。そして、実際に基質を合成し反応を試みた結果、収率は35%であったが期待するアルドール生成物が得られ、エノール中間体を求電子剤でトラップすることに成功した10)。エノール中間体の寿命は短いと考えられるので、アルドール反応は酵素の活性部位内で起こっていると考えた。したがって、酵素の活性中心というキラルな空間内での反応なので、何らかの選択性を伴って進行しているものと期待した。しかし、アルドール生成物を分析したところ残念ながらラセミ体であった。
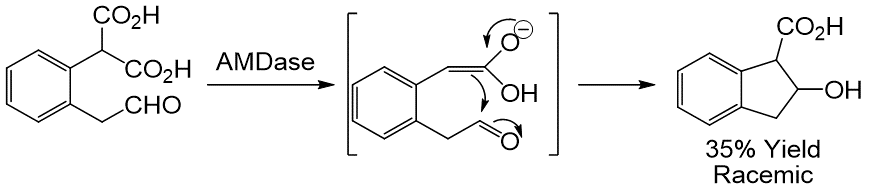
図3 脱炭酸酵素によるアルドール反応
1種類の酵素が、ひとつの活性部位で複数の異なる化学反応を触媒する能力のことをCatalytic Promiscuityという。10年ほど前から使われ始めた言葉であり、辞書を引くとPromiscuityは混乱といった意味である。筆者は、2010年7月に米国Bryant大学で行われたGordon Research Conference (Biocatalysis) に招待講演者として参加した。この会議では、Catalytic Promiscuityという言葉が何度も登場し (筆者も発表で使ったが)、 この分野で広く認知されていることを実感した。先に述べたAMDaseによるアルドール反応は、本酵素のCatalytic Promiscuityを示すことができたよい例と言える。しかし、その酵素が本来持っていない活性を変異等で付与した場合は、Catalytic Promiscuityではない。したがって、次に述べるAMDase変異体による立体選択性の逆転やラセマーゼへの改変は、Catalytic Promiscuityではない。
4.立体選択性の逆転
AMDaseの配列を用いてホモロジー検索を実施したところ、アミノ酸ラセマーゼや各種イソメラーゼと低いながらも相同性を示した (図4)。アミノ酸ラセマーゼに関しては詳細な反応機構が解明されており、2つのシステインが共同してプロトンの引き抜きと供与を行う二塩基機構が提唱されている。AMDaseとラセマーゼの配列を比較すると、AMDaseの活性中心にはシステインがひと一つしか存在しないことがわかる。そこで、エノール中間体に対して一方の面からのみシステインがプロトン供与を行い、光学活性なカルボン酸が生成するのではないかと推定した。
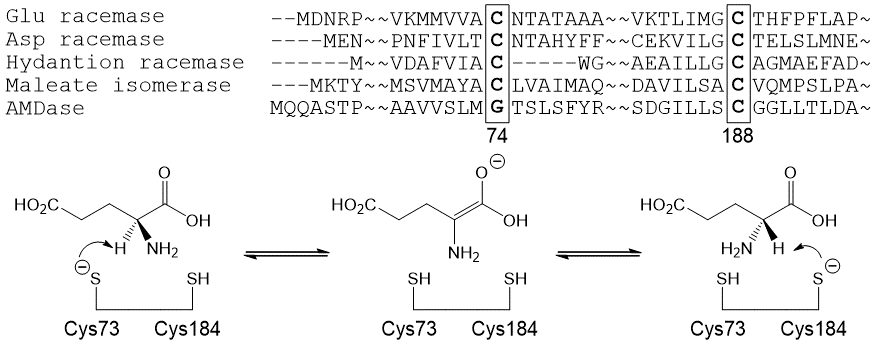
図4 相同性とアスパラギン酸ラセマーゼの反応機構
酵素の立体選択性を逆転することは、酵素の機能改変の中で最も困難な課題のひとつである。AMDaseの立体選択性を逆転する場合、2つの方法が考えられる。1つ目は、基質のフェニル基とメチル基が入る結合部位に変異を導入し、ポケットへの入り方を反転することである (図5-方法A)。しかし、この方法ではポケットの大規模な改変が必要であり難易度は非常に高い。2つ目は、プロトン供与を行うシステインの位置をエノール中間体の逆側に移動することである (図5-方法B)。この場合、188番目のシステインをプロトン供与能の低いものに置換し、反対側にシステインを導入するだけで比較的単純である。問題は反対側の位置の特定であるが、アミノ酸ラセマーゼとのアライメント結果から74番目のグリシンではないかと推定した (図4)。そこで、デザインした二重変異体 (G74C/C188S) を作成し反応を試みたところ、期待通り野生型AMDaseと全く逆の選択性を示した11)(図6)。目論見通り立体選択性の逆転に成功したが、その活性は野生株の約1/1000に低下していた。
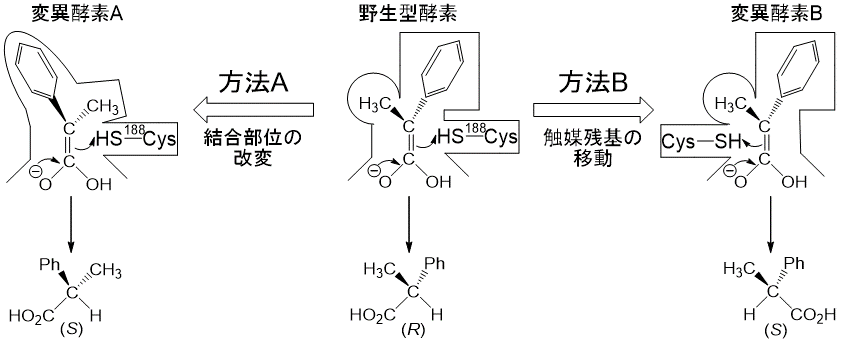
図5 Rational Designによる立体選択性の逆転
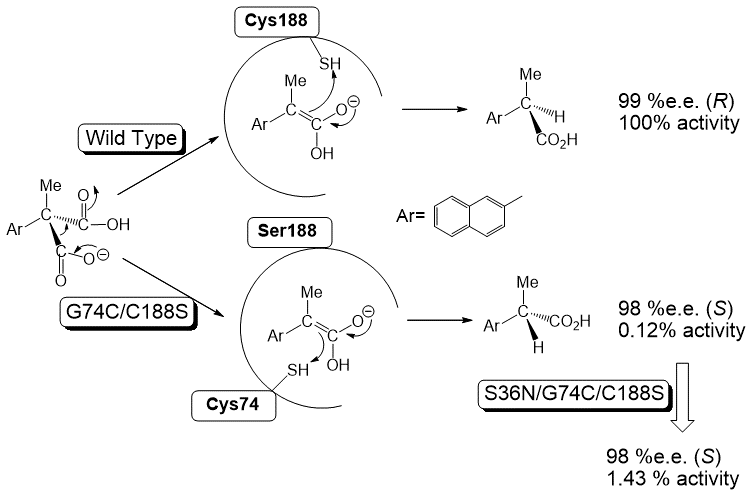
図6 立体選択性の逆転と活性の向上
酵素に変異を導入するとしばしば酵素活性の低下という深刻な問題に直面する。配列や構造からデザインして酵素活性を向上できればよいが、現時点では非常に困難である。そこで、Random Mutationによる活性の向上を試みた。変異導入には、修復系の遺伝子が欠損したMutator Strainを用いた。上記二重変異体AMDase遺伝子を含むプラスミドでStratagene社製E. coli XL1-Redを形質転換し、数回植え継ぐことでランダムに変異を導入した。得られた変異を含むプラスミドを用いて発現用の大腸菌を形質転換した。そして、脱炭酸酸活性を色の変化で検出することのできるプレートを用いて約5万株のスクリーニングを行ったところ、1株が野生株より強い酵素活性を示した。配列を解析したところ、二重変異体 (G74C/C188S) に対して新たな変異G36Nが導入されており、活性は約10倍向上していた12)(図6)。現在、後に述べる立体構造を参照にして、更なる活性の向上を試みている。
5.ラセマーゼへの改変
先に述べたようにAMDaseは、アミノ酸ラセマーゼと相同性を有していた。そこで、AMDaseの活性中心に2つ目のシステインを導入すれば、ラセマーゼに変えることができるのではないかと考えた (図7)。α-アリールプロピオン酸に作用するラセマーゼは全く報告がなく、pKa 20程度のα-アリールプロピオン酸のα位のプロトンを、pKa 8程度のシステインのチオールのアニオンが補酵素の助けも借りずに引き抜けるかどうか大変興味が持たれた。そこで、G74C変異体を作成し光学活性なα-アリールプロピオン酸を作用させたところ、野生型にはないラセマーゼ活性を確認した (図7)。また、脱炭酸活性は野生型の2.2%に低下していた。基質特異性を確認したところ、脱炭酸反応においてよい基質は、ラセミ化反応でもよい基質であった13)(図8)。この事実は、脱炭酸とラセミ化が同じ活性中心で起こっていることを示している。また、アミド、ニトリルやエステルには活性を示さず、カルボキシル基がバインディングに必須であることがわかった。エチル基を有する基質は、脱炭酸反応は進行しないがラセミ化が進行した唯一の化合物であった (Run 4)。現在、基質と酵素のドッキングシミュレーションの結果を用いて、G74Cラセマーゼの機能改変を進めている。
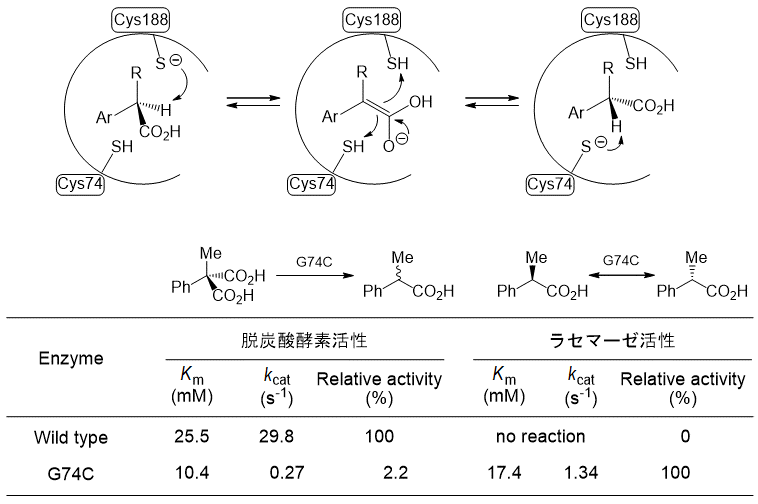
図7 G74C変異体の脱炭酸活性とラセマーゼ活性
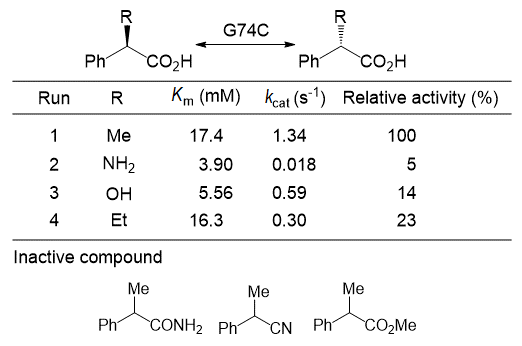
図8 ラセマーゼG74Cの基質特異性
6.構造解析と反応機構
最近、我々のグループ14,15)と他のグループ16,18)は独立にAMDaseのX線結晶構造解析に成功した。我々は、野生型AMDaseだけではなく変異型についてもリガンドが結合した構造の解析に成功した。その結果、野生型と変異型の活性部位は、ほとんど変化がないことが分かった。また、リガンドが結合することにより活性部位の上に疎水性部位が生成し、基質が活性部位に結合することにより、開いた構造から閉じた構造に変化することが推定できた。そして、代表的な基質であるα-メチル-α-フェニルマロン酸を用いて結合モデルを作成し、反応機構を考察した。その結果、G74, L77, Y48の各残基によって構成される小さな疎水性ポケットによって一方のカルボキシル基が不安定化されて、それがトリガーとなり脱炭酸反応が始まる。そして、生成したエノール型中間体に対して188番目 (G74C/C188S変異体では74番目) のシステインがプロトン供与を行い、光学活性カルボン酸が生成するという反応メカニズムを推定した。
Micklefieldらのグループは、アリール基ではなくアルケニル基を持つ基質についても、AMDaseが作用し同様の脱炭酸反応が進行することを報告している16)(図9)。反応の立体選択性は、アリール基の場合と同じであった。また、反応性については、アルケニル型基質の方がKm値は良いが、反応速度はアリール型基質の方が高い値を示した。アルケニル型基質の発見により、AMDaseの汎用性は更に広がったといえる。
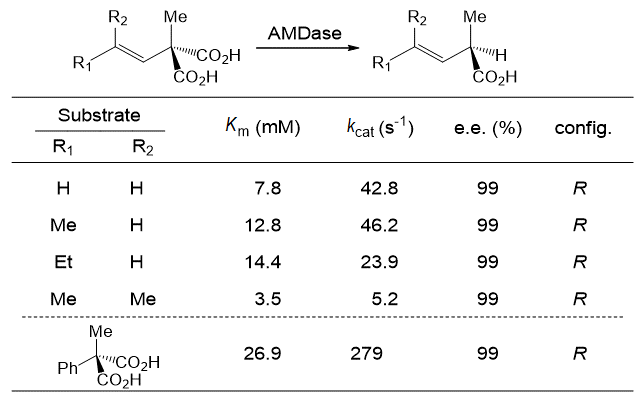
図9 アルケニルマロン酸の脱炭酸反応
また、同グループは相同性検索の結果から、新たなAMDase候補遺伝子を見いだしている (図10)。そして、我々の見いだした酵素の活性 (Run 1) を超えるものはないが、アミノ酸配列の大きく異なったAMDaseを見いだすことにも成功している17)。しかし、A. pyrophilus由来のMurI (Run 6) は、高いGlutamate racemase活性 (Km=0.21 mM, kcat=3.3 s-1) を有することから、脱炭酸酵素と言うよりはラセマーゼが本来の機能であると考えられる。
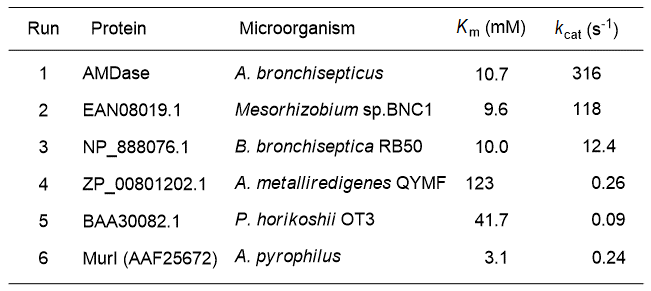
図10 AMDaseホモログと脱炭酸活性
7.おわりに
我々が見いだしたAMDaseの反応機構とその有用性を明らかにした。そして、最小限の変異を導入することにより様々な機能改変を行うことに成功した。S体選択的なAMDaseやα-アリールプロピオン酸に作用するラセマーゼは、天然には存在しないものである。これらの知見は、酵素機能を積極的に改変していく上で重要な情報を提供できるものと考えている。今後は、明らかとなった立体構造をもとに、酵素活性の更なる向上や新たな基質への適用を行う予定である。
本稿では、酵素機能を積極的に改変する研究について紹介してきた。しかし、酵素はいくらでも自分の好きなように改変できるわけではなく、自ずと限界がある。したがって、改良するにしても、元となる酵素の素性がよくないと、改変された酵素も実用性の高いものにならない。そのためにも、新酵素のスクリーニングは必ず推進していかなければならない重要な研究課題であると考えている。以上のような信念のもと、筆者の研究室では、従来の泥臭いスクリーニングから、最近著者が申請代表者として慶應義塾大学理工学部に導入した次世代シークエンサー (Illumina社製 Genome Analyzer IIx) を用いた大規模配列解析による酵素遺伝子探索まで、様々な新酵素のスクリーニングを精力的に実施している。ここで触れなかった他の酵素についても、またの機会にご紹介できればと思っている。
謝辞
本稿で紹介した研究は、慶應義塾大学理工学部で行われたものであり、太田博道 慶應義塾大学名誉教授をはじめとする共同研究者の皆様に感謝致します。なお、研究の一部は文部科学省科研費補助金基盤研究 (B) と平成20年度福澤諭吉記念慶應義塾学事振興基金の助成のもとで行われたものであり、ここに感謝致します。
文献
1) Matsui, T., Yoshida, T., Hayashi, T., Nagasawa, T.: Arch. Microbiol., 186(1), 21 (2006).
2) Miyamoto, K., Ohta, H.: J. Am. Chem. Soc., 112, 4077 (1990).
3) Miyamoto, K., Ohta, H.: Biocatalysis, 5, 49 (1991).
4) Miyamoto, K., Ohta, H.: Eur. J. Biochem., 210, 475 (1992).
5) Miyamoto, K., Yatake, Y., Tamura, K., Terao, Y., Ohta, H.: J. Biosci. Bioeng., 104(4), 263 (2007).
6) Yoshito, Y., Miyamoto, K., Ohta, H.: Appl. Microbiol. Biotechnol., 78(5), 793 (2008).
7) Miyamoto, K., Ohta, H.: Appl. Microbiol. Biotechnol., 38, 234 (1992).
8) Miyazaki, M., Kakidani, H., Hanzawa, S., Ohta, H.: Bull. Chem. Soc. Jpn., 70(11), 2765 (1997).
9) Miyamoto, K., Tsuchiya, S., Ohta, H.: J. Am. Chem. Soc., 114, 6256 (1992).
10) Terao, Y., Miyamoto, K., Ohta, H.: Chem. Lett., 36(3), 420 (2007).
11) Terao, Y., Ijima, Y., Miyamoto, K., Ohta, H.: J. Mol. Catal., B Enzym., 45(1-2), 15 (2007).
12) Terao, Y., Miyamoto, K., Ohta, H.: Appl. Microbiol. Biotechnol., 73(3), 647 (2006).
13) Terao, Y., Miyamoto, K., Ohta, H.: Chem. Commun., (34), 3600 (2006).
14) Nakasako, M., Obata, R., Okubo, R., Nakayama, S., Miyamoto, K., Ohta, H.: Acta Crystallogr. Sect. F Struct. Biol. Cryst. Commun., F64(7), 610 (2008).
15) Obata, R., Nakasako, M.: Biochemistry, 49, 1963 (2010).
16) Okrasa, K., Levy, C., Wilding, M., Goodall, M., Baudendistel, N., Hauer, B., Leys, D., Micklefield, J.: Angew. Chem. Int. Ed. Engl., 48, 1 (2009).
17) Okrasa, K., Levy, C., Hauer, B., Baudendistel, N., Leys, D., Micklefield, J.: Chem. Eur. J., 14, 6609 (2008).
18) Kuettner, E. B., Keim, A., Kircher, M., Rosmus, S., Sträter, N.: J. Mol. Biol., 377, 386 (2008).