【トピックス】
アミノ酸置換によるHalomonas nucleoside diphosphate kinase四次元構造変換と「好塩性」性質の付与
徳永廣子、徳永正雄
鹿児島大・農
1.はじめに
極端な環境条件下 (高温、極低温、高圧、高塩など) で生育できる微生物は、通常とは異なった能力を持つ酵素を働かせ、その厳しい環境に適応していると考えられる。その極限微生物の一種である好塩性細菌が産生するnucleoside diphosphate kinase (ヌクレオシド二リン酸キナーゼ) について、タンパク質が示す「好塩性」という性質とその構造面からまとめてみたい。
2.好塩性細菌と好塩性酵素
好塩性細菌は文字どおり塩環境に適応し、
それに対し耐塩性細菌は塩がない環境で生育するが、高濃度の塩存在下でも生育可能な細菌でありそれが作り出す耐塩性酵素についても良く研究されている。しかし、それらの酵素タンパク質の「耐塩性」共通メカニズムが明らかとなっている状況ではない。Bacillus thermoproteolyticusサーモライシン2) は塩による活性増加及び安定化の増加がみられるがアミノ酸組成は以下に述べる好塩性酵素のように負荷電が際立って多いという特徴があるわけではない。もちろん酸性アミノ酸に富む耐塩性酵素も報告されている3)。この「好塩性」と「耐塩性」は重なっている部分があるはずだが、耐塩性酵素と塩の相互作用はそれぞれの耐塩性酵素自身が個別に生み出す特異的なメカニズムによるものと考えられる。
好塩性酵素にもどる。好塩性酵素のアミノ酸組成は酸性アミノ酸が際立って多く、酸性アミノ酸はそのタンパク質表面に局在分布している。その結果、通常のタンパク質と比較して多量の水和水と陽イオンを表面に結合している。この陽イオンにより表面の負電荷どうしの反発が抑えられ、さらに塩析効果もあいまってタンパク質コア構造が安定化されている。
3.ヌクレオシド二リン酸キナーゼの高次構造
ヌクレオシド二リン酸キナーゼ (以下NDKと略す) はヌクレオシド三リン酸γ位リン酸基をヌクレオシド二リン酸へ転移する働きをする酵素である。ヌクレオシド三リン酸の細胞内プールを制御するという重要な働きを持つだけでなく、細胞増殖、分化、Gタンパク質を介した情報伝達、がん転移抑制、DNA修復など広い機能を有した多機能タンパク質として知られている4,5)。また動物、植物、微生物にわたり広く見出され、そのアミノ酸配列一次構造はよく保存されている。
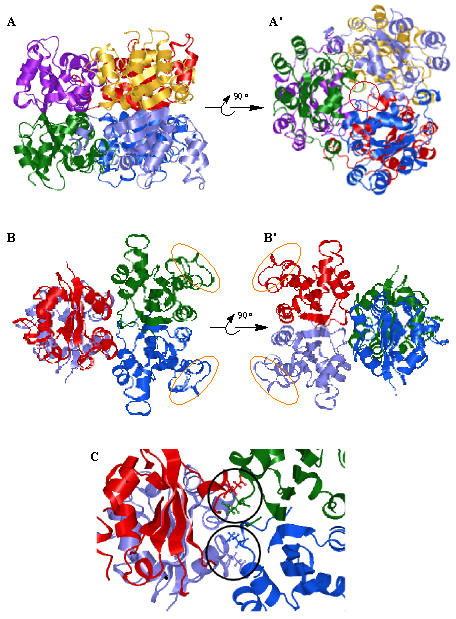
真核生物、古細菌、グラム陽性菌のNDKは6量体構造をとっている。これは2量体を基本とし、その基本構造が3つ集合し6量体を形成している。6量体構造をとるNDKのX線結晶構造解析についてはヒト、Drosophila, Bacillus subtilis, Mycobacterium tuberculosisなど数多く報告されている。筆者らが好塩性タンパク質の研究材料として取り組んできた古細菌・高度好塩性細菌Halobacterium salinarum NDKの構造6) はホモ6量体であり一例として図1に示す。6量体を上から見た3分子鎖からなるリングでは (図1A’)、Kpnループ (killer of prune) と呼ばれる領域が中央に集まり、α-へリックス1 (15-19残基) と隣の分子鎖Trp152* (以下、*記号をつけたものは隣の分子の残基を示す)、Gln98とGln87*、107-11 5残基部分と31*-34*残基部分・C末部152*-155*間に接触が見られる。

一方、E.coli, Myxococcus xanthus, Pseudomonasaeruginosaなどのグラム陰性菌NDKはホモ4量体 (基本構造の2量体が2つ) をとっている。ところが、当研究室において高度好塩性細菌Halobacterium salinarum NDKとともに研究材料としていた中度好塩性細菌Halomanas 593株NDKは基本構造のホモ2量体で存在していることが明らかとなった7)。さらなる多量体構造 (4量体、6量体) を取らずともこの2量体の状態で酵素活性を保持していることが明らかとなり、2量体が活性型最小単位であることが初めて証明された。では何故Halomonas NDKは基本構造の2量体のままで存在しているのであろうか?
Myxococcus NDKの結晶学的構造8) より、その4量体構造における基本構造2量体間の接触面は大きく以下の2つの部分からなる。1つは前半部分β2構造付近 (図2) Arg37-Ser42の部分であり、他の1つはC末端部分Ala12 9-Glu136がそれに相当する。Kpnループは外側に向いている (図1B)。3次元構造が解明されているE.coli NDKも4量体構造をとるが、基本構造2量体2個の組み立て方がMyxococcus NDKと異なっており、基本構造2量体間の接触面にKpnループが存在するのは6量体型に似ている。
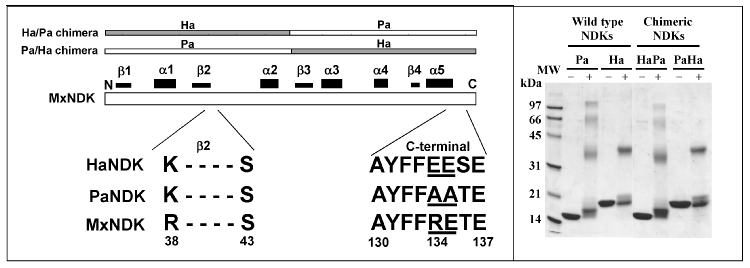

Halomonas NDKが基本構造2量体のままで存在しているのは何故か?これを明らかにするためにHalomonas NDKのホモログである非好塩性細菌Pseudomonas aeruginosa NDK (相同性80%以上) に着目した。Pseudomonas NDKは4量体構造をとるので、この2つを比較することで構造形成に関与するアミノ酸の同定が可能ではないか。この視点のもとに2つのタンパク質のキメラタンパク質を創ることにした。
ちょうど都合のよいことにHalomonas NDK (140アミノ酸、以下HaNDKと略す)、Pseudomonas NDK (142アミノ酸、以下PaNDKと略す) いずれのタンパク質もほぼ中央に制限酵素サイトCpo Iを一つだけ有していた。そのサイトでそれぞれのタンパク質の前半、後半部分の繋ぎ換えを行い、大腸菌内で発現させた。結果としてHaNDKのN末側前半部とPaNDKのC末側後半部からなるキメラタンパク質 (以下HaPa-NDKと略す)、およびその逆のPaNDK・N末側前半部とHaNDK・C末側後半部で構成されるキメラタンパク質 (以下PaHa-NDKと略す) を得た (図2)。また、野生型HaNDK, PaNDKとそれらのキメラタンパク質HaPa-NDK並びにPaHa-NDKをethyleneglycol-bis (succinic acid-N-hydroxysuccinimide ester) を用いてサブユニット間のクロスリンクを行った。
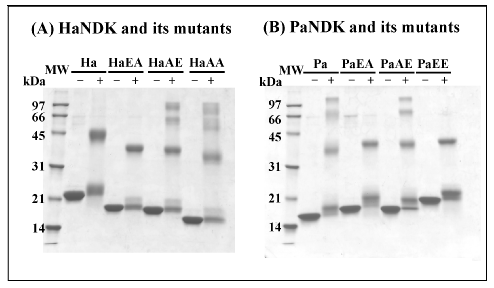
図2に示すようにHaPa-NDKは4量体、一方PaHa-NDKは2量体で存在しており、またPaHa-NDKはSDS-ポリアクリルアミド電気泳動 (以下SDS-PAGEと略す) でHaNDKと同様移動度の遅れを生じ好塩性タンパク質の特徴を獲得したことも明らかとなった9)。このことよりHaNDKにおいてはHaNDKの2量体-2量体間の接触面はN末端前半部分でなくC末端部分であることが確認できた。図2で示したように、このアミノ酸配列はHaNDK, PaNDKではそれぞれAYFFEESE, AYFFAATEであり、下線をつけた133, 134番残基が特徴的に異なっていた。そこでHaNDKの133,134番残基をEA, AE, AAに変換した変異体 (それぞれ順にHaEA, HaAE, HaAAと略す) とPaNDK 133,134番残基をAE, EA, EEに変換した変異体 (それぞれ順にPaAE, PaEA, PaEEと略す) を作成した。図3で明らかなように多量体構造の変換に影響があったのは133番残基で、133番残基をグルタミン酸からアラニンに変えたHaAE, HaAAは4量体に、アラニンからグルタミン酸に変えたPaEA, PaEEは2量体へと構造が変化した。134番残基の変異は影響がなかった。モデリングによるHaNDKの仮想4量体構造中において、2量体2量体間の接触面で133番残基がきわめて近接した位置をとると考えられ、この位置にグルタミン酸が来ると立体障害 (Glu側鎖と向かい側サブユニット主鎖が衝突する) およびマイナス荷電の反発により安定な4量体構造ができないと推測できた。HaNDKのX線解析によりこの推測の正当性が確認できた (新井ら 投稿中)。
![]()
4. 非好塩性タンパク質PaNDKにおける「好塩性と構造可逆性」の獲得
好塩性タンパク質は酸性アミノ酸残基を多く含むが故にSDS-PAGE上で本来の分子量から期待される位置より遅れて泳動される。図2および図3で見られるように好塩性タンパク質HaNDKは非好塩性タンパク質PaNDKに比べて泳動位置が異常に遅れる。この
そこで、この2つの変異体について好塩性タンパク質
の他の指標であるところの、
![]()
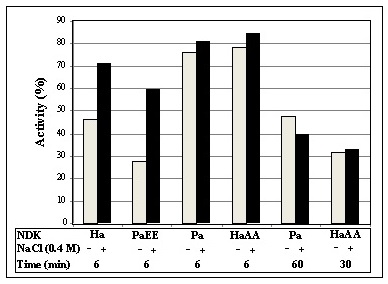
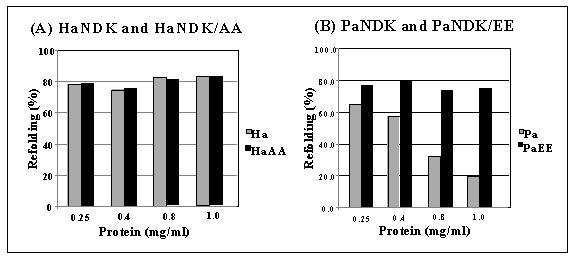
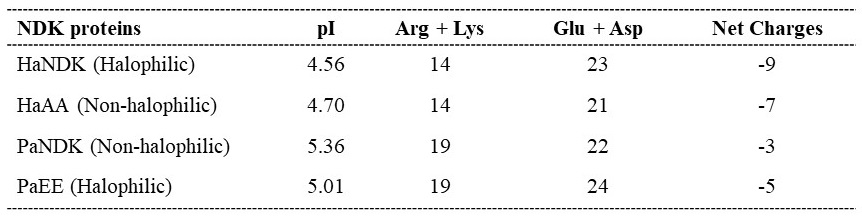
HaNDKは希薄タンパク質濃度溶液では不安定であるが、塩が存在すると安定性が向上する (図4, Ha参照)。野生型PaNDKは塩がある・なしにかかわらず安定性に変化はないが、PaEE変異体では安定性に対する塩の添加効果が出現した。一方、HaAA変異体はこの塩添加効果を消失していた。また、至適反応塩濃度に関してもPaEE変異体は50 mM NaCl存在下で最大活性を示しHaNDK野生型と同じようなパターンとなった。HaNDKは85℃, 5分の熱処理後でも約80%以上の酵素活性を示す。これは熱変性後のタンパク質巻き戻り活性であり、好塩性タンパク質が示す興味ある性質の一つである。この巻き戻り活性がタンパク質濃度に依存せずにPaEE変異体でもみられるようになった10) (図5)。前述したようにHaAA変異体は安定性に対する塩の添加効果の消失、さらに至適塩濃度も「好塩性」を失っていた。しかし、巻き戻り効率については実験範囲内では野生型と同様に高い効率を保持していた。表1 のようにHaAA、PaEEの等電点は4.70, 5.01となっており構造可逆性は他の好塩性タンパク質指標と比較して総電荷値 (等電点の低さ) により強く依存しているように見える。PaEEはHaAAよりも高い等電点を有しているにもかかわらず、「好塩性」指標であるところの塩による安定性増加、活性増加、SDS-PAGE上での移動度の変化を示している。これは「好塩性」に大きな役割を担っている負の総電荷に加えて特定位置のマイナス荷電 (この例では133,134番目) が重要であることを示唆している。
![]()
表1 NDKタンパク質の等電点とネットチャージ
5. タンパク質表面に存在する荷電変化と熱変性からの活性回復 (構造可逆性)
野生型HaNDK, PaNDKとキメラタンパク質PaHa-NDK, HaPa-NDKにHisタグを付加して大腸菌で発現させると、熱変性からの巻き戻り活性が観察できるのは低濃度タンパク質条件下His-HaNDK, His-HaPa-NDKの2つであった。Hisタグのない状態ではタンパク質濃度をうまく選べばこの4種類とも巻き戻り活性をつかまえられるので、Hisタグの正電荷が熱変性からの巻き戻りを邪魔しているのであろうと考えられる。この活性回復が制限された状態を利用し、His-PaHa-NDK (巻き戻り活性を示さない) の表面上に位置している塩基性アミノ酸を順次酸性アミノ酸へ置換しその活性回復に与える効果を調べた。一例を示すと44, 60番目の塩基性アミノ酸を、2つとも酸性アミノ酸に変換した場合ほぼ野生型 (HaNDK) に匹敵する結果を得ることができた。変異導入タンパク質の比活性はHis-PaHa-NDKのそれと大きくは変わらない。これはとりもなおさずタンパク質の表面に負電荷が増加するとタンパク質の熱変性に対する可逆性が増すことを実験的に明らかにできたこととなる。
6.おわりに
今回述べた「好塩化」することでPaNDKタンパク質がより柔軟になるというのは、特別な例であるかもしれないが、タンパク質の長い進化過程を経て現在に至っている酵素タンパク質を参考にして、種々の性質を付加した新たな酵素タンパク質を創製できる1つの可能性が見出せたと考えている。筆者らは「好塩性酵素工学」の重要性と汎用性を提唱している。
文献
1) Madern, D., Ebel, C., Zaccai, G.: Extremophiles, 4, 91 (2000).
2) Inouye, K., Kuzuya, K., Tonomura, B.: Biochim. Biophys. Acta, 1388, 209 (1998).
3) Premkumar, L., Bageshwar, U. K., Gokhman, I., Zamir, A., Sussman, J. L.: Protein Expr. Purif., 28, 151 (2003).
4) Mehta, A., Orchard, S.: Mol. Cell Biochem., 329, 3 (2009).
5) Nordman, J., Wright, A.: Proc. Natl. Acad. Sci., 105, 10197 (2008).
6) Besir, H., Zeth, K., Bracher, A., Heider, U., Ishibashi, M., Tokunaga, M., Oesterhelt, D.: Febs Lett., 579, 6595 (2005).
7) Yonezawa, Y., Izutsu, K., Tokunaga, H., Maeda, H., Arakawa, T., Tokunaga, M.: FEMS Microbiol. Lett., 268, 52 (2007).
8) Williams, R. L., Oren, D. A., Munoz-Dorado, J., Inouye, S., Inouye, M., Arnold, E.: J. Mol. Biol., 234, 1230 (1993).
9) Tokunaga, H., Ishibashi, M., Arisaka, F., Arai, S., Kuroki, R., Arakawa, T., Tokunaga, M.: FEBS Lett., 582, 1049 (2008).
10) Tokunaga, H., Arakawa, T., Tokunaga, M.: Protein Sci., 17, 1603 (2008).