【トピックス】
短鎖機能性ペプチドの探索と配列活性相関
本多裕之
名大院・工
1.はじめに
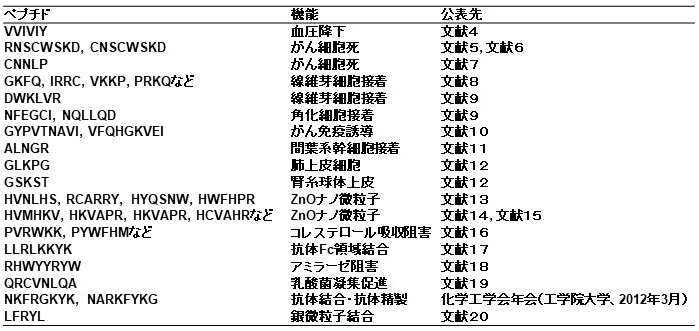
近年、タンパク質や細胞の機能制御に関与する低分子化合物の探索が進められている。ペプチドはタンパク質の一部であり、生体反応に関与する低分子物質として注目を集めている。また、ペプチドはアミノ酸20種類の組み合わせであることから6-merであっても6400万種類にも及ぶ膨大な多様性をもち、安全性も高いことから医療用材料や医薬品への応用が期待される。我々は、高次構造をとらない短鎖機能性ペプチドに絞って、ペプチドアレイ1,2)による機能性ペプチドの探索を研究している。ペプチドアレイは1992年にFrank1)により考案されたもので、国内では國松ら2)の研究が進んでいた。我々は、國松先生に師事しノウハウの享受を受けつつ研究を進め、通常のセルロースメンブレン型ペプチドアレイと、独自に開発した細胞機能アッセイ法 (PIASPAC法)3)を用いて、表14-20)に示すような種々の機能性ペプチドの探索に成功してきた。以下にその例を示し、ライブラリーとしてのペプチドアレイの有用性を解説する。
表1 我々の研究室で探索した機能性ペプチドのリスト
2.セルロースメンブレン型ペプチドアレイ
ペプチドアレイとは多種類のペプチドを固定化したアレイの総称を指す。通常はセルロースメンブレン上の任意の位置に少量の活性化アミノ酸を繰り返しスポットすることでF-moc固相合成反応による伸長反応を行い、任意の配列を合成するが (図1)、アミノ基コートガラス表面にも合成可能である。合成にはピン式スポット合成装置 (Intervis製) を用いており、ペプチドはC末端がメンブレン表面に結合した状態にある。ペプチドアレイは (i) 任意の配列をもつ多種類のペプチドを任意の残基数で設計可能、 (ii) 実験に用いたすべてのペプチドに関して活性データが得られ、 (iii) 非天然のアミノ酸を含んだペプチドも合成可能、という利点をもつ。従来、ペプチドアレイはエピトープマッピングなどを中
心とした分子間相互作用を調べるために用いられている21,22)。
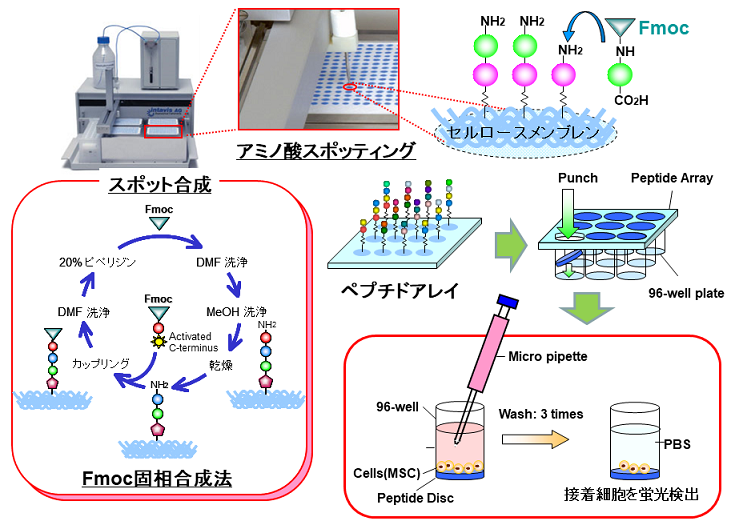
図1 ペプチドアレイの作製と細胞アッセイ法
3.ペプチドアレイ細胞機能アッセイ法 (PIASPAC法) による機能性ペプチドの探索
細胞に対して機能する分子の探索は重要である。そこで我々は、ペプチドアレイをディスク状にパンチアウトし、96穴細胞培養プレートに沈め、あらかじめ蛍光ラベルした目的細胞を、培養液とともに培養プレートに添加し、細胞接着を蛍光プレートリーダーで観察する細胞アッセイ技術を確立した (図1)。我々はこれを、“Peptide array-based interaction assay of solid-bound peptide and anchorage dependent cells (PIASPAC法) ”と名付け、アレイ表面のペプチドと細胞表面上の受容体 (ex.インテグリン) との直接相互作用が検出できることを示した3,5,6,7,11)。
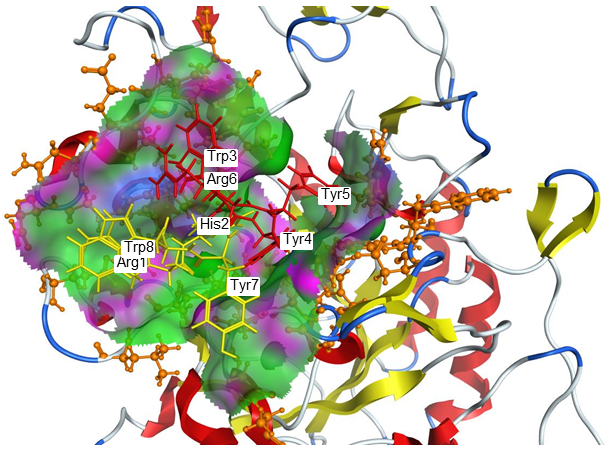
癌細胞選択的にアポトーシス (周囲の正常な組織に影響を与えない細胞死) を誘導するタンパク質としてTRAIL (TNF related apoptosis inducing ligand) が知られている。TRAILと同様な機能を持ったペプチドの探索を目指し、TRAIL配列ライブラリーを作製し、PIASPAC法で探索を行ったところ、RNSCWSKD配列を得た。さらに、残基置換ライブラリーを作製したところ、さらに高い活性をもつ配列CNSCWSKD を得た。可溶化ペプチドを合成し、ヒトリンパ腫細胞Jurkatの培養液に2 mMの濃度で加えたところ、12時間で10%以下の生細胞率となり、24時間で50%以下にまで細胞死に至る濃度は0.3 mM程度であり、RNSCWSKD配列より5倍程度活性が高いこともわかった5,6)。
間葉系幹細胞 (MSC; mesenchymal stem cell) は骨芽細胞・脂肪細胞・軟骨細胞・筋細胞などに分化することができる分化能と自己複製能を合わせもつ細胞であり、再生医療に有効な細胞源である。しかし、MSCは骨髄中にわずかしか存在しないため少量の細胞を効率よく接着させ、増殖させることが重要である。我々はフィブロネクチンのアミノ酸配列をもとに5残基ペプチドライブラリーを作製、MSC接着ペプチドの探索を行った。その結果、細胞接着ペプチドとして知られているRGDと同等の活性を持った配列ALNGR (1430から1434番目に位置) を単離することに成功した11)。
ペプチドアレイでは高活性配列と低活性配列が同時に得られる。この実験結果と、ペプチドの配列データをコンピュータ解析することで、配列機能相関を得ることができる。配列機能相関が得られると、その情報に基づいてさらに探索範囲を絞ったスクリーニングが可能になる。我々はこの方法をペプチドインフォマティクスと呼んでいる。この方法によればファージディスプレイ法などの表面提示型ライブラリースクリーニングよりも確実に機能性ペプチドが探索できる。以降は、このペプチドインフォマティクスによる探索結果について紹介する。
4.ファージディスプレイとの比較:酸化亜鉛認識ペプチド
機能性ペプチドの探索を目指した場合、どういう方法が考えられるであろうか。通常はファージディスプレイ法に代表されるような表面提示型ライブラリーでの探索であろう。この方法は万能であろうか。実は、表面提示型ライブラリーでのスクリーニングに不安を感じている研究者は少なくない。確かに結合するペプチドは得られたけれど、果たして本当にこれが最も高い結合を示すペプチドなのか (検出漏れの危険性)、あるいは、これ以外に同様の活性をもつペプチド配列はないのだろうか (類似性の危険性) という不安である。
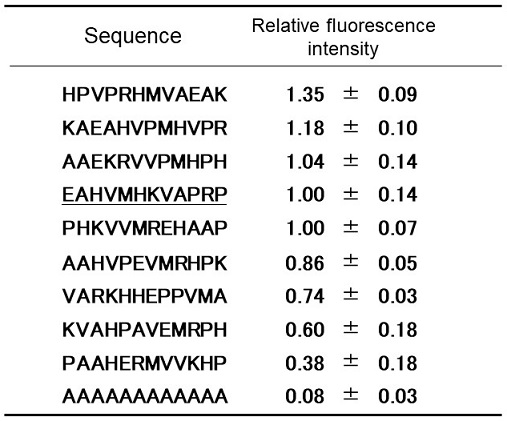
我々は、ファージディスプレイ法で得られた金属微粒子ZnOを認識するペプチドEAHVMHKVAPRP23) をシード配列にして、組成が同じで配列の異なるバリアントペプチドをペプチドアレイに合成し、ZnOとの認識能を評価した。その結果、シード配列より高い認識能をもつ配列の探索に成功した (表2)。また、この配列をベースとして6-merのペプチドを探索したところ、2種類の重要部位HVMHKV、HKVAPRの単離に成功し、さらにHKVAPRの残基置換によりHCVAPRという非常に高結合を有するペプチドの探索に成功した14,15)。これは、 (i) ファージディスプレイ法による探索が万能ではなく、スクリーニング漏れがあり、また (ii) 長鎖でなくても有用なペプチドがあることを示している。とり漏れはファージ粒子との相互作用が無視できないのかもしれない。短鎖でもよいものがあるのは、酵素−基質の認識が3から4個のアミノ酸残基で起きることにつながるのではないかと考えている。この結果は、ファージディスプレイ法だけでなく、他の表面提示型ライブラリー探索 (酵母表面提示法や無細胞タンパク質翻訳提示法) でも同様の問題をはらんでいるのではないかと考えている。
表2 EAHVMHKVAPRPペプチドの配列組み合わせ置換ペプチドのZnO結合活性
表面提示型ライブラリー探索法は109オーダーのライブラリーサイズを持っているといわれている。ペプチドアレイでそのオーダーの合成はできない。ではどうするか。この疑問を解決するため、我々は、数百から1000種類程度のペプチドアレイからコンピュータ解析でルールを抽出して探索する方法を考案した。以下にその概要と成果を説明する。
5.酸化亜鉛認識ペプチドの認識ルール
ペプチドアレイで探索してみると、同程度の高機能性をもつ多種類のペプチドがあることに気づく。これら高機能性ペプチドは共通した特徴を持つと考えられる。そこで、ペプチドアレイで解析した結果を、残基の大きさ、電荷、疎水度といった代表的なアミノ酸特徴量で整理してコンピュータ解析してルールを得ることを試みた6)。6残基のランダムペプチド422種類をスポッティングしたランダムライブラリーを作製して、先のZnO認識ペプチドのスクリーニングを行った。入力データとして、各位置の3つの特徴量 (18入力) を用意し、高結合30配列、低結合30配列のデータを、我々が解析で汎用しているファジィニューラルネットワーク (FNN) で解析した。その結果、「N末端アミノ酸のサイズが比較的大きく、4残基目のアミノ酸の電荷が大きいとき、酸化亜鉛粒子に接着しやすい」という粒子認識ルールを得ることができた (図2)13)。実際にこのルールに合致するペプチドではランダムペプチドに比べ平均1.6倍接着しやすいことが実証できた。また、結合活性の高いペプチド配列を可溶化し、微粒子との親和性を調べた結果、他の酸化金属微粒子と比べて、高い酸化亜鉛微粒子特異性を持っていることが明らかになった。これらのペプチドは分子イメージング素子、配向素子としての可能性がある。
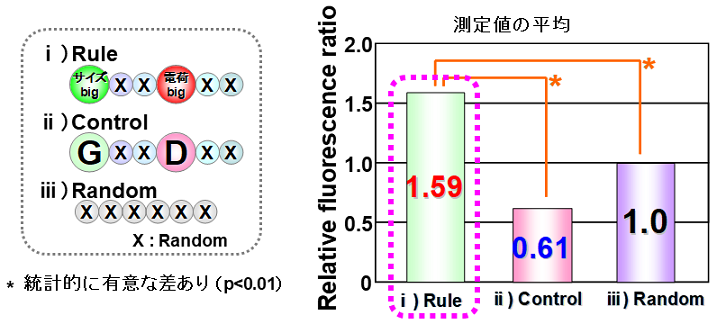
図2 酸化亜鉛微粒子結合ペプチドのルール
6.コレステロール吸収抑制ペプチドのルール
コレステロールは胆汁酸ミセルに取り込まれて小腸壁から吸収される。大豆由来のペプチドVAWWMYは、胆汁酸に結合し、ミセル崩壊を促すことでコレステロールの吸収を抑制できる24)。この原理に基づき、2212種類のペプチドライブラリーをペプチドアレイで作製し、胆汁酸との結合を評価した。アミノ酸特徴量として、疎水度、電荷、サイズを選択して解析したところ、胆汁酸結合ペプチドでは、「N末端3残基目のアミノ酸のサイズが大きく、4残基目のタンパク質安定化指標が大きいと胆汁酸結合活性が高い」といったルールが得られた。このルールで合成すると高活性のペプチドが45%の割合で得られた (ランダムに作成した場合わずか6%) (図3)16)。また、既知の大豆グリシニンタンパク質由来VAWWMYペプチドよりも高結合性を示す28配列が得られた。高活性ペプチドをラットに投与したところ、実際にコレステロール吸収阻害を起こしたことから、今後機能性食品への応用が期待される (図4)。
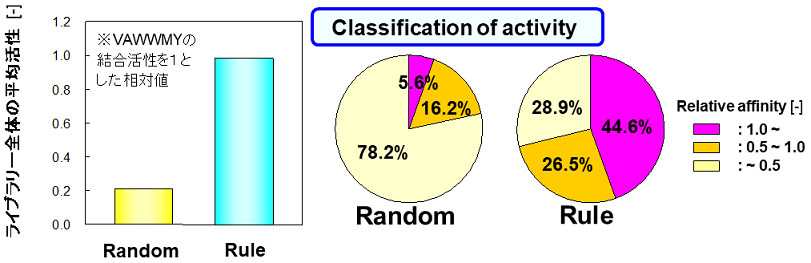
図3 胆汁酸結合ペプチドのルール
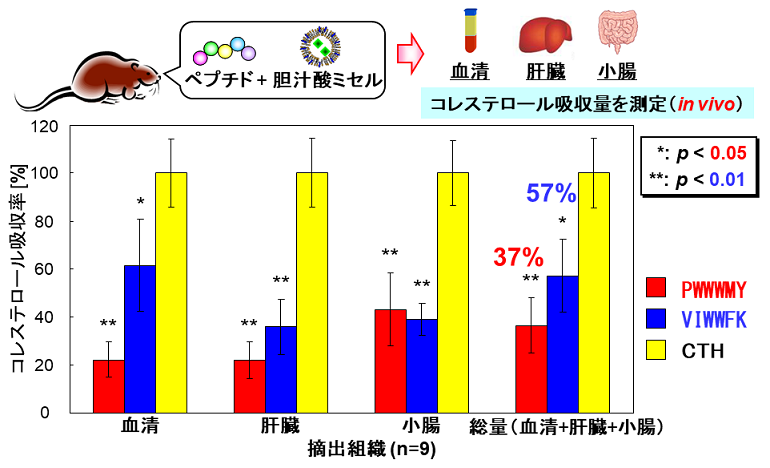
![]()
7.細胞接着ペプチドのルール
上述のPIASPAC法を用いて、同様の実験を線維芽細胞接着ペプチドに対しても実施した。その結果、「N末端アミノ酸のサイズが小さく、2残基目の電荷が大きく、3残基のサイズと電荷が大きく、4残基目のサイズが小さければ細胞接着ペプチドとして機能する」というルールが発見でき、このルールに従ってペプチドをランダムに作製したところ、活性の高い配列は84%と約4倍以上含まれることがわかり、線維芽細胞の接着に重要なルールであることが実証できた (図5)8)。これまでに知られていない配列も多く、再生医療での細胞選別や再狭窄を防ぐ人工血管表面修飾分子として使えるペプチドになる。
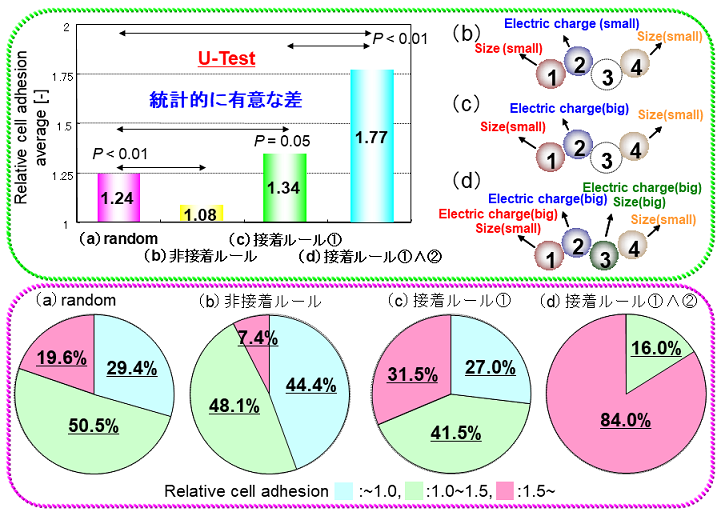
図5 細胞接着ペプチドのルール
8.その他のペプチド
ペプチドアレイは固相合成法でありC末端で拘束されている。この点を解決するため、紫外線照射で解裂・遊離できるフォトリンカー (4-[4-(1-(Fmocamino) ethyl)-2-methoxy-5-nitrophenoxy] butanoic acid) を合成し、基質結合部位に深く結合し拮抗阻害を引き起こすペプチドの探索を行った。膵液アミラーゼを阻害し得るペプチドをこのペプチドアレイで探索した結果、RHWYYRYWが探索できた (図6)18)。このペプチドは既往の糖尿病治療薬、アカルボースよりも親和性が高く、新たな糖尿病治療薬になることが示唆された。
![]()
9.おわりに
ペプチドはバリエーションが多く、探索できるライブラリーサイズとしては極めて大きい。ペプチドライブラリーで探索した結果に基づいて、ペプチドミミック化合物を合成することで新規の薬剤や生理活性化合物を合成することも可能になるであろう。
あらかじめペプチドの特徴を網羅したペプチドを選択しておけば、スクリーニングアレイという使い方ができる。たとえばアミノ酸の3つの特徴量について、その大小のアミノ酸候補を選べば、6残基ペプチドでも66=46656種類のペプチドを合成すれば良い。このペプチドを、DNAマイクロアレイのようにインクジェット方式で固定する方法を適用すれば、4万6千種のペプチドからなるスクリーニングアレイが作成できる。数年後には、多くの研究者がスクリーニングアレイを手にして、ペプチドの無限の可能性に挑戦できるようになるのではないかと期待している。
謝辞
ペプチドの研究に携わってくれた多くの学生諸氏に感謝します。また胆汁酸結合ペプチドに関しては岐阜大学の長岡利先生との共同研究の成果であり、コレステロール吸収阻害のアイディアや動物実験に関する多大なご支援をいただいた。この場を借りて深謝申し上げます。
文献
1) Frank, R.: Tetrahedron, 48, 9217 (1992).
2) 國松己歳:ゲノム医学, 5, 87 (2005).
3) Kato, R., Kaga, C., Kunimatsu, M., Kobayashi, T., Honda, H.: J. Biosci. Bioeng., 101, 485 (2006).
4) Kato, R., Kunimatsu, M., Fujimoto, S., Kobayashi, T., Honda, H.: Biochem. Biophys. Res. Commun., 315, 22 (2004).
5) Okochi, M., Nakanishi, M., Kato, R., Kobayashi, T., Honda, H.: FEBS Lett., 580, 885 (2006).
6) Kaga, C., Okochi, M., Nakanishi, M., Hayashi, H., Kato, R., Honda, H.: Biochem. Biophys. Res. Commun., 362, 1063 (2007).
7) Kato, R., Okuno, Y., Kaga, C., Kumnimatsu, M., Kobayashi, T., Honda, H.: J. Pept. Res., 66, 146 (2006).
8) Kaga, C., Okochi, M., Tomita, Y., Kato, R., Honda, H.: Biotechniques, 44,393 (2008).
9) Nomura, S., Kaga, C., Kato, R., Okochi, M., Honda, H.: J. Chem. Eng. Japan, 41, 206 (2008).
10) Okochi, M., Hayashi, H., Ito, A., Kato, R., Tamura, Y., Sato, N., Honda, H.: J. Biosci. Bioeng., 105, 198 (2008).
11) Okochi, M., Nomura, S., Kaga, C., Honda, H.:Biochem. Biophys. Res. Commun., 371, 85 (2008).
12) Kaga, C., Nomura, S., Okochi, M., Nozu, T., Nagao, T., Nagai, A., Nakayama, T., Suzuki, K., Honda, H.: J. Chem. Eng. Japan, 42, 298 (2009).
13) Okochi, M., Ogawa, M., Kaga, C., Sugita, T., Tomita, Y., Kato, R., Honda, H.: Acta Biomater., 6, 2301 (2010).
14) 本多裕之:生物工学会誌, 87,280 (2009).
15) Okochi, M., Sugita, T., Furusawa, S., Umetsu, M., Adschiri, T., Honda, H.: Biotechnol. Bioeng., 106, 845 (2011).
16) Takeshita, T., Okochi, M., Kato, R., Kaga, C., Tomita, Y., Nagaoka, S., Honda, H.: J. Biosci. Bioeng., 112, 92 (2011).
17) 片山 真, 杉田智哉, 加藤竜司, 大河内美奈, 松島充代子, 川部 勤, 高瀬智和, 吉田安子, 川瀬三雄, 本多裕之:化学工学論文集, 37, 546 (2011).
18) Ochiai, T., Sugita, T., Kato, R., Okochi, M., Honda, H.: Biosci. Biotechnol. Biochem., 76, 819 (2012).
19) Asai, Y., Sugita, T., Kato, R., Okochi, M., Nakagawa, K., Honda, H.: J. Chem. Eng. Japan, in press (2012) .
20) Kuboyama, M., Kato, R., Okochi, M., Honda, H.: Biochem. Eng. J., 66, 73 (2012).
21) Kramer, A., S.-Mergener, J.: Methods. Mol. Biol., 87, 25 (1998).
22) Reineke, U., Volkmer-Engert, R., Schneider-Mergener, J.: Curr. Opin. Biotechnol., 12, 59 (2001).
23) Umetsu, M., Mizuta, M., Tsumoto, K., Ohara, S., Takami, S., Watanabe, H., Kumagai, I., Adschiri, T.: Adv. Mater., 17, 2571 (2005).
24) Nagaoka, S., Nakamura, A., Shibata, H., Kanamura, Y: Biosci. Biotechnol. Biochem., 74, 1738 (2010).