【トピックス】
立体構造を基盤としたナイロン加水分解酵素の開発と耐熱化機構
根来誠司、永井圭介、大島祥平、飯田一希、加藤太一郎、武尾正弘、持地広造、柴田直樹、樋口芳樹
兵庫県大院・工、兵庫県大院・工、兵庫県大院・工、兵庫県大院・工、兵庫県大院・工、兵庫県大院・工、兵庫県大院・工、兵庫県大院・生命理、兵庫県大院・生命理
1.はじめに
地球温暖化への対策として、環境負荷の低減化は重要な検討課題であり、その解決策の一つとして、「生分解性プラスチック」の開発が重要視されている。一方、ε-カプロラクタムの開環重合により合成される6ナイロンや、アジピン酸とヘキサメチレンジアミンとの重縮合により合成される66ナイロンは、強靱で、耐熱性、耐薬品性に優れている。そのため、繊維・プラスチックとして広く利用されており、合成ポリアミド全体の約90 %を占めるが、生物分解を殆ど受けない素材である。強靱性と柔軟性とを併せ持ったナイロン固有の特性は、各ポリマー分子鎖が規則正しく並び水素結合により安定化した結晶性領域と、不規則な非晶領域とが、適切に混在しているためである (図1A)。ナイロンの結晶化度は、20〜50 %であり、結晶化度によって融点・強度等の物性は変化する。結晶性領域として、各分子鎖が相互に逆平行に並んだα結晶と、平行に並んだγ結晶が知られているが、6ナイロンでは、α結晶が安定である1)。
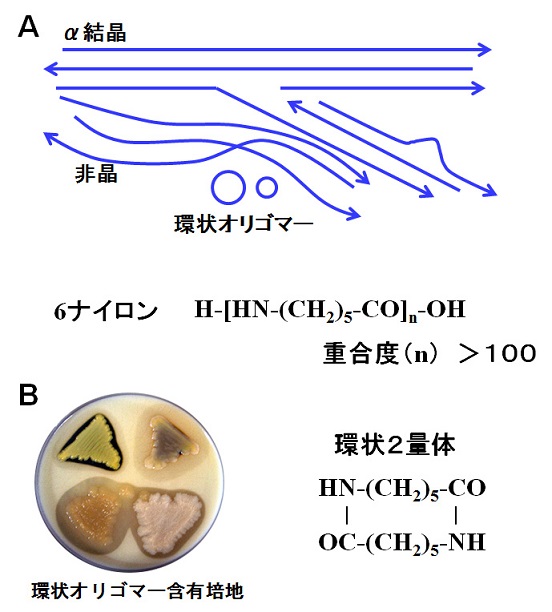

我々は、これまで、ナイロン工場排水などを分離源として、6-アミノカプロン酸オリゴマーを加水分解する約10種類の微生物を取得した2-5)。何れも、同基質を唯一の炭素源・窒素源とした培地で増殖する (図1B)。代表的な分解性微生物について生化学的検討を行った結果、分解様式の異なる3種の酵素、NylA (環状2量体分解酵素)6)、NylB (エキソ型オリゴマー分解酵素)7-12)、NylC (エンド型オリゴマー分解酵素)13,14)を見いだした。近年、耐熱化したNylC変異酵素が、60 ℃の酵素反応で、ポリマーのナイロンに対して分解活性を示す事が確認でき、現在、NylCはナイロン加水分解酵素 (nylon hydrolase) として認定されている15)。本稿では、NylCの特徴、立体構造を基盤とした同酵素の耐熱化機構、および、産業応用への可能性について紹介する。
2.NylCの特徴と立体構造
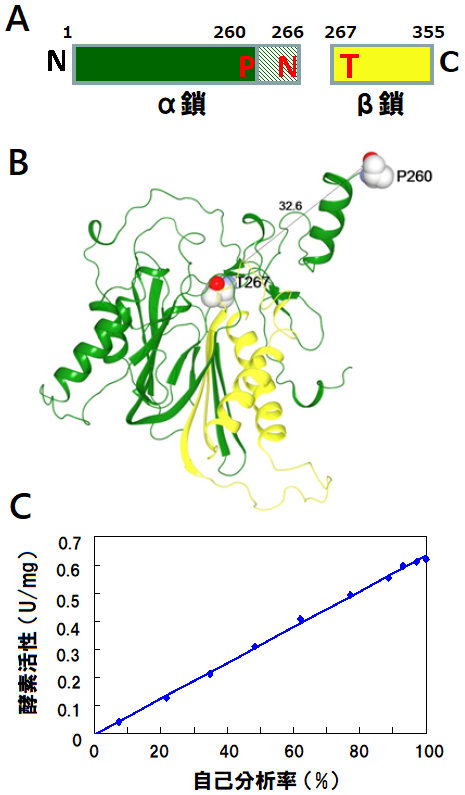
NylCは、Arthrobacter (プラスミドpOAD2上にコード)、好アルカリ性細菌Agromyces、Kocuriaから見いだされている (各々、NylCp2、NylCA、NylCKと命名)。何れも前駆体 (36 kDa) として発現し、Asn266/Thr267間で分断され、α鎖 (27 kDa) とβ鎖 (9 kDa) に分かれる (図2A)。認識配列C末端側の求核性残基 (Thrなど) が自己分断反応を触媒するとともに、分断後は、基質分解における触媒基となる。上記の特徴は、N末端求核性 (N-tn) ファミリーの酵素群に共通している。立体構造解析から、NylCはα鎖とβ鎖がヘテロ2量体を形成し、4本のαヘリックスが2枚のβシートを挟んだαββαサンドイッチフォールドをとることが明らかとなった (図2B)15)。この構造は、D-アミノペプチダーゼ (PDBID:1B65)、β-ペプチジルアミノペプチダーゼ (PDBID:3N5I) などの他のN-tnファミリー酵素のフォールドと類似している。さらに、ネイティブ酵素では、4分子のヘテロ2量体が会合してドーナツ型の分子構造を形成する (図3)。


3.自己分断の分子機構
N-tnファミリーには、自己分断の認識配列として、Asn/Thr型の酵素以外に、Asn/Ser型 (β-ペプチジルアミノペプチダーゼ, アシルホモセリンラクトンアシラーゼ)、Asp/Thr型 (グリコシルアスパラギナーゼ)、Gly/Ser型 (D-アミノペプチダーゼ)、Gly/Thr型 (γ-グルタミルトランスペプチダーゼ)、Gly/Cys型 (イソペニシリン-N-アシルトランスフェラーゼ)、Ile/Cys型 (N-アシルスフィンゴシンデアシラーゼ) などの酵素も報告されている16-22)。我々は、NylCの分断部位の変異酵素を用いた以下の実験から、自己分断が本酵素の活性化に必須であることを明らかにした。
i) Ser267変異酵素では、精製直後は90 %以上が前駆体であるが、37 ℃で保温すると、時間経過とともに自己分断が進行し、48時間後に完全に分断される。また、分断された割合と酵素活性との間に、比例関係が成立する (図2C)。
ii) Ala266/Ala267二重変異酵素では、前駆体状態として得られる。また、37 ℃で保持しても自己分断は進行せず、ナイロン関連基質に対する分解活性も検出限界以下である。
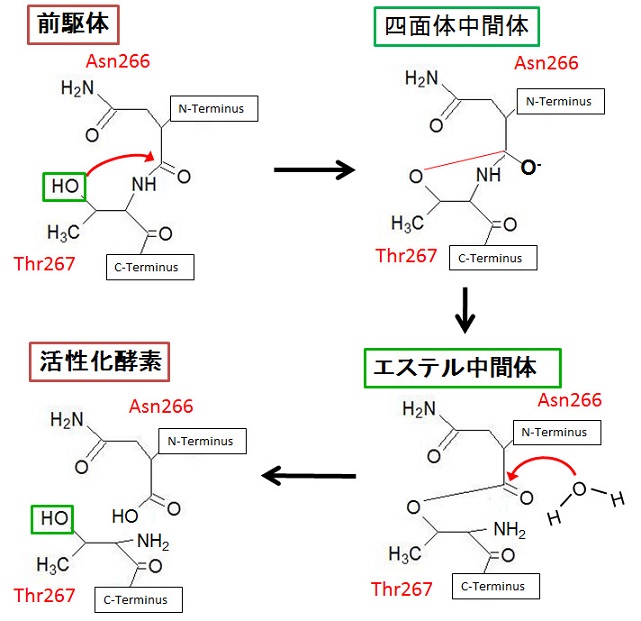
活性型NylCと前駆体の立体構造、およびファミリー酵素の知見から、NylCの自己分断機構を次のように推定した (図4)。
i) 前駆体のThr267-OHが、Asn266/Thr267間のカルボニル炭素を求核攻撃し、四面体中間体を生成する。
ii) 四面体中間体は、N-Oアシルシフトにより、エス
テル中間体に変換される。
iii) エステル中間体は、近接水分子により加水分解される。分断で生じたβ鎖のN末端残基 (Thr267) が、基質分解における触媒基となる。
iv) α鎖のC末端領域は、構造変化により、分断部位とは離れた位置に移動する。Pro260とAsn266の間のループ領域の電子密度は観察されないが、Pro260はThr267から32.6 Å離れている (図2)。

4.耐熱性酵素の取得
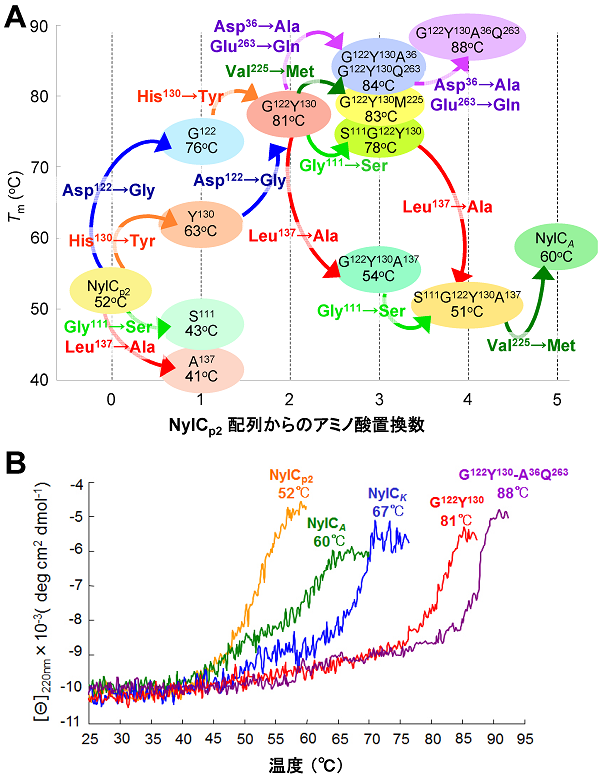
NylCAのアミノ酸配列は、NylCp2の配列と5カ所で異なり、NylCKでは、共通する5カ所以外に、さらに10カ所で異なっている4)。NylCAとNylCKの熱安定性はNylCp2よりも、各々、8〜15 ℃高い (図5)。一方、触媒効率 (kcat/Km) は、何れもNylCp2よりも13倍高く、好アルカリ性細菌由来のNylCは、耐熱性・活性ともに、NylCp2よりも優れている4)。
NylCp2に、NylCA型・NylCK型のアミノ酸置換を行い、熱安定性の加算効果について検討した。NylCp2とNylCA間で異なっている5アミノ酸置換のうち、D122G、H130Y、V225Mは熱安定性に対してプラスに、G111S、L137Aはマイナスに作用することがわかる (図5)。また、NylCAとNylCK間で異なっている10置換のうち、サブユニット界面に位置するE263Q、D36Aに着目し、D122G/H130Y二重変異酵素 (G122Y130) (Tm=81 ℃) に導入したところ、Tm値が88 ℃まで上昇し、熱安定性が最も高い変異酵素 (G122Y130A36Q263) を得ることができた。さらに、マイナスに作用する変異効果を含めると、5アミノ酸置換で、47 ℃の温度範囲で熱安定性が変化することが分かった15) (図5)。

5.タンパク質の熱安定化機構
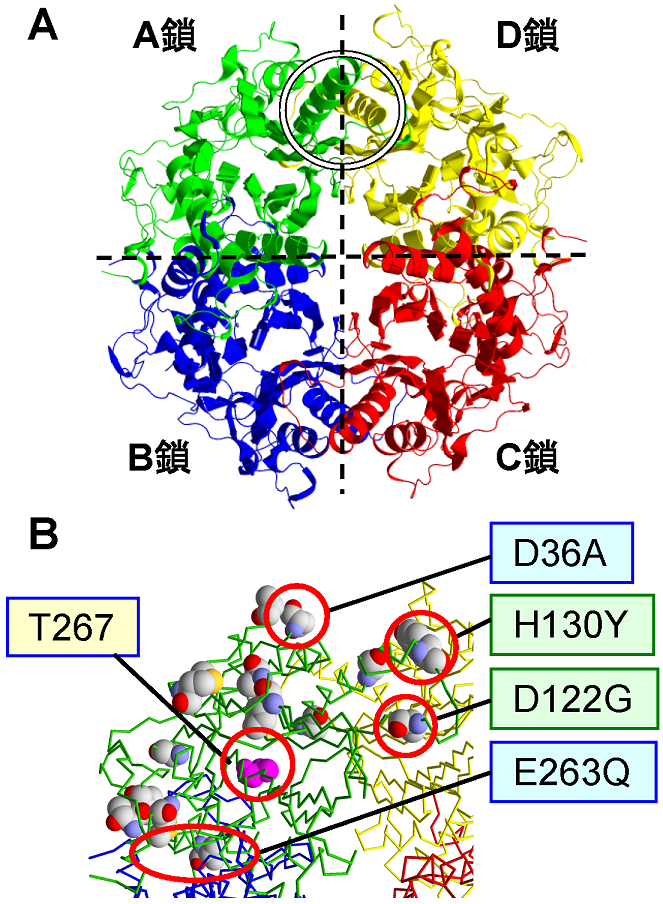
NylCの耐熱性は、サブユニットA/D界面 (D122G, H130Y, L137A, G111S, D36A)、およびA/B界面 (V225M, E263Q) のアミノ酸置換で大きく変化する (図5)。以下に、各置換の構造安定性に及ぼす効果について考察する15)。
122位:NylCテトラマーは、D2対称 (3つの二回回転軸が互いに直交) をとることから、A鎖とD鎖のへリックスH4は、図3Aに示す位置関係で近接している。A鎖の122位近傍では、塩基性アミノ酸のLys159-NH3+ (A鎖) と酸性アミノ酸のGlu115-COO− (D鎖) とが近接しているため (2.79 Å) 、両残基は静電的に安定化している。NylCAでは、122位がGlyであるが、親型NylCp2では、Gly122がAspに置換されているため、Asp122 (A鎖) とGlu115 (D鎖) との間で静電的反発を生じ、不安定化することが予想される (図6A)。すなわち、親型NylCp2へのD122G置換は、安定性を上昇させる (図5A)。
130位:Tyr130-Oη (A鎖) は、へリックスH4末端のGlu126-Oεと水素結合を形成し、ループを安定化する (図6B)。一方、NylCp2では、Tyr130がHisに置換されているため130位での水素結合が失われ、ループ領域が不安定化すると推定できる。したがって、親型NylCp2へのH130Y置換は、耐熱性を上昇させる。
137位:137位は、H130Y変異により安定化するループの他端に存在する (図6B)。G122Y130、S111G122Y130変異酵素へのL137A置換は、何れも、耐熱性を27 ℃低下させる (図5A)。したがって、Leu137では、疎水的相互作用により同ループが安定化しているが、側鎖の小さいAla137置換体では、疎水的相互作用による安定化効果が低下すると推定できる。
111位:NylCAでは、親水性残基のSer111-Oγ (A鎖) が、隣接するD鎖の3個の疎水性残基〔Tyr98, Ala137 (NylCp2ではLeu137),Leu139〕と同一サブユニットのTyr112 (A鎖) に囲まれており、不安定化している (図6C)。NylCp2、G122Y130、G122Y130A137変異体では、111位がGlyであるため、Ser111に比べてA/D界面における安定性が高い。したがって、G111S置換は、親型酵素の安定性を低下させる。
36位:Asp36 (D鎖) は、隣接サブユニット (A鎖) のGlu126に近接している (4.40 Å) (図6B)。一方、3種の野生型NylCの中で、耐熱性が最も高いNylCKでは、Asp36 (NylCp2、NylCA)がAlaに置換されている。すなわち、D36A変異は、Asp36 (D鎖) とGlu126 (A鎖) との静電的反発を低減化させると推定できる。実際、G122Y130、G122Y130Q263変異体へのD36A置換は、耐熱性を上昇させる。
225位:Met225 (A鎖) は、隣接サブユニットとの直接の接触はないが、 Met225に近接するGln299 (A鎖) は、Arg296-Nη2 (B鎖) と接触する (3.01 Å) (図6D)。さらに、Gln299 はHis245-Nε2 (2.81 Å) と近接している。したがって、225位におけるアミノ酸置換は、間接的に、A/B界面の相互作用に影響を与えると推定できる。実際、G122Y130、S111G122Y130A137変異体へのV225M変異は、両酵素の耐熱性を上昇させる (図5)。
263位:Glu263はα鎖の末端に位置するが、この領域を含むVal261-Asn266の間は、NylCAのX線結晶構造解析では電子密度が観測されない。一方、耐熱性の高いNylCKでは、酸性アミノ酸のGlu263はGlnに置換されている (図3B)。したがって、Glu263に起因する静電的反発が解消され、サブユニットが安定化すると思われる。
以上のように、A/B界面、A/D界面におけるサブユニット間相互作用は、タンパク質の構造安定性と密接に関連していると結論できる。
6.ナイロンの酵素分解
ガスクラスター2次イオン質量分析法 (GC-SIMS) は、材料の表面分析に有効な手法であり、試料の励起段階で、クラスター化したアルゴンを照射することで、アルゴン1原子当たりのエネルギーを4eV以下に抑制し、試料中の親ピークの検出を可能とする方法である。本法により、固相表面上で進行する酵素作用を検出することが可能となり、NylCが6ナイロン、66ナイロン、修飾ナイロンへ作用することが示された (図7)。しかし、6ナイロンや66ナイロンの分解率 (全てのアミド結合が加水分解された時に生じるアミノ基に対して、実際に遊離するアミノ基の割合) は1〜2 %に止まる。すなわち、ポリマー会合体の一部の結合が切断されても、周囲のアミド結合は他の分子と水素結合を形成しており、高分子本体からのオリゴマーの離脱が進まない15)。言い換えれば、分解産物の連続的な離脱が進むニック数 (崩壊のための閾値) を超えるまでは、ポリマーは酵素反応に抵抗性を示す。したがって、ナイロン分解のための酵素側の要件として、i) 高温反応が可能な耐熱性酵素、ii) 単位鎖長当たり多くのニックを与える高活性酵素の取得が必要であり、ポリマー側の要件として、iii) 一定間隔でポリマー分子間の水素結合を攪乱するモノマーの挿入、iv) 強度を保ちながら重合度を低く抑え、且つ分子量分布の揃ったポリマーの構築が必要であると推定している。実際、以下の結果は、iii) のアプローチが有効であること示す。
アジピン酸クロリド (有機溶媒相) とヘキサメチレンジアミン (水相) からなる66ナイロンの界面重縮合反応系に、アジピン酸とは炭素鎖長の異なるモノマー (グルタル酸クロリド、コハク酸クロリド)、または、2級アミンのN,N’-ジエチルヘキサメチレンジアミンの何れかを添加して共重合せ、分解性に及ぼす効果を調べた。その結果、添加モノマーの種類・割合に応じて、NylCによる分解率が変化した (図8) (平成24年度日本生物学会大会発表)。同様に、アジピン酸、或いは、ヘキサメチレンジアミンとは炭素鎖長の異なるモノマーを用いて重合させたナイロンにおいて、ylCによる分解率が66ナイロンの約4倍に上昇し、前処理 (化学的限定分解) とNylB反応を組み合わせることで、初発のポリマーの80 %以上がモノー化する条件を見いだしている。したがって、原理的には、ナイロンから有機酸やアルコールなど、他の有用物質への酵素変換も可能であると思われる。ナイロンのケミカルリサイクル (化学処理によるモノマー化反応) では高温反応を要するが、上記の反応は全て100 ℃以下で実施するため、境・エネルギー面でも優位であろう。
現行の生分解性試験では、1ヶ月 (活性汚泥法)〜4ヶ月 (土壌分解性試験) という長期間を有するが、ナイロン加水分解酵素による分解性を指標にすると、数時間の酵素反応で判定できるため、生分解性ポリアミド開発における予備スクリーニングとしても利用できる。さらに、ナイロン繊維の表面加工など、今後、同酵素が産業分野で利用されることを期待している。
文献
1) Dasgupta, S., Hammond, W. B., Goddard, W. A.: J. Am. Chem. Soc., 118, 12291 (1996).
2) Negoro, S.: Appl. Microbiol. Biotechnol.w z, 54, 461 (2000).
3) Kato, K., Ohtsuki, K., Koda, Y., Maekawa, T., Yomo, T., Negoro, S., Urabe, I.: Microbiol., 141, 2585 (1995).
4) Yasuhira, K., Tanaka, Y., Shibata, H., Kawashima, Y., Ohara, A., Kato, D., Takeo, M., Negoro, S.: Appl. Environ. Microbiol., 73, 7099 (2007).
5) Yasuhira, K., Uedo, Y., Takeo, M., Kato, D., Negoro, S.: J. Biosci. Bioeng., 104, 521 (2007).
6) Yasuhira, K., Shibata, N., Mongami, G., Uedo, Y., Atsumi, Y., Kawashima, Y., Hibino, A., Tanaka, Y., Lee, Y.-H., Kato, D., Takeo, M., Higuchi, Y., Negoro, S.: J. Biol. Chem., 285, 1239 (2010).
7) Negoro, S., Ohki, T., Shibata, N., Mizuno, N., Wakitani, Y., Tsurukame, J., Matsumoto, K., Kawamoto, I., Takeo, M., Higuchi, Y.: J. Biol. Chem., 280, 39644 (2005).
8) Ohki, T., Wakitani, Y., Takeo, M., Yasuhira, K., Shibata, N., Higuchi, Y., Negoro, S.: FEBS Lett., 580, 5054 (2006).
9) Negoro, S., Ohki, T., Shibata, N., Sasa, K., Hayashi, H., Nakano, H., Yasuhira, K., Kato, D., Takeo, M., Higuchi, Y.: J. Mol. Biol., 370, 142 (2007).
10) Ohki, T., Shibata, N., Higuchi, Y., Kawashima, Y., Takeo, M., Kato, D., Negoro, S.: Protein Sci., 18, 1662 (2009).
11) Kawashima, Y., Ohki, T., Shibata, N., Higuchi, Y., Wakitani, Y., Matsuura, Y., Nakata, Y., Takeo, M., Kato, D., Negoro, S.: FEBS J., 276, 2547 (2009).
12) Kawashima, Y., Yasuhira, K., Shibata, N., Matsuura, Y., Tanaka, Y., Taniguchi, M., Miyoshi, Y., Takeo, M., Kato, D., Higuchi, Y., Negoro, S.: J. Mol. Cat. B: Enzymatic, 64, 81 (2010).
13) Kakudo, S., Negoro, S., Urabe, I., Okada, H.: Appl. Environ. Microbiol., 59, 3978 (1993).
14) Negoro, S., Kakudo, S., Urabe, I., Okada, H.: J. Bacteriol., 174, 7948 (1992).
15)Negoro, S., Shibata, N., Tanaka, Y., Yasuhira, K., Shibata, H., Hashimoto, H., Lee, Y.-H., Oshima, S., Santa, R., Oshima, S., Mochiji, K., Goto, Y., Ikegami, T., Nagai, K., Kato, D., Takeo, M., Higuchi, Y.: J. Biol. Chem., 287, 5079 (2012).
16) Fanuel, L., Goffin, C., Cheggour, A., Devreese, B., Driessche., G. Van., Joris, B., Beeumen, J. Van., Fre're, J.-M.: Biochem. J., 341, 147 (1999).
17) Geueke, B., Heck, T., Limbach, M., Nesatyy, V., Seebach, D., Kohler, H. P.: FEBS J., 273, 5261 (2006).
18) Elkins, J. M., Kershaw, N. J., Schofield, C. J.: Biochem. J., 385, 565 (2005).
19) Sankaranarayanan, R., Cherney, M. M., Garen, C., Garen, G., Niu, C., Yuan, M., James, M. N.: J. Mol. Biol., 397, 979 (2010).
20) Okada, T., Suzuki, H., Wada, K., Kumagai, H., Fukuyama, K.: Proc. Natl. Acad. Sci. USA, 103, 6471 (2006).
21) Marc, F., Weigel, P., Legrain, C., Glansdorff, N., Sakanyan, V.: J. Biol. Chem., 276, 25404 (2001).
22) Michalska, K., Hernandez-Santoyo, A., Jaskolski, M.: J. Biol. Chem., 283, 13388 (2008).