【トピックス】
バイナリーパターンデザインによるデノボタンパク質WA20のドメインスワップ二量体構造
小林直也、新井亮一
信州大・繊維
1.はじめに
デノボタンパク質 (de novo protein) とは、天然タンパク質のアミノ酸配列をもとにせず、新規にアミノ酸配列を設計した人工タンパク質である。タンパク質を自在にデザインし、望みの機能を実現することができるようになれば、医薬品開発や新規酵素の開発、さらにはナノバイオテクノロジーの発展等にも大きく貢献できると考えられ、タンパク質工学研究の究極的目標である。しかしながら、20種類のアミノ酸をランダムに100残基つなげる場合、20100=約1.3×10130通りもの莫大な配列組み合わせがあり、その中から安定な構造や優れた機能を持つタンパク質の配列を探し出すことは極めて困難である。デノボタンパク質の創製において、安定な構造や優れた機能を持つタンパク質の配列をいかにしてデザインし、選択するかは非常に重要であるが、現在でも大変困難な課題である。
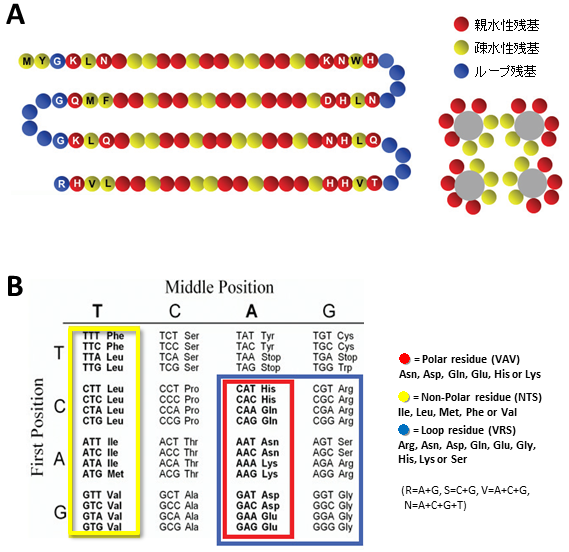
これまでに、デノボタンパク質の創製は、主に二種類の方法で行われてきた。一つは、立体化学的および物理化学的なアプローチにより合理的に設計する方法である1,2)。もう一つは、ランダムな組合せのアミノ酸配列ライブラリーから選択する方法である3)。さらに、プリンストン大学のMichael Hecht教授の研究室では、これらの合理的な方法とランダムな組合せの方法の両方の要素を取り入れたバイナリーパターン法によりデザインした配列ライブラリーを用いて、半合理的にデノボタンパク質を創製する研究が行われてきた4,5)。バイナリーパターン法とは、水溶性球状タンパク質の表面には親水性アミノ酸が多く、内部には疎水性アミノ酸が多く存在する性質に着目して、目的タンパク質の二次構造・三次構造に応じて、親水性アミノ酸 (Asn、Asp、Gln、Glu、His、Lys) と疎水性アミノ酸 (Ile、Leu、Met、Phe、Val) の繰り返し配列パターンをデザインする方法である (図1)。これまでに、バイナリーパターン法を用いてαへリックスの周期に従って3〜4残基ごとに親水性アミノ酸と疎水性アミノ酸のパターンをデザインした両親媒性αヘリックス構造4-6)や、親水性アミノ酸と疎水性アミノ酸のパターンを1残基毎に交互にデザインしたβストランド構造を持つデノボタンパク質の創製が報告されてきた7)。

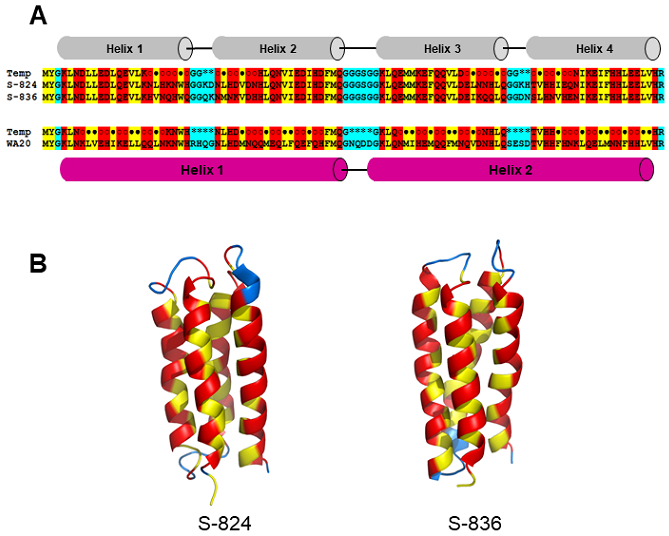
Hecht研究室では、このバイナリーパターン法を用いて、特に、両親媒性αヘリックス4本を順にループでつないで束とした4本ヘリックスバンドル構造のデノボタンパク質ライブラリーがこれまでに3世代作製されてきた。第1世代ライブラリーは、74残基のアミノ酸配列からなり、このライブラリーから得られたデノボタンパク質のいくつかは比較的安定な構造を形成したものの、多くは不安定な構造を有するものであった4)。そこで、より安定な構造を形成させるため、疎水性コアを形成するアミノ酸残基数を増加させた102残基のアミノ酸配列からなる第2世代ライブラリーが作製された6)。この第2世代ライブラリーから、4つの安定な構造をもつデノボタンパク質が得られた。そのうちS-824とS-836の2つにおいて、核磁気共鳴 (NMR) 法により立体構造が決定され、デザインした通りに、内側に疎水性アミノ酸、外側に親水性アミノ酸が配置された単量体4本ヘリックスバンドル構造であった (図2)8,9)。

さらに、機能を持つデノボタンパク質を創製するために、バイナリーパターンを保ちつつ、αヘリックスの中程の部分とループ部分のアミノ酸残基をライブラリー化した第3世代ライブラリーが作製された (図1)10,11)。このライブラリーから無作為に単離した高発現デノボタンパク質クローン群より、構造が特に安定で、天然タンパク質と同様に、変性剤によって二状態転移を示し、弱い酵素活性も有するデノボタンパク質WA20が得られた11)。そこで、筆者らは、WA20の構造特徴を詳細に解明することを目的として、X線結晶構造解析やX線小角散乱解析によるWA20の立体構造解析を行い、興味深い結果が得られたのでご紹介させて頂きたい12)。
2.WA20の結晶構造 「ドメインスワップ二量体構造」
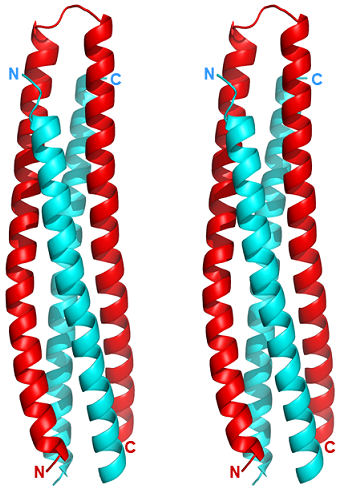
デノボタンパク質WA20は大腸菌を用いて発現、精製、結晶化し、X線結晶構造解析により2.2 Å分解能で立体構造を決定した12)。非常に意外だったことに、WA20の結晶構造は、ライブラリー設計の際に想定していたS-8248) やS-8369) (図2B) のような単量体の4本ヘリックスバンドル構造ではなく、二量体の4本ヘリックスバンドル構造であった (図3)。それぞれのWA20単量体は、40〜46残基の長いαヘリックス2本からなる“ヌンチャク”のような構造を取り、その構造が、もう一方の単量体の構造とお互いに挟み込むように絡み合ったドメインスワップ二量体構造、いわば“クロスヌンチャク型構造”を形成していた。(ドメインスワップとは、複数のタンパク質分子が、互いに部分的に絡み合うようにして、他の分子の構造の一部分をあたかも自らの分子の構造の一部分として取り込むように折り畳みながら、多量体形成することである。) WA20の全体の大きさ及び概形は、長さ約8 nm、直径は約3 nmの円筒状であった。

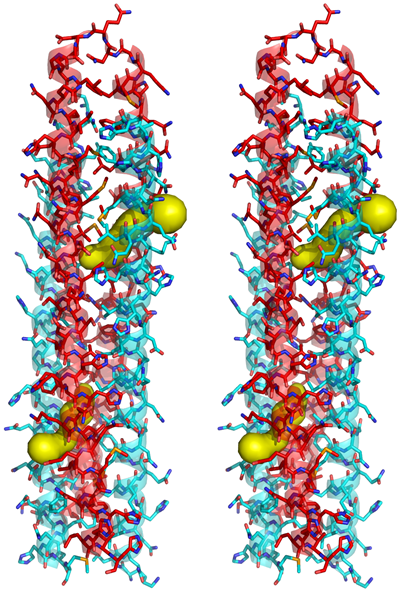
WA20の結晶構造では、バイナリーパターン法によるデザイン通りに、疎水性アミノ酸の側鎖 (黄色) は内側に向いており、親水性アミノ酸の側鎖 (赤色) は外側に向いていた (図4)。二量体形成の接触面は、疎水性クラスターのコアが形成されており (図5A)、二量体接触面は塩橋や水素結合により、さらに安定化していた (図5B)。WA20の熱安定性を測定したところ、変性温度Tmは約70 ℃と高かった12)。WA20の二量体構造では、単量体の4本ヘリックスバンドル構造の場合と比較して疎水性コア部分が約2倍程度増加しており、高い安定性の要因となっていると考えられる。
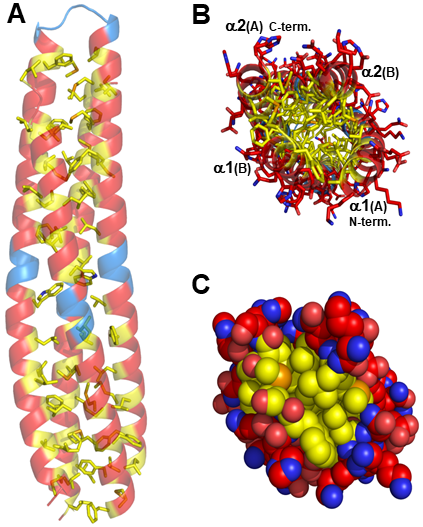

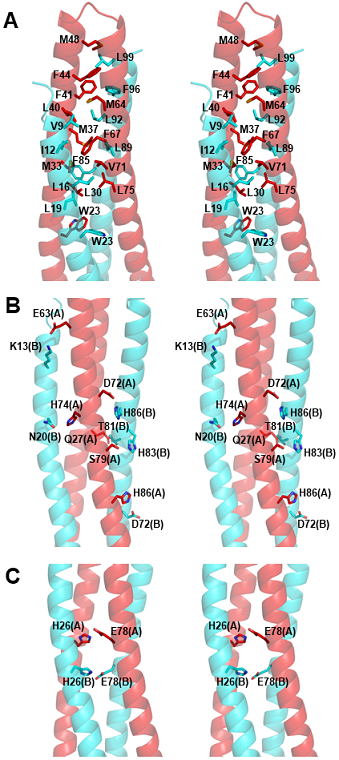

WA20の二量体形成の鍵となるアミノ酸残基を推定するために、WA20 (二量体) とS-824 (単量体) の構造およびアミノ酸配列を比較すると、25-28残基と77-80残基のループ設計領域において重要な違いが見られた (図2A)。S-824では、この領域のアミノ酸配列は-GGKD-と-GGKH-でグリシンが多く、ループ構造となっていた。一方、WA20ではこの領域のアミノ酸配列は-RHQG-と-SESD-であり、長いαヘリックスの中央付近に位置していた (図4A)。特に、His26とGlu78の側鎖間で塩橋を形成し、この領域の構造を安定化していると考えられる (図5C)。
3.X線小角散乱法によるWA20の溶液構造解析
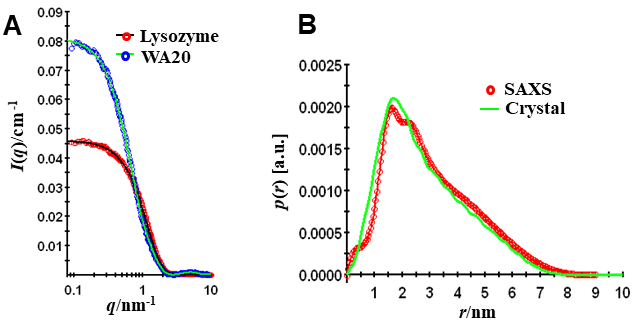
X線小角散乱法により、溶液中のWA20の構造解析を行った。図6AにWA20と卵白リゾチームの散乱強度を示す。リゾチームは、分子量の標準 (14.3 kDa) として用いた。前方散乱強度 I (q→0) は同濃度条件下において分子量に比例することより、WA20の溶液中の分子量は約25.3 kDaであると推定した。WA20単量体の理論分子量は12.5 kDaであることから、WA20は溶液中でも二量体を形成していると考えられる。続いて、間接フーリエ変換 (IFT) 法を用いて、WA20の二体間距離分布関数 p(r) を求めた (図6B)。p(r) には溶液中の分子形状が反映される。この関数 p(r) の横軸との切片の値Dmaxは、分子の最大長を表す。解析されたWA20のDmaxは約8 nmであり、WA20結晶構造の長さ約8 nmと一致する結果であった。また、p(r) の形状は、溶液中の分子概形が円筒状構造であることを示唆している。さらに、WA20の p(r)は、WA20の結晶構造からシミュレーションにより得られた p(r) とよく似ていた (図6B)。以上の結果より、WA20は溶液中においても二量体の4本ヘリックスバンドル構造を形成していると考えられる。

4.WA20の弱い酵素活性に関する考察
WA20は、ヘムと結合して紫外可視吸収スペクトルでソーレー帯のピーク(約410 nm)を示し、弱いペルオキシダーゼ活性を持つことが報告されている11)。ソーレー帯の吸収ピークは、WA20のHisやMet残基の側鎖が軸配位子としてヘムの鉄イオンに配位したことを示唆している。また、WA20がヘムと結合することにより、弱いペルオキシダーゼ活性部位が形成されることが推察される。WA20の配列には、HisやMet残基が比較的豊富に(WA20二量体の中にHis残基が26個、Met残基が16個)含まれており、WA20の立体構造から、ヘムの軸配位子となりうると予想されるアミノ酸残基ペアの候補はいくつか見つかったが、実際のヘムの結合部位を確かめるためには、さらなる実験が必要である。
また、天然の酵素と比較すると非常に低い活性ではあるが、WA20は、弱いエステラーゼ活性やリパーゼ活性を持つことも報告されている11)。WA20は、WA20非添加時と比較して、エステラーゼ活性では約400倍、リパーゼ活性では約500倍に反応速度を促進した。WA20のミカエリス・メンテン定数Kmは、天然酵素の値と同程度であり、代謝回転数kcatは天然酵素に比べて約10000分の1の値であった。そこで、エステラーゼ活性やリパーゼ活性の基質結合部位を推定するために、WA20結晶構造上のポケット部位を探索したところ、二つの比較的大きなポケット部位がA鎖とB鎖の間に見つかった (図7)。これらより、WA20には基質結合部位は存在するが、触媒残基は未だ適切でないということが推察される。今後、このようなポケット部位を持つWA20のドメインスワップ二量体構造をもとにして、分子進化工学的手法により触媒残基を最適化することにより、新規人工設計酵素開発のためのシンプルでユニークな基本骨格構造として利用できる可能性が期待される。
5.おわりに
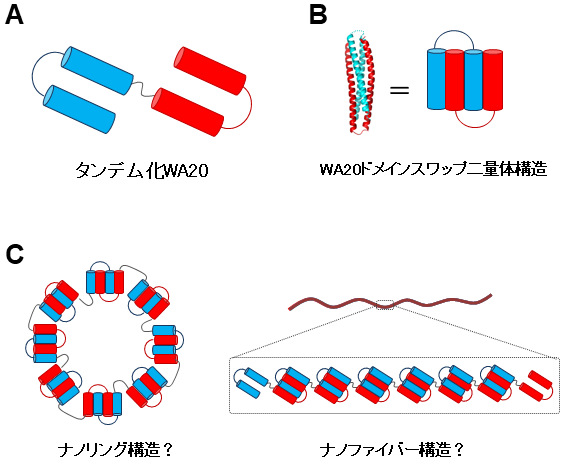
今後の展開として、WA20のユニークなドメインスワップ二量体構造を利用した応用の可能性も検討している。例えば、WA20を直列に2つ連結したタンデム化WA20を作製すれば、ドメインスワップ二量体構造を連続的に形成して、自己組織的に多量体ナノ構造 (図8) を構築できる可能性が考えられる。タンデム化WA20の構築の際、2つのWA20を連結するリンカーの部分が重要であると考えられるので、単独でαヘリックスを形成してドメイン間の距離を制御できるヘリックスリンカー (EAAAK)n13,14) や、フレキシブルリンカー (GGGGS)nのように、長さと性質の異なるリンカーを導入したタンデム化WA20タンパク質の作製を試みている。予備実験として、タンデム化WA20タンパク質を大腸菌で発現、精製して、電気泳動を行ってみたところ、変性条件のSDS-PAGEでは単一のバンドが観察されるのに対して、非変性条件のNative-PAGEでは、ラダー状にバンドが複数本観察された。このことは、タンデム化WA20が自己組織的に多量体形成し得ることを示唆していると考えられる。今後、さらにリンカー等を工夫したタンデム化WA20を作製し、他にも様々な自己組織的デノボタンパク質パーツを開発して、それらを自在に組み合わせることにより、多様な構造や機能を持つ自己組織的バイオナノ構造のデザイン・創製につながることが期待される。人工設計したデノボタンパク質を自己組織化するナノスケールのブロックパーツに見立てて、ナノ構造を創出するという研究は、未だ報告例は少ない。タンパク質がもつ特異的自己組織化能を活かした “バイオナノブロック” の創出へ向けて、デノボタンパク質研究の応用の裾野を広げていくことは、合成生物学分野やナノバイオテクノロジー分野の研究に新たな可能性を拓くことが期待される。

謝辞
本研究は、信州大学の佐藤高彰准教授、木村曉歩さん、松尾京子さんや、プリンストン大学のMichael H. Hecht教授、Luke H. Bradley博士、Anna F. Wangさん、Jesse M. Plattさんらとの共同研究の成果であり、心より感謝致します。また、X線回折実験は、高エネルギー加速器研究機構放射光科学研究施設PF構造生物ビームラインBL-5A, BL-6A, AR-W12Aにおいて行われました (課題番号2008G512, 2008G511, 2010G596)。本研究は、文部科学省テニュアトラック普及・定着事業、科学研究費補助金、日本学術振興会海外特別研究員制度等の支援を受けて行われました。この場を借りて厚く御礼申し上げます。
文献
1) Dahiyat, B. I., Mayo, S. L.: Science, 278, 82 (1997).
2) Kuhlman, B., Dantas, G., Ireton, G. C., Varani, G., Stoddard, B. L., Baker, D.: Science, 302, 1364 (2003).
3) Keefe, A. D., Szostak, J. W.: Nature, 410, 715 (2001).
4) Kamtekar, S., Schiffer, J. M., Xiong, H., Babik, J. M., Hecht, M. H.: Science, 262, 1680 (1993).
5) Hecht, M. H., Das, A., Go, A., Bradley, L. H., Wei, Y.: Protein Sci., 13, 1711 (2004).
6) Wei, Y., Liu, T., Sazinsky, S. L., Moffet, D. A., Pelczer, I., Hecht, M. H.: Protein Sci., 12, 92 (2003).
7) West, M. W., Wang, W., Patterson, J., Mancias, J. D., Beasley, J. R., Hecht, M. H.: Proc. Natl. Acad. Sci. USA, 96, 11211 (1999).
8) Wei, Y., Kim, S., Fela, D., Baum, J., Hecht, M. H.: Proc. Natl. Acad. Sci. USA, 100, 13270 (2003).
9) Go, A., Kim, S., Baum, J., Hecht, M. H.: Protein Sci., 17, 821 (2008).
10) Bradley, L. H., Kleiner, R. E., Wang, A. F., Hecht, M. H., Wood, D. W.: Protein Eng. Des. Sel., 18, 201 (2005).
11) Patel, S. C., Bradley, L. H., Jinadasa, S. P., Hecht, M. H.: Protein Sci., 18, 1388 (2009).
12) Arai, R., Kobayashi, N., Kimura, A., Sato, T.,
Matsuo, K., Wang, A. F., Platt, J. M., Bradley, L. H., Hecht, M. H.: J. Phys. Chem. B, 116, 6789 (2012).
13) Arai, R., Ueda, H., Kitayama, A., Kamiya, N., Nagamune, T.: Protein Eng., 14, 529 (2001).
14) Arai, R., Wriggers, W., Nishikawa, Y., Nagamune, T., Fujisawa, T.: Proteins, 57, 829 (2004).