【トピックス】
固体NMRによる膜タンパク質の構造と機能の解析について ― 最近の展開 ―
川村 出
横国大院・工
1.はじめに
細胞の恒常性を保つ上で、細胞膜中の膜タンパク質は物質輸送を行うことや細胞外からの情報や刺激を内部に伝達するなど多くの重要な役割をしている。また、ヒトのタンパク質の30%程度は膜タンパク質とされ、その中の多くは創薬のターゲットとされているなど、この分野に貢献するために膜タンパク質の構造情報の獲得は極めて重要である。一方でプロテインデータバンク (PDB) の膜タンパク質の論文発表済み構造登録数は830件程度であり、これは全登録数のおよそ1%程度であることからも、膜タンパク質の構造解析は極めて難しいことがわかる。近年、膜タンパク質の構造解析やその機能の理解に大きな寄与をもたらしている手法として固体NMR分光法がある。ここでは筆者が関わってきたロドプシンタンパク質の研究を含めた膜タンパク質の構造やその反応の直接観測に関する固体NMRの研究成果を紹介したい。
2.膜タンパク質の固体NMR構造解析
核酸、タンパク質などの生体高分子の3次元構造決定には、主にX線結晶構造解析や溶液NMRが用いられ、それぞれ結晶化および可溶化した試料が必要となるが、膜タンパク質の場合には細胞膜中に存在して機能するため、細胞膜中での構造を直接測定することが理想的である。固体NMRは試料の状態に依存せずにNMR信号を観測することができるため、細胞膜中の膜貫通型タンパク質、抗菌ペプチドや膜表在性タンパク質などへの研究が展開されている1-6)。これは膜タンパク質のような分子運動の少ない系において生じる異方的な相互作用 (化学シフト相互作用や磁気双極子相互作用) を平均化する高分解能技術 (ハイパワーデカップリングおよびマジックアングル回転法) や異方的な相互作用を巧みに利用するリカップリング技術が発展したためである。
3.アナベナセンサリーロドプシンの構造決定
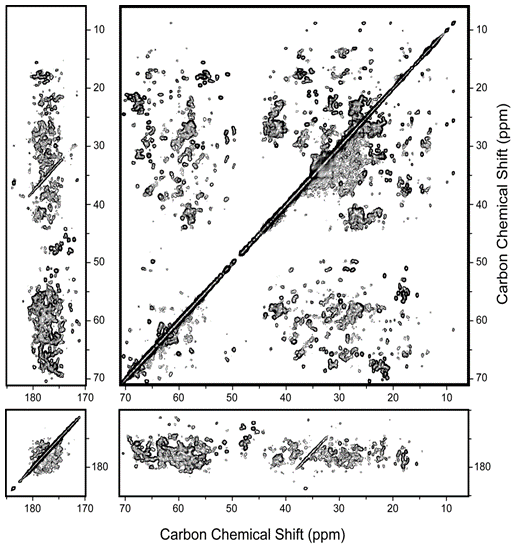
アナベナセンサリーロドプシンASRは真正細菌のシアノバクテリア中で発見された7回膜貫通型のレチナールタンパク質であり、ASRの細胞質側領域と水溶性のトランスデューサーASRTが相互作用して、光合成に必要な光捕集系タンパク質の発現制御に関与する光センサーと考えられている7)。タンパク質内部のレチナールの光異性化反応をトリガーとして光反応が進行する。このタンパク質の構造を決定するために、はじめに固体高分解能NMR測定による連鎖帰属を試みた。大腸菌による大量発現系を用いて均一13C, 15N標識したASRを発現、精製した後、DMPC/DMPA脂質二重膜に再構成し、固体NMR試料管に水和した試料をパッキングした。アミノ酸の指紋領域の分解能を評価するための13C-13C相関スペクトル (図1) から、線幅の狭い良く分離したNMR信号が得られ、レチナール結合部位であるシッフ塩基の15N NMR信号からASR中のレチナールはAll-trans型の配座のみをとっており、脂質膜中のASRの構造は極めて均一性の高いことがわかった。つづいて、3次元NMR測定を行い、アミノ酸残基間および残基内のスピン系を構築し、連鎖帰属を試みた8)。その結果、膜貫通領域においては約90%の信号帰属を達成し、BCループはβシート構造と示すことができた。

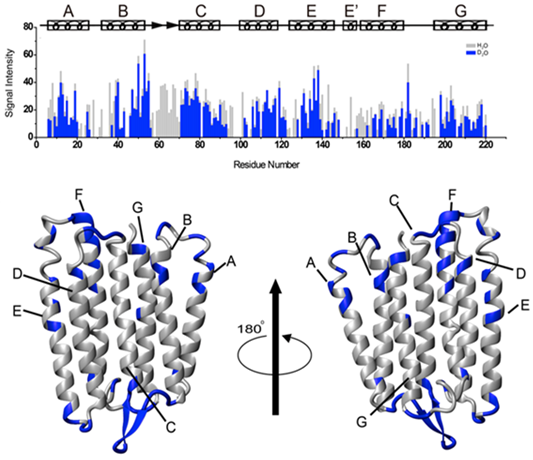
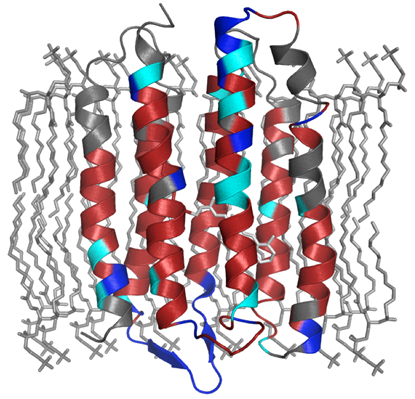
さらに暗条件で重水素交換の実験を行い、重水との交換により交差ピークの信号強度が変化することを利用して、ASRの脂質膜中でのトポロジーを評価した (図2)。その結果、タンパク質の細胞質側は細胞外側に比べてH/D交換が起きており、ASRの細胞質側は脂質膜からやや突出していることが示唆された8)。加えて重水溶媒中で試料に光照射を行い、レチナールの光異性化反応を進行させて、暗所での重水素交換速度と比較したところ、F, Gヘリックスの交換が細胞膜内まで進行し、光活性時にこれらの構造が変化していることも示唆された9)。これらの結果により図3のような細胞膜中での構造モデルを提唱するに至った。この後にタンパク質に常磁性タグを付したNMR測定と電気泳動などの生化学実験からASRは細胞膜中で三量体構造を形成していることが確認された。加えてNMR測定から得られた分子間および分子内の距離情報や化学シフト値から得られる二面角情報をもとにして、細胞膜中でのASRの三量体構造を決定した (PDB 2M3G)10)。すでに発表されているASRの結晶構造11)と比較するとBCループの構造情報やレチナール近傍のコンフォメーションを決定することができた。今後、細胞膜中での高分解能構造を利用し、光信号伝達機構を原子レベルで解明することにつながっていくと見られる。


4.固体NMRによる膜タンパク質の活性反応の直接観測
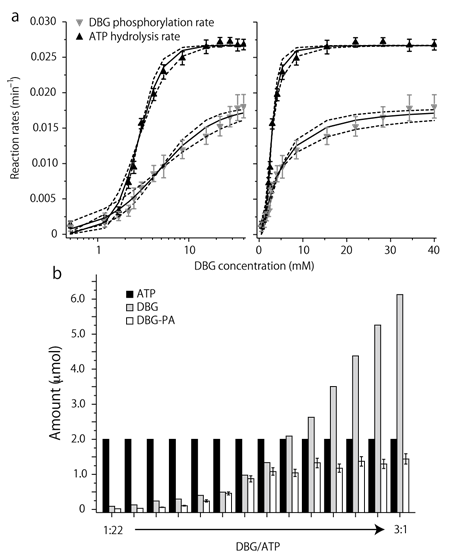
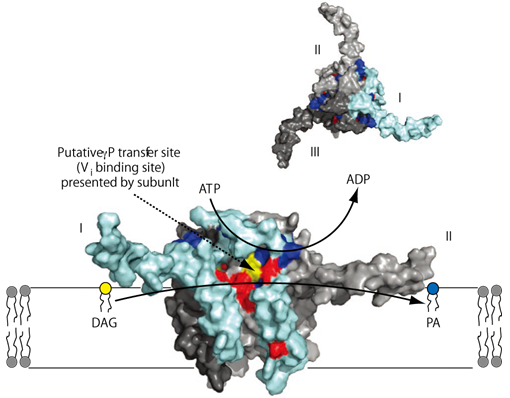
脂質制御の重要な役割を担う大腸菌の膜タンパク質ジアシルグリセロールキナーゼ (DGK) がある。DGKは溶解しているMgATPから膜結合しているジアシルグリセロール (DAG) へのγリン酸基の転移をおこなうことで、膜中にフォスファチジン酸 (PA) を産生させる反応を触媒する。フランクフルト ゴーシュ大学のC. Glaubitzらは時間分解31P固体NMR法によりATP加水分解反応とDAGのリン酸化反応の両方を同時かつ直接的に検出することに成功した12)。これはNMRの化学シフト値と信号強度によって二つの反応を評価することができるためである。ATPの加水分解反応およびジアシルブチリルグリセロールDBGのリン酸化反応の反応速度をDBG濃度に対してプロットしたグラフ (図4a) を示す。DGKのATP加水分解速度はDBG-PA産生速度よりも1.5倍速く、それぞれのシグモイダルな成長曲線をヒル式によってフィッティングした。ここからATP加水分解反応はDBGリン酸化反応に対して、正かつ強い協同性を示すことが示唆された。さらに反応初速度とKmの両逆数プロットによる解析からATPとDGBのDGKへの結合は独立に起きており、どちらも先行基質になりうることも示唆された。また、DBGを過剰に導入した図4bの実験から分かるようにDBG-PAを1分子産生するためにATPをおよそ1.5から2分子消費していることが示唆された。このような細胞膜と膜外の反応メカニズムを同時に評価することによって、膜中でホモトライマーを形成しているDGKの構造の中で、ATPγリン酸基のDAGへの転移反応が起きる予想結合部位を示すモデルを構築することができた (図5)。


また、我々は固体NMR測定中に試料に対して波長選択的に光を照射することができるIn-situ光照射-固体NMR法を開発し、細菌が有害な光に対して忌避応答 (負の走光性) を示すための光受容体フォボロドプシンに適用した13,14)。このタンパク質の発色団レチナールは特定の波長の光エネルギーを吸収してK中間体に励起される。このときに、レチナールはAll-trans型から13-cis,15-anti型に光異性化を起こす。その後、レチナールのシッフ塩基部位から脱プロトン化が起きることで他の中間体とは極大吸収波長が異なるM中間体に変化し、最終的には基底状態に戻るフォトサイクル反応を示す。この中で特にM中間体はフォボロドプシンからトランスデューサーに信号伝達される活性状態と考えられている。そのため我々は比較的長いライフタイムを持つM中間体を選択的に捕捉するために、低温で520 nmの光を用いることで固体NMR測定中にAll-trans型から13-cis型へのレチナール光異性化反応を起こし、フォボロドプシンのM中間体の観測に成功した。この時に得られたM中間体の信号は複数観測され、少なくとも2つ以上のM中間体の存在が示唆された。現在までに温度や光の波長を変化させた実験を行い、さらにM中間体の特徴を評価している15)。
これらの成果のように、細胞膜中の膜タンパク質のような複雑な系においても測定中に反応物と生成物を巧みに評価し、その反応について分子構造を基盤として深く 理解することができる。
謝辞
本研究はカナダ グエルフ大学のL. S. Brown教授、V. Ladizhansky教授および横浜国立大学の内藤 晶教授と共同で行った研究成果である。また、本研究の一部はテニュアトラック普及・定着事業 個人選抜型、科研費 基盤研究 (C) (24570127)、内藤記念科学振興財団、サントリー生命科学財団の支援を受けて遂行した。
文献
1)Cady, S. D., Schmidt-Rohr, K., Wang, J., Soto, C. S., Degrado, W. F., Hong, M.: Nature, 463, 689 (2010).
2)Park, S. H., Das, B. B., Casagrande, F., Tian, Y., Nothnagel, H. J., Chu, M., Kiefer, H., Maler, K., DeAngelis, A. A., Marassi, F. M., Opella, S. J.: Nature, 491, 779 (2012).
3)Naito, A.: Solid State Nucl. Magn. Reson., 36, 67 (2009).
4)Kawamura, I., Yamaguchi, S., Nishikawa, H.,Tajima, K., Horigome, M., Tuzi, S., Saito, H., Naito, A.: Polymer J., 44, 863 (2012).
5)Tsutsumi, A., Javkhlantugs, N., Kira, A., Umeyama, M., Kawamura, I., Nishimura, K., Ueda, K., Naito, A.: Biophys. J., 103, 1735 (2012).
6)Uekama, N., Aoki, T., Maruoka, T., Kurisu, S., Hatakeyama, A., Yamaguchi, S., Okada, M.,Yagisawa, H., Nishimura, K., Tuzi, S.: Biochim.Biophys. Acta, 1788, 2575 (2009).
7)Jung, K. H.: Photochem. Photobiol., 83, 63 (2007).
8)Shi, L., Kawamura, I., Jung, K. H., Brown, L. S., Ladizhansky, V.: Angew. Chem. Int. Ed., 50 1302 (2011).
9)Wang, S., Shi, L., Kawamura, I., Brown, L. S., Ladizhansky, V.: Biophys. J., 101, L23 (2011).
10)Wang, S., Munro, R. A., Shi, L., Kawamura, I., Okitsu, T., Wada, A., Kim, S. Y., Jung, K. H., Brown, L. S., Ladizhansky, V.: Nature Methods, 10, 1007 (2013).
11)Vogeley, L., Sineshchekov, O. A., Trivedi, V. D., Sasaki, J., Spudich, J. L., Luecke, H.: Science, 306,1390 (2004).
12)Ullrich, S. J., Hellmich, U. A., Ullrich, S., Glaubitz, C.: Nature Chem. Biol., 7, 263 (2011).
13)Kawamura, I., Kihara, N., Ohmine, M., Nishimura, K., Tuzi, S., Saito, H., Naito, A.: J. Am. Chem. Soc., 129,1016 (2007).
14)Tomonaga, Y., Hidaka, T., Kawamura, I., Nishio, T., Ohsawa, K., Okitsu, T., Wada, A., Sudo, Y., Kamo, N., Ramamoorthy, A., Naito, A.: Biophys. J., 101, L50 (2011).
15)Naito, A., Kawamura, I.: Adv. Biol. Solid StateNMR (RSC) Chap., 20, 387 (2014).