【トピックス】
ジャイアントリポソームを用いたin vitro進化分子工学
松浦友亮
阪大院・工・生命先端工
1.はじめに
近年、試験管内で生体分子を組み合わせ生命システムを再構築することが可能となってきた。in vitro合成生物学と呼ばれるこの分野では構築したシステムを用いることで、生命システムの動作原理の理解だけでなく、新規機能性分子の創出を目指している。本稿では、細胞サイズのジャイアントリポソーム内 (>1µm) で無細胞翻訳系を用いてタンパク質を合成する人工細胞の構築とこれを用いたタンパク質・酵素の進化分子工学を紹介する。進化分子工学は、ダーウィン進化を模倣し遺伝子変異導入と機能選択を繰り返す事により有用なタンパク質を創出する技術である。我々の開発したジャイアントリポソームを用いる手法は、唯一膜タンパク質を対象とできるin vitro進化分子工学的手法である。
2.in vitro進化分子工学
進化分子工学的手法は、変異と選択のステップを繰り返し行うことで生体高分子の性質を改良、進化させる方法である。本手法が使われ始めた1990年代の当初は有機溶媒中で働く酵素の創出、最近では製剤の大量生産、生体内のドーパミンを可視化、神経ガスを分解する酵素の創出などが報告されている。また、得られた多数の変異体の配列-機能相関解析などから、タンパク質の機能発現メカニズムの解明にも寄与してきた。このように本手法は、有用タンパク質・酵素の創出やその機能解析に大きく貢献してきた1,2)。
進化分子工学的手法は、タンパク質を合成する方法によってin vivo法とin vitro法の二つに分けられる。in vivo法はその名の通り細胞を使ってタンパク質を合成するのに対して、in vitro法では試験管内タンパク質合成系である無細胞翻訳系を用いる。in vitro法は、細胞を用いるin vivo法と比べて、形質転換のステップがないため、より多様性の大きい遺伝子ライブラリーを取り扱える、細胞増殖を待つ必要がないために通常1日で変異と選択のステップが行えるなどの利点がある3,4)。
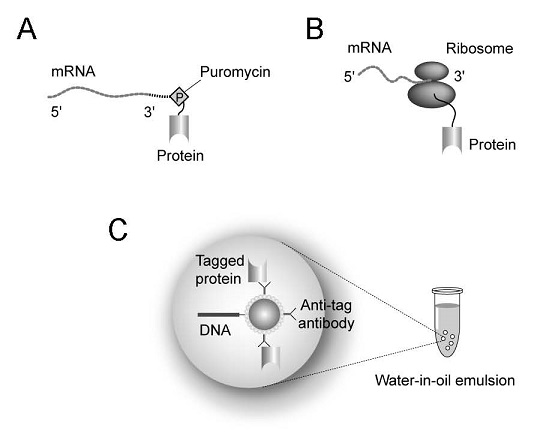
in vitro法には、mRNA display、ribosome display、in vitro compartmentalization (IVC)などがあり、高機能・高活性の抗体、抗体様分子、酵素の創出に用いられてきた。これらに共通する性質は無細胞翻訳系を用いて遺伝子型と表現型を物理的に対応させている点にある (図1) 。すなわち、図1に示したようにいずれの方法でも1分子の遺伝子とこれがコードするタンパク質・酵素が対応付けされている。次節では筆者らが近年開発したジャイアントリポソームを用いて遺伝子型と表現型を対応付けしているin vitro進化分子工学的手法について解説する5,6)。

3.リポソームを用いたin vitro進化分子工学
従来のin vitro進化工学手法は、抗体、抗体様分子、酵素などの可溶性タンパク質を対象としており、膜タンパク質を対象とした手法は開発されていなかった。膜タンパク質はゲノム上にコードされるタンパク質の3割、製剤のターゲットの5割を占めるといわれている。有用な膜タンパク質の創出や機能解析を進めるためにも膜タンパク質進化工学手法の開発が待たれていた。我々が近年開発したジャイアントリポソームを用いたin vitro進化分子工学的手法がこれをはじめて可能とした。
約10年前にアメリカのグループが無細胞翻訳系を封入した細胞サイズのジャイアントリポソームを作製し、内部でタンパク質を合成する‘人工細胞’を構築する技術を報告した7)。我々はこの技術を発展させることで膜タンパク質の進化分子工学を達成することを考えた (図2) 5,6)。
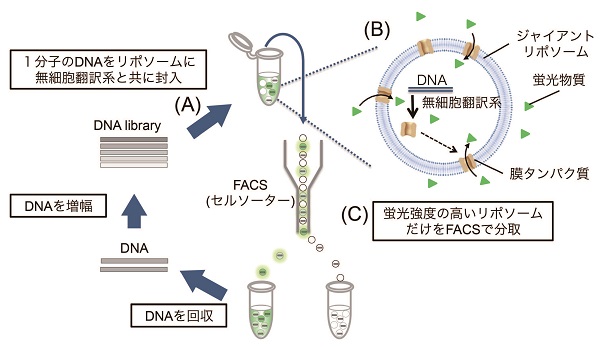

まず、スクリーニングの対象とする膜タンパク質をコードするDNAライブラリーを用意する (A) 。本手法で取り扱えるDNAライブラリーの多様性の大きさは107程度である。次に、各リポソームにDNAを約1分子となるように無細胞翻訳系と共に封入し、リポソーム内部でタンパク質合成反応を行うと合成された膜タンパク質は膜に挿入される (B) 。膜タンパク質の活性が蛍光シグナルに変換されるような仕組みをリポソーム内部に組み込んでおく。これにより高い蛍光強度をもつリポソームには高い活性をもつ膜タンパク質とそれをコードする遺伝子が含まれていることになる。蛍光強度の高いリポソームを蛍光フローサイトメーター (FACS) で分取するとDNAライブラリーから高機能型の膜タンパク質がコードされる分子が濃縮されることになる (C) 。本手法は、合成されるタンパク質が膜タンパク質でなく酵素であって、酵素活性を蛍光シグナルに変換する仕組みがリポソーム内に組み込まれていれば、酵素の進化分子工学にも適用できる。
上記のようにジャイアントリポソームの膜上に膜タンパク質をディスプレイできることから上記手法をリポソームディスプレイ法 (liposome display) と名付けた。リポソームディスプレイ法では、ジャイアントリポソーム内で1分子のDNAを鋳型としてタンパク質合成を行うことで遺伝子型と表現型を物理的に対応させている。本手法を確立するためには、適した無細胞翻訳系の開発8,9)、リポソーム調製方法の開発10)、リポソームのFACSによる計測技術の確立11)など多岐にわたる技術開発が必要であった。これらの詳細は本稿の主旨とは異なるので記載しない。興味をお持ちの方は方法論だけを詳細に記載した参考文献を参照いただきたい5)。次節でリポソームを用いたin vitro進化分子工学の応用例を2つ示す。
4.膜タンパク進化・酵素進化への応用
αヘモリシンは黄色ブドウ球菌由来のpore形成膜タンパク質であり12)、ナノポアシーケンスなど様々な応用展開が期待されている重要な分子である13,14)。一方で、進化分子工学を用いた工学利用に適した分子の開発は行われていない。そこで我々は人工脂質二重膜でより高い活性を発現するαヘモリシンをリポソームディスプレイ法を用いて作り出すことを目指した。
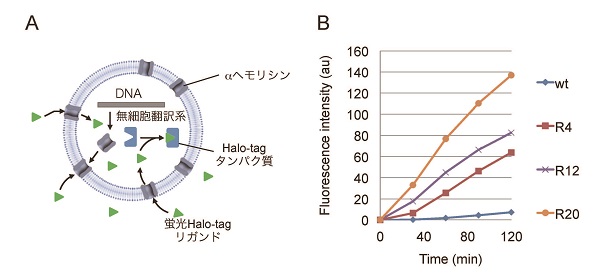
まず、ランダム変異を導入したαヘモリシン遺伝子ライブラリーを調製し、リポソーム内に遺伝子が1分子ずつ入るように封入後、タンパク質合成を行った。ここでαヘモリシンの活性を蛍光シグナルに変換する仕組みが必要である。我々は、これを達成するために蛍光Halo-tagリガンドとHalo-tagタンパク質15)を用いた (図3A) 。蛍光Halo-tagリガンドは負電荷をもつため、通常のリン脂質二重膜は透過しないが、αヘモリシンのporeが形成されると膜を通過する。この蛍光基質をリポソーム外部に加えると、poreの数が多いほど多くの蛍光基質が内部に流入する。流入した蛍光基質は、リポソーム内部に封入しておいたHalo-tagタンパク質と共有結合を形成しトラップされる。このようにして、それぞれの変異体遺伝子から合成されるαヘモリシンのpore形成活性を蛍光度に変換する仕組みをリポソーム内に組み込んだ。そこで、図2に示すように蛍光強度の高いリポソームをセルソーターにより分取し、遺伝子を増幅、再びリポソームに封入して同様の選択実験を繰り返した。その結果、選択実験を繰り返すに従って、遺伝子プールの活性は向上していった (図3B) 。最終的には、人工脂質二重膜において野生型のヘモリシンよりも約30倍活性の高いヘモリシン変異体の取得に成功した。機能向上の分子的なメカニズムについてはオリジナル論文を参照いただきたい5,6)。

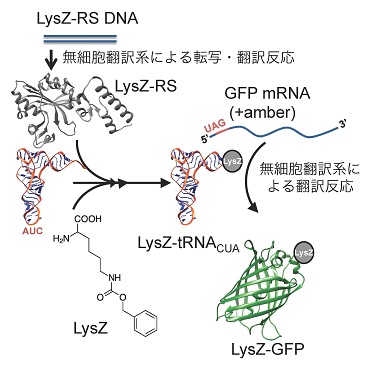
前節で述べたように、リポソームディスプレイ法では膜タンパク質だけでなく、酵素の進化工学も可能である。ここでは非天然アミノ酸をtRNAにチャージできる高機能型アミノアシル化tRNA合成酵素 (ARS) の創出について紹介する16)。LysZ-RSは、理化学研究所の柳沢らにより開発された有用非天然アミノ酸N-benzyloxy carbonyl-L-lysine (LysZ) を認識するARS変異体 (LysZ-RS) である17,18)。一方で、LysZ-RSの活性は天然のARSと比べると低く高機能化が望まれていた。
LysZ-RSの進化分子工学のためには、まずリポソーム内でLysZ-RSの活性を蛍光シグナルに変換する仕組みが必要である。我々はこれを図4に示す方法で達成した。具体的には、LysZ-RSの活性により、LysZをチャージしたtRNAが生成し、これがGFP遺伝子上流に存在する終始コドン (amber) の位置にLysZを導入する。これにより、GFPが生成しリポソームが蛍光シグナルを持つ。一方で、LysZ-RSの活性がない場合はGFPが合成されずリポソームは蛍光を持たない。このようにLysZ-RS活性とGFP合成活性が比例する反応系を構築した。リポソームディスプレイ法は、in vitroのシステムであるために比較的自由に目的の反応系を組み上げることができる (図4) 。

次に、ヘモリシンと同様にLysZ-RS遺伝子にランダム変異を導入した遺伝子ライブラリーを作製し、蛍光強度の高いリポソームをFACSで分取しLysZ-RS変異体の遺伝子を回収した。この選択プロセスを3ラウンド行った結果、無細胞翻訳系だけでなく大腸菌においてもオリジナルのLysZ-RSと比べて高活性な変異体の取得に成功した16)。ただし現時点では1.7倍程度の活性向上のためさらなる向上を目指しているところである。
5.おわりに
これまで酵素や可溶性タンパク質を標的分子とする進化分子工学的手法は多く開発され、様々な変異体が作られてきた。それは酵素の市場規模の大きさに依存するところが大きいと考えられる (市場規模は酵素自身が4000億円、酵素を用いた生成物も考慮すると10兆円以上 (天野エンザイムホームページより引用)) 。現在、膜タンパク質を用いた産業はほとんど存在しないが、リポソームディスプレイ法を用いることで工学利用可能な膜タンパク質の創成が可能となることから今後新しい産業分野の創成を期待しているところである。
謝辞
本文で紹介した研究は、大阪大学大学院工学研究科の渡邉肇教授、大阪大学大学院情報科学研究科の四方哲也教授の研究室で行われたものです。また、LysZ-RSの研究は北陸先端科学技術大学院大学の芳坂貴弘教授との共同研究で行われたものです。諸先生方、研究員、学生の皆様に深く感謝します。特にヘモリシン関連の全ての研究に携わられた元ERATOプロジェクト博士研究員の藤井聡志博士に感謝します。
文献
1) Tracewell, C. A., Arnold, F. H.: Curr Opin Chem Biol, 13, 3 (2009).
2) Bornscheuer, U. T., Huisman, G. W., Kazlauskas, R. J., Lutz, S., Moore, J. C., Robins, K.: Nature, 485, 185 (2012).
3) Lane, M. D., Seelig, B.: Curr Opin Chem Biol, 22, 129 (2014).
4) Matsuura, T., Yomo, T.: J Biosci Bioeng, 101, 449 (2006).
5) Fujii, S., Matsuura, T., Sunami, T., Nishikawa, T., Kazuta, Y., Yomo, T.: Nat Protoc, 9, 1578 (2014).
6) Fujii, S., Matsuura, T., Sunami, T., Kazuta, Y., Yomo, T.: Proc Natl Acad Sci USA, 110, 16796 (2013).
7) Noireaux, V., Libchaber, A.: Proc. Natl. Acad. Sci. USA, 101, 17669 (2004).
8) Kazuta, Y., Matsuura, T., Ichihashi, N., Yomo, T.: J Biosci Bioeng, 118, 554 (2014).
9) Matsuura, T., Kazuta, Y., Aita, T., Adachi, J., Yomo, T.: Mol Syst Biol, 5, 297 (2009).
10) Nishimura, K., Matsuura, T., Nishimura, K., Sunami, T., Suzuki, H., Yomo, T.: Langmuir, 28, 8426 (2012).
11) Nishimura, K., Hosoi, T., Sunami, T., Toyota, T., Fujinami, M., Oguma, K., Matsuura, T., Suzuki, H., Yomo, T.: Langmuir, 25, 10439 (2009).
12) Song, L., Hobaugh, M. R., Shustak, C., Cheley, S., Bayley, H., Gouaux, J. E.: Science, 274, 1859 (1996).
13) Branton, D., Deamer, D. W., Marziali, A., Bayley, H., Benner, S. A., Butler, T., Di Ventra, M., Garaj, S., Hibbs, A., Huang, X., Jovanovich, S. B., Krstic, P. S., Lindsay, S., Ling, X. S., Mastrangelo, C. H., Meller, A., Oliver, J. S., Pershin, Y. V., Ramsey, J. M., Riehn, R., Soni, G. V., Tabard-Cossa, V., Wanunu, M., Wiggin, M., Schloss, J. A.: Nat Biotechnol, 26, 1146 (2008).
14) Astier, Y., Bayley, H., Howorka, S.: Curr Opin Chem Biol, 9, 576 (2005).
15) Los, G. V., Encell, L. P., McDougall, M. G., Hartzell, D. D., Karassina, N., Zimprich, C., Wood, M. G., Learish, R., Ohana, R. F., Urh, M., Simpson, D., Mendez, J., Zimmerman, K., Otto, P., Vidugiris, G., Zhu, J., Darzins, A., Klaubert, D. H., Bulleit, R. F., Wood, K. V.: ACS Chem Biol, 3, 373 (2008).
16) Uyeda, A., Watanabe, T., Kato, Y., Watanabe, H., Yomo, T., Hohsaka, T., Matsuura, T.: Chembiochem, 16, 1797 (2015).
17) Yanagisawa, T., Ishii, R., Fukunaga, R., Kobayashi, T., Sakamoto, K., Yokoyama, S.: Chem Biol, 15, 1187 (2008).
18) Yanagisawa, T., Hino, N., Iraha, F., Mukai, T., Sakamoto, K., Yokoyama, S.: Mol Biosyst, 8, 1131 (2012).