【トピックス】
プロテインフォールディングメモリーによる酵素タンパク質の改変
里村 淳、黒田浩一、植田充美
京大院・農/日本学術振興会特別研究員、京大院・農、京大院・農
1.はじめに
タンパク質の折りたたみ (フォールディング) は、「折り紙」によく例えられる。折り紙で、一枚の紙が折りたたまれて意味のある形になるように、タンパク質では、数珠つなぎになったアミノ酸から意味のある形をもち、機能を有する酵素タンパク質などが折られていく。一方で、折り紙とタンパク質のフォールディングには相違点もある。折り紙では、何の変哲もない一枚の紙から紙飛行機や鶴が折られていく。何を折るのかを決めるのは折る人であり、折り紙は何に折られるかを決めることができない。タンパク質のフォールディングではその反対で、何に折られるのかを決めるのはタンパク質自身で、他の因子はその運命を決定することはできないと考えられてきた。例えば、プロテアーゼならばプロテアーゼ用のアミノ酸配列をもち、リパーゼならばリパーゼ用のアミノ酸配列もつ。誤ってリパーゼがプロテアーゼに折られることはない。このように、タンパク質のフォールディングはそのアミノ酸配列によって一義的に決定されると考えられてきた。このタンパク質のフォールディングに関する基本的な考え方は「アンフィンセンのドグマ」と呼ばれている1)。これを提唱したアンフィンセンは1972年にノーベル化学賞を受賞している。
本稿では、プロペプチドの配列を改変することで起こる、アンフィンセンのドグマに反した「プロテインフォールディングメモリー」という現象を紹介する。プロペプチドとは、プロテアーゼやリパーゼの前駆体タンパク質に見られる短いペプチドであり2,3)、成熟領域と相互作用することで、自身の構造をインプリントした (鋳型とした) 後、切断除去される。このように、プロペプチドは成熟体と直接結合した状態で生産されて、成熟体のフォールディングを促進することから、しばしば「分子内シャペロン」と呼ばれている4,5)。我々は、ペプチダーゼやリパーゼのプロペプチドに変異を導入すると、プロペプチドのフォールディング作用が変化して、成熟酵素の安定性と基質特異性が改変されることを明らかにしてきた (図1)6,7)。変異プロペプチドは元の野生型プロペプチドと同様に、成熟酵素から切断除去されるため、完成した酵素タンパク質のアミノ酸配列は変化しない。本研究は、タンパク質はそのアミノ酸配列の情報によって、常に一定の構造に折りたたまれるわけではなく、折り紙が様々な形に折られるように、同じタンパク質でも相互作用する因子の働きによって、様々な形に折られることを証明した。ここでは、このプロテインフォールディングメモリーという現象について、Carboxypeptidase Y (CPY)6)とRhizopus oryzae lipase (ROL)7)の二例を挙げて紹介する。
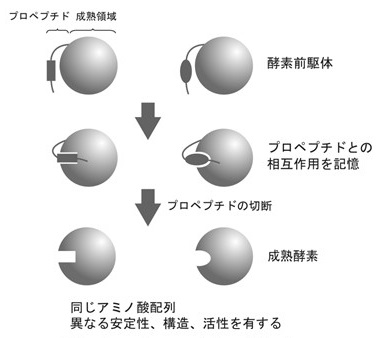
図1 プロテインフォールディングメモリー
2.Carboxypeptidase Y (CPY)
CPYは、酵母Saccharomyces cerevisiae に見られる、N末端に91アミノ酸残基のプロペプチドをもつペプチダーゼである8)。プロペプチドは、CPY成熟領域のフォールディングを補助した後に、最終的にプロテイナーゼBという酵母の内在タンパク質により切断除去される5)。酵母には、CPYのインヒビターペプチド (IC) も存在する9)。ICとCPYプロペプチドは部分的に相同なアミノ酸配列を有するため、両者は共に成熟CPYと相互作用することができる10)。しかし、CPYインヒビターであるICと分子内シャペロンであるCPYプロペプチドでは働きが異なるために、成熟CPYとの相互作用様式は同一ではないと予想された。そこで、我々はCPYプロペプチドの一部の配列をICの配列と交換することで、成熟領域のフォールディングに与える影響を調べることにした。
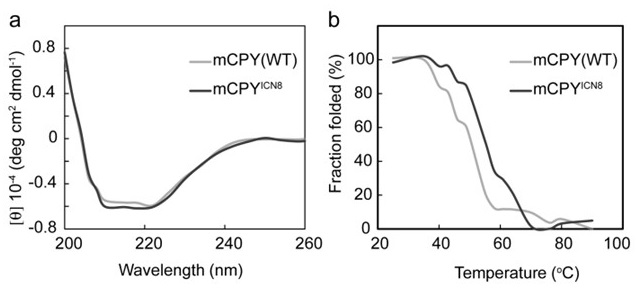
配列を入れ替えたプロペプチドによって折りたたまれた成熟CPY(mCPYICN8) と、野生型のプロペプチドによって折りたたまれた成熟CPY(mCPY(WT)) はどちらも同じ位置でプロペプチドの切断を受けており、両者のアミノ酸配列は同一であった。しかし、mCPYICN8はmCPY(WT) よりも高いペプチダーゼ活性を有していた。さらに、mCPYICN8の酵素活性を詳細に調べるために、速度論解析を行った (表1)。mCPYICN8は、mCPY(WT) と同様の基質親和性 (KM) を示す一方、mCPY(WT) よりも高い分子触媒活性 (kcat) を示した。また、酵素の活性化エネルギーをアレニウスの式に従って決定したところ、mCPYICN8はmCPY(WT) よりも小さい活性化エネルギーを示した。これより、mCPYICN8では、反応の活性化エネルギーが低下して、分子活性が上昇していることが示唆された。最後に、CDスペクトルにより両者の二次構造の違いと熱力学的安定性を調べた。CDスペクトルの感度では、mCPYICN8とmCPY(WT) の二次構造の違いは見られなかったが (図2a)、熱力学的安定性はmCPYICN8で向上していた (図2b)。配列を交換したプロペプチドは成熟領域の局所的な構造を変化させることで、その活性や熱力学的安定性を向上させていることが予想された。
表1 mCPY(WT)とmCPYICN8の速度論解析


3.Rhizopus oryzae lipase (ROL)
ROLは、69アミノ酸残基のプロペプチドを有するリパーゼであり、天然ではRhizopus oryzaeという接合菌に見いだされる。ROLのプロペプチドもCPYのプロペプチドと同様に「分子内シャペロン」活性を有している11)。先の研究で、我々はROLのプロペプチドのうち、特にSer38からGlu57のアミノ酸残基がROLの成熟化に関わっていることを明らかにしてきた12)。また、この領域は近縁種のリパーゼとも相同性が高く、成熟酵素のフォールディングに重要な役割を果たしていることがうかがえた7)。さらに、この部位には電荷を有するアミノ酸や親水性アミノ酸が多く存在することから、成熟部位との静電相互作用によりフォールディングを促進していると考えられた。そこで、我々は特に電荷を有するアミノ酸を多く含むGln50からTrp55の領域を改変することで、同じアミノ酸配列であるが、活性の異なる成熟リパーゼの取得を試みた。
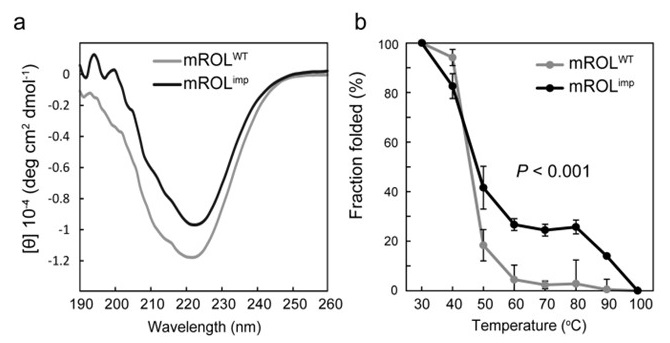
この6アミノ酸領域をより親水性の高い6アミノ酸に交換したところ、プロテインフォールディングメモリーが観察された。この変異プロペプチドによって折りたたまれたROL(mROLimp) は、WTプロペプチドによって折りたたまれたROL(mROLWT) と比べて、基質の炭素鎖長に関わらず、いずれの基質においても高い活性を示した (図3a)。さらに、mROLimpはmROLWTと比べて、中鎖基質に対する分解性が向上していた (図3b)。なお、エドマン分解法により成熟体のN末端アミノ酸配列を解析したところ、このプロペプチド変異ROLは、WTと同じ位置でプロペプチドの切断を受けていることを確認している。さらに、二つのリパーゼを用いて速度論解析を行ったところ、mROLimpはmROLWTよりも高い分子触媒活性 (kcat) を示し、活性化エネルギーが低下していた (表2)。CDスペクトルにより二次構造を調べたところ、CPYの場合と異なりmROLimpでは二次構造の差異が確認できた (図4a)。つまり、mROLWTとmROLimpは同一のアミノ酸配列でありながら、異なる立体構造を有していることになる。これらの現象は、成熟ROLが変異したプロペプチドとの相互作用を「記憶 (メモリー) 」することでもたらされる。プロペプチドに対する僅か6アミノ酸残基の改変が、成熟領域に記憶され、成熟酵素の活性や構造に大きな変化をもたらしたことは特筆に値する。さらに、mROLimpの熱力学的安定性も、mROLWTより向上していることがわかった (図4b)。これは天然に存在するタンパク質が必ずしも最も安定な状態にあるわけではなく、相互作用する因子の影響によって、さらに安定な状態にフォールディングされうることを示している。
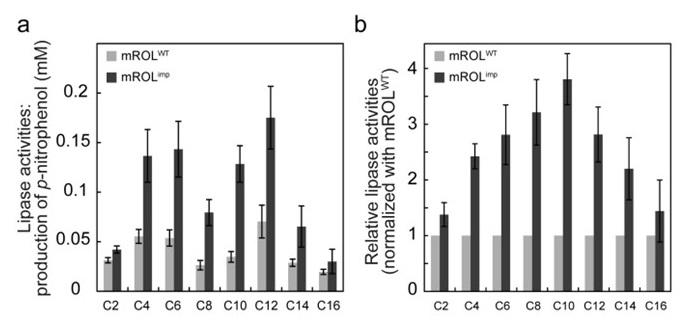

表2 mROLWTとmROLimpの速度論解析


最後に、mROLimpの基質特異性が変化していたことより、さらに異なる基質を認識するかどうかを検討した。ROLは、活性中心にAsp、His、Serをもつ。この活性中心は多くのセリンプロテアーゼやセリンペプチダーゼにも見られる13)。セリンプロテアーゼ・ペプチダーゼはタイプIからタイプIIIに分類され、ROLの活性中心はタイプIIIのセリンプロテアーゼ・ペプチダーゼのものに酷似している。そこで、我々は、mROLimpがタイプIIIのセリンペプチダーゼに属するCPYに特異的な基質を認識できることを競合アッセイにより検証した。競合アッセイでは、CPYの基質であるSuc-Ile-Ile-Trp-MCAをリパーゼの基質であるp-nitrophenyl butyrateと混合した状態でリパーゼ活性測定を行った。ROLがCPY基質を認識すると競合阻害を受けて、リパーゼ活性が低下する。すなわち、リパーゼ活性を測定することでCPY基質との結合を評価できる。mROLWTではCPY基質存在下でも、リパーゼ活性は低下しなかったのに対し、mROLimpでは、CPY基質存在下においてリパーゼ活性が低下していた (図5)。したがって、mROLimpはCPY基質を認識することが判明した。
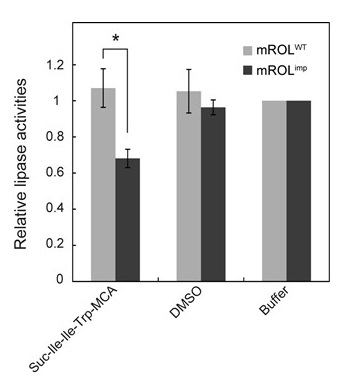

mROLimpにおける基質特異性の変化およびペプチダーゼ基質認識能の獲得は、変異プロペプチドに起因する活性中心の立体配座変化によってもたらされたと考えられる。ROLに近縁のリパーゼでは、活性中心の近くに位置する「Lid domain」と呼ばれる疎水性の高いドメインが基質認識に関わっていることが知られている14)。ROLにおいても、この「Lid domain」に変異を導入すると、基質特異性が変化することから、基質認識に重要な役割を担っていると予想される15)。我々は、変異プロペプチドによって、特に、「Lid domain」の位置や構造が変化して、疎水性の高い中鎖基質への特異性向上や、イソロイシンなど疎水性アミノ酸を含むCPY基質に対する認識能がmROLimpにもたらされたと推察している。しかし、変異プロペプチドと成熟領域との相互作用様式や、mROLimpの立体構造は未解明であり、今後の課題である。
4.おわりに
本研究において、タンパク質の立体構造はそのアミノ酸配列のみによって決まるわけではなく、プロペプチドなどの相互作用する因子によって変動しうることを示した。アンフィンセンのドグマ以降、天然に存在するタンパク質の立体構造は最も安定な状態にあると考えられてきたが、本研究では、プロペプチドの働きなどによりフォールディング経路が変化すると、さらに安定な状態にまで至ることが明らかになった16)。これまでにも、特定のタンパク質のアミノ酸を同義置換によって頻出コドンからレアコドンに変えると、局所的な翻訳速度が減少することで、フォールディング経路が変化するという現象が報告されている17)。フォールディング経路が変化することで、同じアミノ酸配列であるが構造や阻害剤感受性が異なるタンパク質が作られる。また、プリオンタンパク質のように、同じアミノ酸配列であっても、正常型と異常型をとるタンパク質も知られている18)。これらの現象は全てタンパク質の立体構造はアミノ酸配列のみで決定されるわけではなく、経過するフォールディング経路によって、生産されるタンパク質の構造や安定性が変化することを示している。現在のところ、プロテインフォールディングメモリーによって、タンパク質の立体構造がどのように変化していくかは分かっていない。しかし、これらを解明することで、タンパク質のフォールディングの真髄とともに、プリオンをはじめとするフォールディング病の発症機構などを明らかにできると期待している。
文献
1) Anfinsen, C. B.: Biochem. J., 128, 737 (1972).
2) Inouye, M.: Enzyme, 45, 314 (1990).
3) Takahashi, S., Ueda, M., Atomi, H., Beer, H. D., Bornscheuer, U. T., Schmid, R. D., Tanaka, A.: J. Ferment. Bioeng., 86, 164 (1998).
4) Takahashi, S., Ueda, M., Tanaka, A.: Appl. Microbiol. Biotechnol., 52, 534 (1999).
5) Susanne, O. S., Van Den Hazel, M. C., Kielland-Brandt, M. C., Winther, J. R.: Eur. J. Biochem., 220, 19 (1994).
6) Nagayama, M., Maeda, H., Kuroda, K., Ueda, M.: Biochemistry, 51, 3547 (2012).
7) Satomura, A., Kuroda, K., Ueda, M.: PLOS ONE, 10 (2015).
8) Winther, J. R., Sorensen, P.: Proc. Natl. Acad. Sci. USA, 88, 9330 (1991).
9) Mima, J., Suzuki, H., Takahashi, M., Hayashi, R.: J. Biochem., 132, 967 (2002).
10) Nagayama, M., Kuroda, K., Ueda, M.: Biosci. Biotechnol. Biochem., 76, 153 (2012).
11) Haas, M. J., Bailey, D. G., Baker, W., Berka, T. R., Cichowicz, D. J., Derewenda, Z. S., Genuario, R. R., Joerger, R. D., Klein, R. R., Scott, K.: Eur. J. Lipid Sci. Tech., 101, 364 (1999).
12) Takahashi, S., Ueda, M., Tanaka, A.: Appl. Microbiol. Biotechnol., 55, 454 (2001).
13) Shibamoto, H., Matsumoto, T., Fukuda, H., Kondo, A.: J. Mol. Catal. B: Enzym., 28, 235 (2004).
14) Brzozowski, A. M., Derewenda, U., Derewenda, Z. S., Dodson, G. G., Lawson, D. M., Turkenburg, J. P., Bjorkling, F., Huge-Jensen, B., Patkar, S. A., Thim, L.: Nature, 351, 491 (1991).
15) Shiraga, S., Ishiguro, M., Fukami, H., Nakao, M., Ueda, M.: Appl. Microbiol. Biotechnol., 68, 779 (2005).
16) http://www.kyoto-u.ac.jp/ja/research/research_results/2015/150514_2.html.
17) Kimchi-Sarfaty, C., Oh, J. M., Kim, I. W., Sauna, Z. E., Calcagno, A. M., Ambudkar, S. V., Gottesman, M. M.: Science, 315, 525 (2007).
18) Zou, W., Gambetti, P.: Cell. Mol. Life Sci., 64, 3266 (2007).