【トピックス】
超好熱性アーキアThermococcus kodakarensis由来分岐鎖ポリアミン合成酵素の構造と機能
秀瀬涼太、藤原伸介
関学大院・理工、関学大院・理工・生物機能基材研究開発セ
1.はじめに
ポリアミンは二つ以上のアミノ基を含む脂肪族炭化水素の総称であり、ウイルスからヒトに至るまであらゆる生物に含まれる。ポリアミンは細胞の分化や増殖などに必須であり、熱など様々なストレスに対する適応においても重要である1,2)。プトレスシン[4]、スペルミジン[34] (鍵括弧内の数字は、両端にあるアミノ基と中間のアミノ基で挟まれるメチレン基の数を示す。) はほとんど全ての生物にみられる共通のポリアミンである。一方、55℃以上を生育至適温度にもつ生物である (超) 好熱菌は、カルドペンタミン [3333]、カルドヘキサミン [33333]などに代表される長鎖ポリアミンや、N4-アミノプロピルノルスペルミジン [3(3)3]やN4-アミノプロピルスペルミジン[3(3)4]、N4-ビス (アミノプロピル) ノルスペルミジン[3(3)(3)3]、N4-ビス (アミノプロピル) スペルミジン[3(3)(3)4]などに代表される分岐鎖ポリアミンなど常温生物には見られない特殊なポリアミンを有している3,4)。これら長鎖ポリアミンや分岐鎖ポリアミンは、 (超) 好熱菌の生育温度の上昇に伴い、細胞内に著量蓄積されることから、高温環境での生育において重要な役割を有すると考えられる5,6)。例えば、長鎖ポリアミンや分岐鎖ポリアミンは、高温においてそれぞれDNAやRNAの安定化効果を有する7)。また、N4-ビス (アミノプロピル) ノルスペルミジン[3(3)(3)3]が翻訳効率を向上する効果を有することが知られている8)。これまで長鎖ポリアミンを合成する酵素としてスペルミジン/スペルミン合成酵素と相同性を有するタンパク質が超好熱性アーキアPyrobaculum aerophilumなどから同定されているが9)、分岐鎖ポリアミンの合成に関わる酵素は全く未知であった。近年、我々は超好熱性アーキアThermococcus kodakarensisが分岐鎖ポリアミンN4-アミノプロピルスペルミジン[3(3)4]及びN4-ビス (アミノプロピル) スペルミジン[3(3)(3)4]を有することを明らかにし、本菌より分岐鎖ポリアミン合成酵素BpsAを初めて同定した10)。本稿では、本酵素の発見の経緯及びその生理的役割、さらに速度論的解析やX線結晶構造解析から見えてきた触媒反応機構について最近の知見を紹介する。
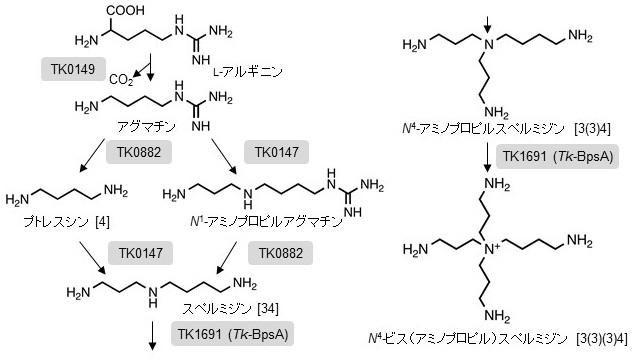
2.(超) 好熱菌のスペルミジン生合成機構
ポリアミンはアルギニン、オルニチン、メチオニンなどのアミノ酸を初発基質として合成される1)。多くの常温生物では、オルニチンデカルボキシラーゼの触媒反応によりオルニチンからプトレスシン[4]が生成する。植物やいくつかのバクテリアでは、アルギニンからアグマチンを介してプトレスシン[4]が生成する11,12)。スペルミジン[34]やスペルミン[343]は、アミノプロピル基転移酵素であるスペルミジンまたはスペルミン合成酵素が脱炭酸化されたS-アデノシルメチオニン (dcSAM) のアミノプロピル基をポリアミン基質へ転移する反応を触媒することにより生成する。これに対して、T. kodakarensisや好熱性バクテリアThermus thermophilusは常温生物にはみられないポリアミン生合成経路を有している (図1)6,13,14)。この経路では、まずアルギニンを初発基質としてアルギニン脱炭酸酵素の作用によりアグマチンを生成する。次に、スペルミジン合成酵素と相同性を有する酵素がアグマチンからN1-アミノプロピルアグマチンを生成する反応を触媒する。N1-アミノプロピルアグマチンウレオヒドロラーゼの触媒作用によりN1-アミノプロピルアグマチンが脱尿素されることで、スペルミジンが生成する。これまでに我々は、T. kodakarensisにおいてtk0147遺伝子やtk0882遺伝子がN1-アミノプロピルアグマチン合成酵素及びN1-アミノプロピルアグマチンウレオヒドロラーゼをコードすることを明らかにしている6)。また、これら酵素は、触媒効率は極めて低いがプトレスシン[4]を経由してスペルミジンを合成する経路にも関与する。これらの遺伝子破壊株はスペルミジンを合成できないため、野生株と比べて本菌の至適温度85℃での生育遅延が生じ、生育上限温度93℃では生育できない6)。これは、スペルミジンや分岐鎖ポリアミンが高温での生育に重要であることを示している。興味深いことに、tk0147遺伝子破壊株で、培地にスペルミジンを添加することでN4-ビス (アミノプロピル) スペルミジン[3(3)(3)4]の量が野生株と同等に回復することから、TK0147以外のアミノプロピル転移酵素がスペルミジンを基質として分岐鎖ポリアミンを生成すると考えられた。

3.分岐鎖ポリアミン合成酵素の同定と生理的意義
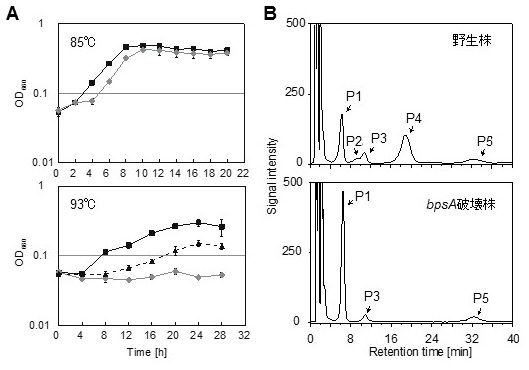
スペルミジン/スペルミン合成酵素のアミノ酸配列の相同性検索の結果から、T. kodakarensisはTK0147以外に相同なタンパク質を有していないことから、未知アミノプロピル転移酵素は既知のスペルミジン/スペルミン合成酵素とは一次配列が全く異なることが予想された。我々はT. kodakarensis KU216株 (ΔpyrF) の細胞抽出液からスペルミジンを基質としてN4-ビス (アミノプロピル) スペルミジン合成活性を示すタンパク質を各種クロマトグラフィーを用いて濃縮した。濃縮画分を質量分析計に供し、得られたデータからMASCOT検索によって4種のタンパク質TK0545、TK0548、TK1691、TK0967を同定した。これらはそれぞれ、S-アデノシルメチオニン合成酵素・アスパラギン酸アミノ基転移酵素・機能未知タンパク質・Xaa-Proアミノペプチダーゼと注釈づけられていた。これらタンパク質遺伝子の高発現系を大腸菌で構築し、精製した組換えタンパク質を得た。スペルミジンを基質としてN4-ビス (アミノプロピル) スペルミジン合成活性を測定したところ、TK0545・TK0548・TK0967は合成活性を示さなかった一方で、TK1691が合成活性を有していた10)。TK1691 (BpsA, branched-chain polyamine synthase A) は直鎖ポリアミンであるスペルミジン [34]や第三級ポリアミンであるN4-アミノプロピルスペルミジン [3(3)4]に対してアミノプロピル基転移活性を有していた。このことから、Tk-BpsAはスペルミジンを基質としてN4-アミノプロピルスペルミジン[3(3)4]を経由してN4-ビス (アミノプロピル) スペルミジン[3(3)(3)4]を合成する二機能性酵素であることが示された。次に、tk-bpsA遺伝子の生理的意義を調べるため、tk-bpsA遺伝子破壊株 (DBP1株) を作製した。DBP1株は野生株と比べて、85℃では誘導期の延長が見られた一方、最高菌体収量や比増殖速度は変化がなかった (図2A)。一方で、93℃ではDBP1株は生育しなかったため、tk-bpsA遺伝子は高温における生育に必須であることが明らかとなった。さらに、DBP1株をN4-ビス (アミノプロピル) スペルミジン[3(3)(3)4]を添加した培地で培養すると、生育がわずかに回復した。次に、tk-bpsA遺伝子破壊によるポリアミン組成の変化を検証した。その結果、DBP1株ではN4-アミノプロピルスペルミジン[3(3)4]やN4-ビス (アミノプロピル) スペルミジン[3(3)(3)4]の生成は見られなかったが、著量のスペルミジン[34]の蓄積が見られた (図2B)。この結果は、スペルミジン[34]の過剰蓄積が、85℃の生育温度においてN4-ビス (アミノプロピル) スペルミジン[3(3)(3)4]の機能を代替していることを示唆している。以上の結果から、Tk-BpsAがスペルミジン[34]を基質としてN4-ビス (アミノプロピル) スペルミジン[3(3)(3)4]の生成に関与することが明らかとなった。

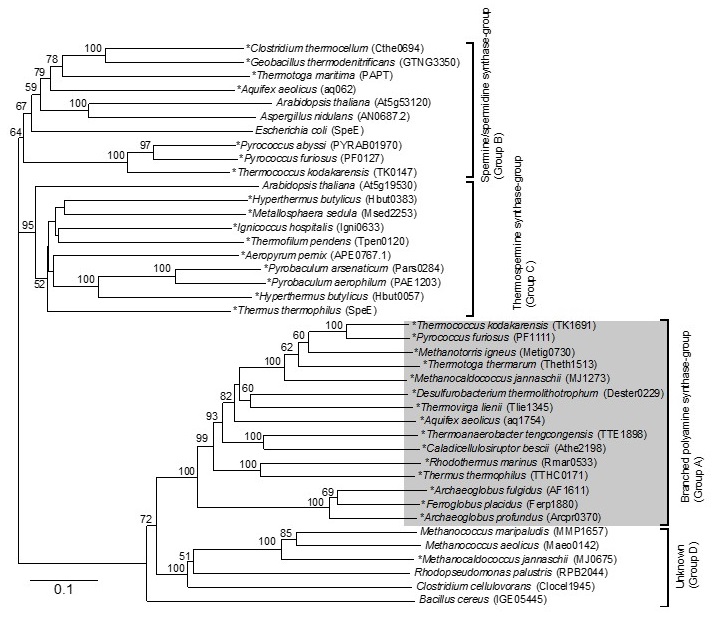
分岐鎖ポリアミンは (超) 好熱菌にのみ見られるため、BpsAのオルソログの分布も (超) 好熱菌に限られることが予想された。ゲノムDNA配列が既知の生物を対象にBpsAオルソログの分布を調べた結果、BpsAオルソログは (超) 好熱菌にのみ存在することが明らかとなった (図3)。また、クレンアーキオータ門に属する (超) 好熱菌にはBpsAオルソログの存在は認められなかった。これは、これら (超) 好熱菌には分岐鎖ポリアミンの存在が確認されていない事実と一致している15)。さらに、スペルミジン/スペルミン合成酵素とBpsAオルソログの系統解析の結果、同じアミノプロピル基転移酵素でありながら、全く異なるグループを形成していることが明らかとなった。スペルミジン/スペルミン合成酵素の触媒反応に関わるアミノ酸残基はBpsAオルソログには保存されていないことから、既知のアミノプロピル転移酵素とは全く異なる機構で反応が進行すると考えられた。系統解析の結果から、アミノプロピル基転移酵素は分岐鎖ポリアミン合成酵素 (グループA)、スペルミジン/スペルミン合成酵素 (グループB)、サーモスペルミン合成酵素 (グループC) の3つのグループに分類することができる。さらに、分岐鎖ポリアミン合成酵素と相同性を有する機能未知のタンパク質群が存在する (グループD)。グループDのタンパク質は、好熱菌に限らず常温菌においても見出される。常温性メタン生成アーキアMethanococcus maripaludisのMMP1657には、グループAのアミノプロピル基転移酵素に特徴的なGly-Asp-Asp-Aspモチーフが存在する。しかし、M. maripaludisにおいて分岐鎖ポリアミンは見出されておらず15)、後述するTk-BpsAの構造で明らかとなったポリアミンの結合に関わるアミノ酸残基の保存性が低いことから、MMP1657が属するタンパク質群 (グループD) は分岐鎖ポリアミン合成活性を有さないことが予想される。

4.分岐鎖ポリアミン合成酵素の酵素学的性質
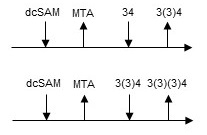
既知のアミノプロピル基転移酵素の多くは (ヒト、ウシ由来のスペルミン合成酵素やヒト、超好熱性バクテリアThermotoga maritima由来のスペルミジン合成酵素など)、逐次Bi Bi機構でアミノプロピル基転移反応が進行する16-19)。例えば、ヒト由来スペルミン合成酵素では、スペルミジンのN1位のアミノ基が周辺に配向するアスパラギン酸残基などにより脱プロトン化される。求核性の増したN1位のアミノ基は直接dcSAMのアミノプロピル基のα炭素を求核攻撃することで、スペルミンが生成する16)。Tk-BpsAのアミノプロピル基転移活性をdcSAMとスペルミジンまたはN4-アミノプロピルスペルミジン[3(3)4]を用いて反応速度論の解析を行ったところ、Lineweaver-Burk plotで平行線が得られたため、反応はピンポンbi bi機構で進行することが判明した (図4)。Tk-BpsAの様々な基質に対する特異性を調べたところ、直鎖ポリアミ
ンのスペルミジン [34]、ノルスペルミジン[33]、スペルミン[343]や分岐鎖ポリアミンのN4-アミノプロピルノルスペルミジン[3(3)3]やN4-アミノプロピルスペルミジン [3(3)4]に対して活性を示した。一方、N4-アミノプロピルスペルミン[3(3)43]に対して活性を示さなかった。これら基質ポリアミンに対する速度論的パラメーターを調べた結果、スペルミジン[34]に対するkcat /Km値は、N4-アミノプロピルスペルミジン[3(3)4]に対するkcat /Km値の約1%程度であった。T. kodakarensis細胞の主要なポリアミンはスペルミジン[34]とN4-ビス (アミノプロピル) スペルミジン[3(3)(3)4]であり、N4-アミノプロピルスペルミジン[3(3)4]の量はこれらに比べて微量である。これらの結果から、Tk-BpsAの反応特性がスペルミジン[34]とN4-ビス (アミノプロピル) スペルミジン[3(3)(3)4]の存在量を決定していると考えられた。

5.分岐鎖ポリアミン合成酵素の構造と機能
Tk-BpsA- N4-アミノプロピルスペルミジン[3(3)4]-メチルチオアデノシン (MTA) 三者複合体のX線結晶構造を1.8 Å分解能で決定した (図5A)。Tk-BpsAは、ホモダイマーを形成しており、その二量体形成面に一つのFe3+が配位していた。Tk-BpsAの構造は、S-アデノシルメチオニン依存的メチル基転移酵素と類似していた。Tk-BpsAの三者複合体のX線結晶構造は、スペルミジン/スペルミン合成酵素の構造と全く異なっていた。三者複合体のX線結晶構造解析の結果から、基質結合ポケットはアスパラギン酸残基やグルタミン酸残基などの酸性アミノ酸残基や、スレオニン残基やチロシン残基などヒドロキシル基を有するアミノ酸残基が多く存在した (図5B)。反応機構及び二機能性を生み出す仕組みを明らかにするため、触媒中心と考えられるアミノ酸残基 (Asp159) やN4-アミノプロピルスペルミジン[3(3)4]の結合に関与すると予測されるいくつかのアミノ酸残基 (Asp126, Glu259, Tyr316) に着目した。Asp159はBpsAオルソログで高度に保存されているGly-Asp-Asp-Aspモチーフに存在している。D159A変異型酵素はスペルミジン[34]及びN4-アミノプロピルスペルミジン[3(3)4]に対する活性を完全に失った。次に、三者複合体構造からTyr316, Glu259, Asp126は、N4-アミノプロピルスペルミジン[3(3)4]の分岐したアミノプロピル基の結合に必須と考えられた (図6)。Y316A変異型酵素やE259A変異型酵素のスペルミジンに対する触媒効率kcat /Km値は野生型酵素と比べてそれぞれ50%以下及び15%以下に減少した。またY316A変異型酵素のN4-アミノプロピルスペルミジン[3(3)4]に対するkcat値は野生型酵素と比べて1%以下に減少したが、Km値は野生型酵素と同等であったことから、Tyr316は基質の結合ではなく、触媒反応に関わることが予測された。一方、E259A変異型酵素ではN4-アミノプロピルスペルミジン[3(3)4]に対する活性が消失した。Tyr316のヒドロキシル基はGlu259のカルボキシル基と水素結合ネットワークを形成していると考えられるため、Glu259は触媒反応に関わると考えられた。これらの結果と上述の速度論的解析結果を踏まえて、次のような反応機構を予測した。Asp159のカルボキシル基がdcSAMのアミノプロピル基のα炭素を求核攻撃することで、Asp159がアミノプロピル化される。そして、スペルミジン[34]またはN4-アミノプロピルスペルミジン[3(3)4]のN4位のアミノ基がTyr316とGlu259により脱プロトン化される。求核性の増したN4位のアミノ基がアミノプロピル化されたAsp159を求核攻撃することで反応が進行する (図7)。一方、D126A変異型酵素はスペルミジン[34]を基質とした時の酵素活性は野生型酵素と同等であったが、N4-アミノプロピルスペルミジン[3(3)4]を基質とした時に、酵素活性が検出できなかった。この結果は、N4-アミノプロピルスペルミジン[3(3)4]の分岐したアミノプロピル基とAsp126との結合こそが二機能性酵素としての活性発現に重要であることを示唆している。
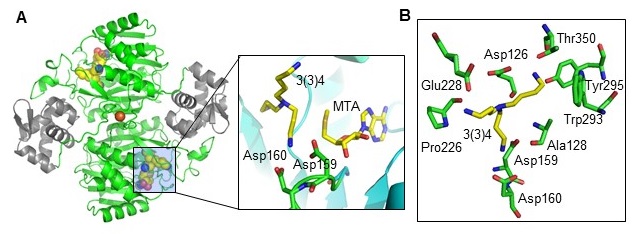

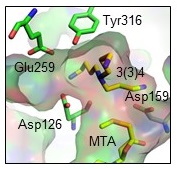

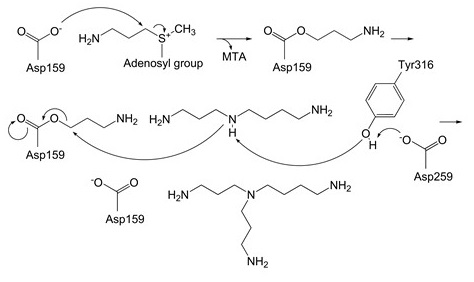
図7 Tk-BpsAの推定反応機構
6.おわりに
ここまで、Tk-BpsAの発見の経緯と生理的意義及び構造や速度論的解析をもとに推定した触媒反応機構について述べた。 (超) 好熱菌がもつ分岐鎖ポリアミンは種によって多様であるが、これはBpsAオルソログの基質特異性の違いによって説明されるものと考えている。Tk-BpsAで見られたポリアミン基質の結合に重要なアミノ酸残基が、BpsAオルソログ間で対応するアミノ酸残基が異なるためである。分岐鎖ポリアミンは (超) 好熱菌の高温適応において重要な因子であるが、その機能はRNAの構造安定化に関わること、翻訳効率を向上させること以外不明である。大腸菌では、スペルミジンは細胞膜の安定化20)、RNAポリメラーゼの活性化21)、リボソームの高次構造形成22)に関与する。さらに、スペルミジンはポリアミンモジュロンとよばれる特定の遺伝子の転写産物の翻訳に寄与している23)。そのため、分岐鎖ポリアミンが (超) 好熱菌において特定の遺伝子の転写や翻訳に関与すると考えられる。分岐鎖ポリアミンが (超) 好熱菌においてどのように機能しているかという謎の探究は、 (超) 好熱菌の高温適応機構の解明につながるものと思われる。一方、クレンアーキオータ門に属する (超) 好熱菌は分岐鎖ポリアミンを有さない事実は、分岐鎖ポリアミンは (超) 好熱菌の高温適応の必要十分条件ではないことを示唆している。さらには、常温生物に分岐鎖ポリアミンが存在しない理由も興味深い。分岐鎖ポリアミンの謎は深まるばかりである。
謝辞
本研究は、関西学院大学大学院理工学研究科生命科学専攻の岡田 和真さん、江淵 優希さん、木村 成吾さん、大阪大学大学院工学研究科応用化学専攻 井上 豪 先生、溝端 栄一 先生、Tse Ka Manさん、名古屋市立大学大学院薬学研究科生命薬科学専攻 樋口 恒彦 先生、梅澤 直樹 先生、城西大学大学院薬学研究科薬学専攻 新津 勝 先生、高尾 浩一 先生、立命館大学総合理工学院 今中 忠行 先生、福田 青郎 先生、共和化工環境微生物研究所 大島 泰郎 先生の協力により遂行されました。この場をお借りして、ご指導賜りましたこと、心から御礼を申し上げます。本研究は日本学術振興会 基盤研究 (B) 「超好熱菌の低温適応機構に関する包括的理解 (課題番号26292045) 」の支援を受けました。
文献
1) Wallace, H. M., Fraser, A. V., Hughes, A.: Biochem. J., 376, 1 (2003).
2) Jänne, J., Alhonen, L., Keinänen, T. A., Pietilä, M., Uimari, A., Pirinen, E., Hyvönen, M. T., Järvinen, A.:J. Cell Mol. Med., 9, 865 (2005).
3) Fujiwara, S., Hidese, R., Inoue, I., Fukuda, W.: Polyamines, a universal molecular nexus for growth, survival and specialized metabolism, 1, 143 (2015).
4) Oshima, T.: Plant Physiol. Biochem., 48, 521 (2010).
5) Oshima, T., Hamasaki, N., Senshu, M., Kakinuma, K., Kuwajima, I.: J. Biol. Chem., 262, 11979 (1987).
6) Morimoto, N., Fukuda, W., Nakajima, N., Masuda, T., Terui, Y., Kanai, T., Oshima, T., Imanaka, T., Fujiwara, S.: J. Bacteriol., 192, 4991 (2011).
7) Terui, Y., Ohnuma, M., Hiraga, K., Kawashima, E., Oshima, T.: Biochem. J., 388, 427 (2005).
8) Uzawa, T., Hamasaki, N., Oshima, T.: J. Biochem., 114, 478 (1993).
9) Knott, J. M.: FEBS Lett., 583, 3519 (2009).
10) Okada, K., Hidese, R., Fukuda, W., Niitsu, M., Takao, K., Horai, Y., Umezawa, N., Higuchi, T., Oshima, T., Yoshikawa, Y., Imanaka, T., Fujiwara, S.: J. Bacteriol., 196, 1866 (2014).
11) Tabor, C. W., Tabor, H.: Microbiol. Rev., 49, 81 (1985).
12) Imai, A., Matsuyama, T., Hanzawa, Y., Akiyama, T., Tamaoki, M., Saji, H., Shirano, Y., Kato, T., Hayashi, H., Shibata, D., Tabata, S., Komeda, Y., Takahashi, T.: Plant Physiol., 135, 1565 (2004).
13) Ohnuma, M., Terui, Y., Tamakoshi, M., Mitome, H., Niitsu, M., Samejima, K., Kawashima, E., Oshima, T.: J. Biol. Chem., 280, 30073 (2005).
14) Fukuda, W., Hidese, R., Fujiwara, S.: Polyamines, a universal molecular nexus for growth, survival and specialized metabolism, 1, 15 (2015).
15) Hamana, K., Hosoya, R., Itoh, T.: J. Jpn. Soc. Extremophiles, 6, 25 (2007).
16) Wu, H., Min, J., Zeng, H., McCloskey, D. E., Ikeguchi, Y., Loppnau, P., Michael, A. J., Pegg, A. E., Plotnikov, A. N.: J. Biol. Chem., 283, 16135 (2008).
17) Raina, A., Hyvonen, T., Eloranta, T., Voutilainen, M., Samejima, K., Yamanoha, B.: Biochem. J., 219, 991 (1984).
18) Wu, H., Min, J., Ikeguchi, Y., Zeng, H., Dong, A., Loppnau, P., Pegg, A. E., Plotnikov, A. N.: Biochemistry, 46, 8331 (2007).
19) Korolev, S., Ikeguchi, Y., Skarina, T., Beasley, S., Arrowsmith, C., Edwards, A., Joachimiak, A., Pegg, A. E., Savchenko, A.: Nat. Struct. Biol., 9, 27 (2002).
20) Souzu, H.: Biochim. Biophys. Acta., 861, 361 (1986).
21) Abraham K. A.: Eur. J. Biochem., 5, 143 (1968).
22) Kakegawa, T., Hirose, S., Kashiwagi, K., Igarashi, K.: Eur. J. Biochem., 158, 265 (1986).
23) Igarashi, K., Kashiwagi, K.: Methods Mol. Biol., 720, 51 (2011).