【トピックス】
真核生物における新規ピロロキノリンキノン依存性糖質酸化酵素の発見
松村洋寿、武田康太、中村暢文、吉田 誠、五十嵐圭日子、鮫島正浩
秋田大院・理工、東京農工大院・工、東京農工大院・工、東京農工大院・農、東大院・農生科、東大院・農生科
1.はじめに
ピロロキノリンキノン (PQQ) は、ニコチンアミド (NAD、NADP)、フラビン (FAD、FMN) に次いで発見された酸化還元補酵素であり、筆者らの知る限りPQQに関する最初の論文は、Acinetobacter calcoaceticus が産生するグルコース脱水素酵素 (sGDH) 中に、構造未知の補酵素が存在するとしたHaugeらによる報告 (1964年) である1)。その後1979年に、Duineらのグループ2) 及びSalisburyらのグループ3) によって、メタノール資化性細菌のメタノール脱水素酵素から補酵素が単離され、PQQの構造が決定された (図1)。当時、PQQを利用する酵素が、微生物だけでなく動植物などのその他の生物種にも広く分布しているのではないかと予想された。しかしながら、研究が進むにつれて、PQQの生合成経路が特定の細菌にしか存在せず、PQQ依存性の酵素も細菌類にのみ分布すると考えられるようになった。ところが近年、植物中にPQQが少量ではあるが存在しているという報告があり、植物がPQQを生合成している可能性が示唆された。ただし、植物にはPQQ生産性のメタノール資化性細菌が共生していることが知られていることから、植物自体にPQQの生合成が可能かは今後更なる検討が必要である。
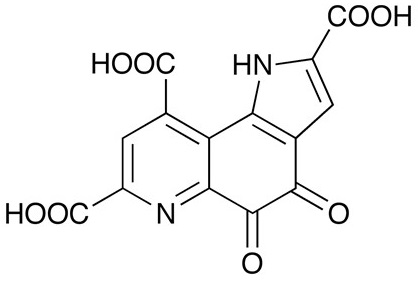
図1 PQQの化学構造
一方、PQQが植物や哺乳類において生理的に重要な役割を担っていることも報告されている4-7)。1989年にRuckerらのグループはPQQを添加していない合成飼料でマウスを飼育すると、成長障害、脱毛、皮膚の脆弱化などが起こることを報告し、PQQが哺乳類にとって必須栄養素であり、高等動物のビタミンである可能性を提唱した8)。このような栄養学的な知見から、ビタミンB2 (リボフラビン) やビタミンB3 (ナイアシン) と同様に、PQQも生体内の酸化還元反応に関与するビタミンの候補として注目が集まった。しかしながら、ヒトをはじめとする高等動物において、PQQが必須栄養素であるという確証が得られなかったことから、ビタミンとしては認められず、ビタミン様物質の1つとして分類された。その後2003年に、理化学研究所のKasahara らにより、マウスにおけるリジンの分解代謝に関与する2-aminoadipate 6-semialdehyde脱水素酵素 (AASDH) の遺伝子を解析したところ、そのアミノ酸一次配列中に細菌由来のPQQ依存性脱水素酵素のPQQ結合配列が存在したことから、AASDHがPQQ依存性の酵素である可能性が報告された9)。これにより、PQQが哺乳類にとってのビタミンであるか否かという議論が再燃したが、AASDHがPQQを酸化還元補酵素として実際に利用することが確認されていないことや、前述したようにPQQを補酵素として利用する酵素が細菌からしか見つかっていないことから、未だにPQQはビタミンとして認定されていない。
そのような状況のなか、2015年に著者らは真核生物由来では初めてとなるPQQ依存性糖質酸化酵素をきのこ (担子菌Coprinopsis cinerea、和名: ウシグソヒトヨタケ) から発見した10,11)。興味深いことに、このPQQ依存性酵素は、既知のPQQタンパク質とのアミノ酸一次配列の相同性は低いにもかかわらず、PQQに強く結合し、さらにPQQの存在下において高い活性を示すことが分かった。また、きのこだけでなく、カビやアメーバなどの真核生物も、本酵素に高い相同性を示す遺伝子を有していることが明らかになった。
2.発見に至る経緯
著者らが、この新規PQQ依存性酵素を発見したきっかけは、きのこの菌体外酸化還元酵素の探索ならびに機能解析の研究からであった。きのこは、生態系における分解者に位置づけられる生物で、様々な酵素を菌体外に分泌して有機物を分解、消化することで、成長するためのエネルギー源を得ている。著者らは、そのようなきのこの生物機能に着目して、植物バイオマス多糖の生分解に関与する酵素の機能解析と、それらの酵素を利用したバイオマスから有用物質生産のためのバイオ変換プロセス構築へ向けて研究を進めている。
きのこが産生する酵素のなかに、セロビオース脱水素酵素 (CDH) というユニークな酵素が存在する。1978年に担子菌Phanerochaete chrysosporiumから発見されたこの酵素は、植物細胞壁の主成分であるセルロースがセルラーゼによって加水分解された際に生成するセロビオースやセロオリゴ糖を基質として、ラクトンにする酸化反応を触媒する菌体外酸化還元酵素である12)。CDHの研究の進展により、糸状菌によるセルロースの分解は、セルラーゼやβ-グルコシダーゼ等の加水分解酵素のみならず、酸化還元酵素が関与していることが明らかになってきた。そのような背景から、著者らはCDHに着目し、きのこの菌体外に産出される酸化還元酵素の探索を試みてきた。
CDHは、FADを有するフラビンドメインとヘムbを有するシトクロムドメインがリンカーで結合されて成る、いわゆるフラボヘムタンパク質である13)。糖質の生合成、代謝等に関与する酵素は、糖質関連酵素 (Carbohydrate Active enZymes: CAZy) データベースに登録されており、フラビンドメインは、CAZyにおいて補助活性 (Auxiliary Activities: AA) ファミリー3、シトクロムドメインはAAファミリー8に分類されている14)。CDH、シトクロムドメインの分光学的解析15) から、MetとHis残基がヘムの配位子であり、さらにX線結晶構造解析13) により他のシトクロム類には見られない免疫グロブリン様βサンドイッチ構造16) をとっていることが明らかになっている。このシトクロムドメインは、唯一糸状菌にのみ相同遺伝子がみられることが分かっていたため、本研究ではこのシトクロムドメインを用いた相同性検索を行った結果、新規PQQ依存性酵素を発見することに成功した。
3.新規PQQ 依存性糖質酸化酵素CcSDHのドメイン構造
P. chrysosporiumCDHのアミノ酸配列 (accession no. AAB92262) を用いたBLAST検索により、C. cinereaのゲノム配列データベースを探索したところ、CDHのシトクロムドメインに相同性を示す領域 (Chromosome 6:1274274–1277156) が見出された。本領域からプライマーを設計し C. cinerea strain 533817) から抽出したmRNAを鋳型としてRT-PCRを行ったところ、726アミノ酸をコードするopen reading frameを含む2883 bpのcDNAが得られた (図2)。この塩基配列は、accession no. AB901366としてDDBJデータベースに登録されている。本遺伝子がコードするタンパク質を、SignalP ver. 4.1 server18) を用いて解析したところ、N末端側18アミノ酸残基がシグナル配列であることが明らかにされた。このことは、本酵素がC. cinereaの菌体外に分泌されていることを示唆している。また、NCBIのタンパク質データベースを用いたアミノ酸配列のホモロジー検索の結果、図2に示すようにシグナル配列に続くN末端側の配列は、CDHのシトクロムドメインに高い相同性を示し、ヘムbの配位子として推定されるメチオニン (Met83) とヒスチジン (His182) 残基、ジスルフィド結合を形成すると考えられるシステイン残基 (Cys138及びCys141) が、保存されていることも明らかになった。一方、本タンパク質のC末端には、糖結合モジュールファミリー1 (CBM1) に相同性を示す配列が存在していた。CBM1は、糸状菌の多くの糖質加水分解酵素に見られるCBMであり、結晶性セルロースに吸着することが知られていることからも、in vivoにおいて、本酵素は菌体外に分泌されセルロース表面に存在する可能性が示唆される。
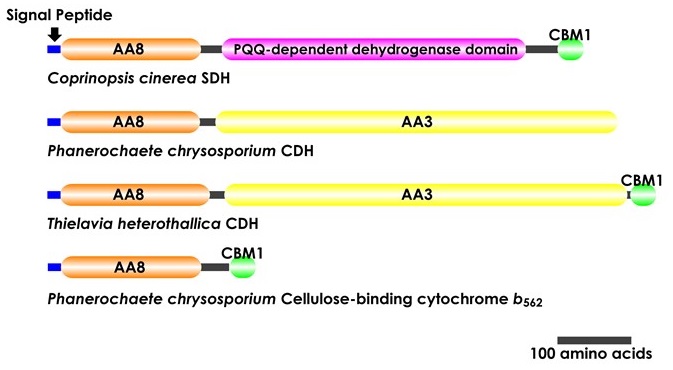

本タンパク質のアミノ酸配列解析結果で最も興味深かったのは、シトクロムドメインとCBM1に挟まれる領域に、未知のドメインが存在していたことである。この未知のドメインは、CDHにおけるフラビンドメイン (AAファミリー3) とは相同性を示さなかった。一方、PQQを補酵素とするタンパク質であることが知られているグルコース脱水素酵素およびソルボソン脱水素酵素には、低いながら相同性を示していた。最も高い相同性が得られたのは、Deinococcus radiodurans由来L-ソルボソン脱水素酵素 (26%) であった。
4.CcSDHの組換え体作製と酵素活性測定
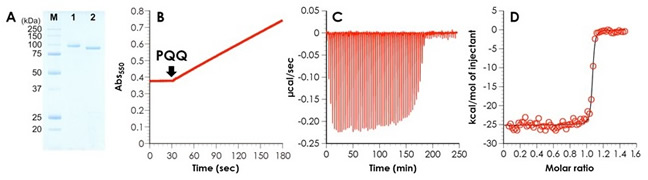
C. cinereaから得られた当該新規タンパク質をメタノール資化性酵母Pichia pastorisで、大量生産し精製することに成功した。この組換えタンパク質は、後述するように糖の酸化活性を示したことから、当初CcSDH (C. cinerea Sugar DeHydrogenase) と名付けたが、後にピラノースを特異的に酸化することが明らかになったことから、C. cinerea Pyranose DeHydrogenase (CcPDH) とした。SDS-PAGEの結果、精製したCcSDHは98±8 kDaにシングルバンドを示した (図3A)。この分子量は、アミノ酸配列から算出した77 kDaより大きいが、N-またはO-グリコシド結合によりCcSDHが糖鎖修飾されているためであると考えられる。エンドグリコシダーゼ H (Endo H) で処理した後、SDS解析を行ったところ、分子量の低下が実際に観測された。
これまでのCDHの研究をもとに、CDHと同様にCcSDHもシトクロムドメインを有していることから、未知のドメインは酸化還元活性を示すと予測し、NAD、NADP、FAD、またはPQQを2電子酸化反応の補酵素として試し、様々な糖やアルコール、アルデヒドに対する活性を、シトクロムcを電子受容体として用いて検討した (測定方法については、「5.CcSDHのPQQ依存性及びPQQ親和性」において詳述する)。その結果、PQQ存在下においてのみCcSDHの活性は観測され、PQQ依存性酵素に特徴的なCa濃度依存性を示した。
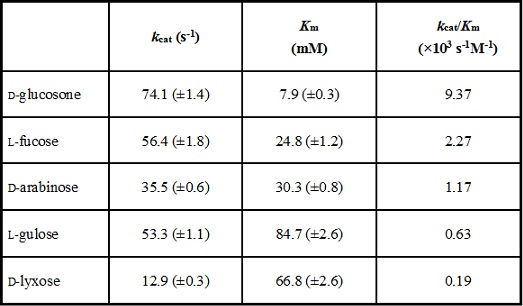
基質特異性においても、興味深い結果が得られた。CcSDHは、D-glucose、D-fructose、といった一般的な糖に活性を示さなかったのに対して、D-glucosone、L-fucoseなどの希少糖などに高い酸化活性を示した。各糖質に対する触媒効率は、D-glucosone > L-fucose > D-arabinose > L-gulose > D-lyxoseであり (表1)、最も高い触媒効率が得られたD-glucosoneに対するkcat /Kmは9.37 × 103 s-1M-1であった。今回、活性が見られた糖質に対するKm値は、全てmMオーダーであり、一般的な酵素の生理的な基質に対するKm値としては高い値であった。しかし、既知のPQQ依存性脱水素酵素であるA. calcoaceticusのsGDHでも、その生理的な基質であるD-glucoseに対するKm値が25 mMと高いことが知られていることから19)、CcSDHが最も高い触媒効率を示したD-glucosoneのKm値は7.9 mMであることは、PQQ依存性酸化還元酵素としては妥当な値であると考えられた。
表1 CcSDHの各単糖に対する速度論的パラメーター
5.CcSDHのPQQ依存性及びPQQ親和性
CDHの反応では、フラビンドメインによってセロビオースが酸化された際に生じる電子を、シトクロムドメインが分子内電子伝達によって受け取る。次いで、シトクロムcへ分子間電子伝達を行うことにより、CDHは再酸化される20)。したがって、CDHの活性は、シトクロムcの還元状態から測定することができる21,22)。CcSDHのシトクロムドメインも、同様にシトクロムcへの電子伝達が可能であると予測して酵素活性を測定した結果、上述の糖存在下でPQQを添加するとシトクロムcの還元が起こることが明らかとなった (図3B)。
次に、CcSDHの糖質酸化酵素ドメインのみをP. pastorisによって生産し、等温滴定カロリメトリー (ITC) によってPQQに対する結合定数を求めた (図3C)。図3Dに示すように、CcSDHの糖質酸化酵素ドメインのPQQに対する解離定数Kdは、1.11 nMと非常に低い値であった。ITC測定の結果により、 糖質酸化酵素ドメインがPQQに対する強い親和性を示し、1分子の糖質酸化酵素ドメインに対して、1分子のPQQが結合することが明らかになった。以上の結果から、CcSDHと既知のPQQ依存性酵素とのアミノ酸一次配列の相同性は高くはなかったが、著者らはCcSDHをPQQ依存性酵素であると結論付けた。

6.新規PQQ依存性糖質酸化酵素ファミリーの探索
その他の生物種におけるCcSDHのPQQ依存性糖質酸化酵素ドメインに類似したタンパク質の存在の有無を、BLAST検索により調べたところ、細菌やきのこだけでなく、カビやアメーバなど他の真核生物もこの酵素をコードする遺伝子を有することが明らかとなった。得られた CcSDHのPQQ依存性糖質酸化酵素ドメインおよびそのホモログ、既知のPQQ依存性脱水素酵素のアミノ酸配列に関して、ClustalXを用いて系統樹を作成したところ、図4に示すように、CcSDHは、既知のPQQ依存性の酵素とは明らかに異なるクレードを形成していた。
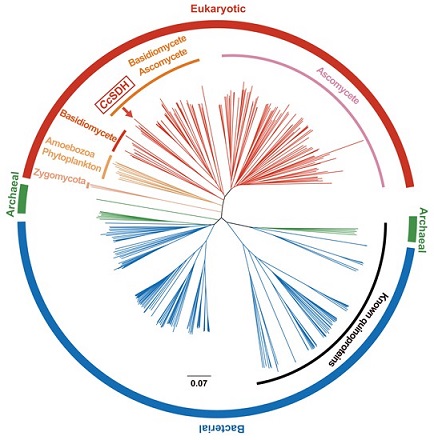

X線結晶構造解析によって、全てのPQQ依存性脱水素酵素において、4つの逆平行なシート構造が集まってプロペラの羽 (W-blade) のような構造になり、それらが6つ (6枚羽根) もしくは8つ (8枚羽根)集まって特徴的なスーパーバレル (βプロペラ構造) をとることが明らかになっており23-25)、6枚羽根と8枚羽根のβプロペラ構造を示すタンパク質間のアミノ酸一次配列に相同性はみられないことも明らかになっている。
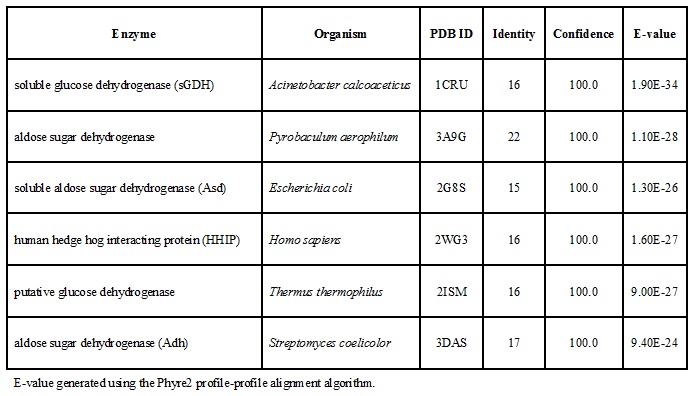
Protein Homology/analogy Recognition Engine (Phyre) ver. 2.026) タンパク質構造予測サーバーを用いて、CcSDHの三次構造をモデリングし、既知のPQQ依存性脱水素酵素の三次構造と比較を行ったところ、予測された構造は、PQQ依存性脱水素酵素に特徴的な6枚羽根のβプロペラ構造を示しており (図5)、構造既知の6枚羽根のプロペラ構造をもつ細菌由来PQQ依存性糖質脱水素酵素 (A. calcoaceticusのsGDH27-29) やPyrobaculum aerophilum由来アルドース脱水素酵素30)) などと類似していた (表2)。
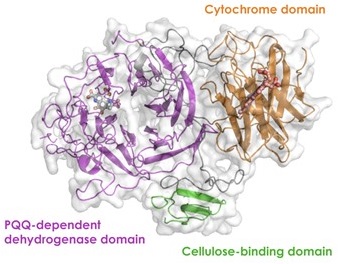

表2 Phyre2サーバーを用いたCcSDHの構造モデリングに対する相同性検索結果
更に興味深いことに、CcSDHはヒトヘッジホッグタンパク質 (HHIP)32) に対してアミノ酸配列の相同性を有しており、その相同性はバクテリアのPQQ依存性糖質脱水素酵素と同程度であった。高次構造の相同性の高かったPQQ依存性糖質脱水素酵素とHHIP、そしてCcSDHのアミノ酸一次配列アライメントを図6に示す。構造既知のPQQ依存性脱水素酵素において、触媒部位と考えられているHis残基は、これら全てのタンパク質間で保存されていたが、驚くべきことに、細菌の糖質脱水素酵素において、PQQ結合に寄与すると予測されているアミノ酸残基は、CcSDHとHHIPでは保存されていなかった。しかし、CcSDHがPQQ依存的に糖質酸化活性を示したこと、PQQに強い親和性を示した結果をふまえると、PQQの結合に寄与するアミノ酸が、本新規PQQ依存性糖質酸化酵素ファミリーにおいては異なっている可能性があり、真核生物におけるPQQ結合モチーフの多様性が推測される。


7.おわりに
本研究で発見した新規タンパク質は、AAファミリー8に分類されるシクトロムドメイン、PQQ依存性糖質酸化酵素ドメイン、そして糖結合モジュールを有しており、希少糖に作用するという興味深い性質を示した。本酵素は、植物細胞壁分解に関与していると考えられたことから、CAZyデータベースにおいてCcSDHの糖質酸化酵素ドメインが分類されるAAファミリー12が新設されることになった。
植物や哺乳類において、PQQの生理学的重要性は知られていたものの、著者らの知る限り、CcSDHの発見以前にPQQが真核生物由来の酵素において補酵素として働くという確証は得られておらず、実際にPQQ依存的に酵素反応を示す真核生物由来の酵素を精製し機能解析したのは、本研究が初めてである。一方、前述したように、自然界では、わずか数種類の細菌のみがPQQを生合成することが可能であることから、本酵素がどのようにPQQを獲得し活性を発現するのか、そのメカニズムについても興味が持たれる。
CcSDHのPQQ依存性糖質酸化酵素ドメインのホモログは、今のところ真核生物の中でも比較的下等な生物にしか見出されていないが、今回の真核生物における新規のPQQ依存性糖質酸化酵素ファミリーの発見は、高等動物を含むその他の真核生物においてもPQQを補酵素とする未知のタンパク質が存在する可能性を示唆していると我々は考えている。今日、PQQ依存性アルコール脱水素酵素が食酢の製造に利用される酢酸菌の酸化発酵に関与していることが知られており、PQQ依存性グルコース脱水素酵素は、血中酸素の影響を受けない特性を示すことから、グルコースセンサーとして製品化されている。真核生物において、新しい触媒機能を持つPQQ依存性酵素が見出されれば、バイオ変換技術やバイオセンサーなど酵素工学への応用展開が期待できる。今後、PQQがビタミンであるかということも含めた真核生物おけるPQQの重要性、PQQ依存性酵素の探索並びにその機能解明について更に研究、議論されることが期待される。
謝辞
本研究は、日本学術振興会科学研究費補助金基盤研究 (C) (中村暢文)、新学術領域研究「植物細胞壁の情報処理システム」 (五十嵐圭日子)、科学技術振興機構 先端的低炭素化技術開発 (ALCA) (五十嵐圭日子)、日本学術振興会特別研究員奨励費 (松村洋寿) の助成を受けたものです。厚く御礼申し上げます。
文献
1) Hauge, J. G.: J. Biol. Chem., 239,3630 (1964).
2) Westerling, J., Frank, J., Duine, J. A.: Biochem. Biophys. Res. Commun., 87, 719 (1979).
3) Salisbury, S. A., Forrest, H. S., Cruse, W. B., Kennard, O.: Nature, 280, 843 (1979).
4) Choi, O., Kim, J., Kim, J. G., Jeong, Y., Moon, J. S, Park, C. S., Hwang, I.: Plant Physiol., 146, 657 (2008).
5) Stites, T. E., Mitchell, A. E., Rucker, R. B.: J. Nutr., 130, 719 (2000).
6) Misra, H. S., Rajpurohit, Y. S., Khairnar, N. P.: J. Biosci., 37, 313 (2012).
7) Harris, C. B., Chowanadisai, W., Mishchuk, D. O., Satre, M. A., Slupsky, C. M., Rucker, R. B.: J. Nutr. Biochem., 24, 2076 (2013).
8) Killgore, J., Smidt, C., Duich, L., Romero-Chapman, N., Tinker, D., Reiser, K., Melko, M., Hyde, D., Rucker, R. B.: Science, 245, 850 (1989).
9) Kasahara, T., Kato, T.: Nature, 422, 832 (2003).
10) Matsumura, H., Umezawa, K., Takeda, K., Sugimoto, N., Ishida, T., Samejima, M., Ohno, H., Yoshida, M., Igarashi, K., Nakamura, N.: PLOS one, 9, e104851 (2014).
11) Takeda, K., Matsumura, H., Ishida, T., Samejima, M., Ohno, H., Yoshida, M., Igarashi, K., Nakamura, N.: PLOS one, 10, e0115722 (2015).
12) Ayers, A. R., Ayers, S. B., Eriksson, K. E.: Eur. J. Biochem., 90, 171 (1978).
13) Hallberg, B. M., Bergfors, T., Backbro, K., Pettersson, G., Henriksson, G., Divne, C.: Structure, 8, 79 (2000).
14) Levasseur, A., Drula, E., Lombard, V., Coutinho, P. M., Henrissat, B.: Biotechnol. Biofuels, 6, 41 (2013).
15) Cox, M. C., Rogers, M. S., Cheesman, M., Jones, G. D., Thomson, A. J., Wilson, M. T., Moore, G. R.: FEBS Lett., 307, 233 (1992).
16) Iyer, L. M., Anantharaman, V., Aravind, L.: Bioinformatics, 23, 2660 (2007).
17) Yanagi, S. O., Kawasumi, T., Takebe, I., Takemaru, T.: Agric. Biol. Chem., 52, 281 (1988).
18) Petersen, T. N., Brunak, S., von Heijne, G., Nielsen, H.: Nat. Methods, 8, 785 (2011).
19) Igarashi, S., Hirokawa, T., Sode, K.: Biomol. Eng., 21, 81 (2004).
20) Igarashi, K., Yoshida, M., Matsumura, H., Nakamura, N., Ohno, H., Samejima, M., Nishino, T.: FEBS J., 272, 2869 (2005).
21) Samejima, M., Eriksson, K. E.: Eur. J. Biochem., 207, 103 (1992).
22) Igarashi, K., Verhagen, M. F., Samejima, M., Schulein, M., Eriksson, K. E., Nishino, T.: J. Biol. Chem., 274, 3338 (1999).
23) Anthony, C., Ghosh, M.: Prog. Biophys. Mol. Biol., 69, 1 (1998).
24) Matsushita, K., Toyama, H., Yamada, M., Adachi, O.: Appl. Microbiol. Biotechnol., 58, 13 (2002).
25) Toyama, H., Mathews, F. S., Adachi, O., Matsushita, K.: Arch. Biochem. Biophys., 428, 10 (2004).
26) Kelley, L. A., Sternberg, M. J.: Nat. Protoc., 4, 363 (2009).
27) Oubrie, A., Rozeboom, H. J., Kalk, K. H., Olsthoorn, A. J., Duine, J. A., Dijkstra, B. W.: EMBO J., 18, 5187 (1999).
28) Oubrie, A., Rozeboom, H. J., Kalk, K. H., Duine, J. A., Dijkstra, B. W.: J. Mol. Biol., 289, 319 (1999).
29) Oubrie, A., Rozeboom, H. J., Dijkstra, B. W.: Proc. Natl. Acad. Sci. U S A.,96, 11787 (1999).
30) Sakuraba, H., Yokono, K., Yoneda, K., Watanabe, A., Asada, Y., Satomura, T., Yabutani, T., Motonaka, J., Ohshima, T.: Arch. Biochem. Biophys., 502, 81 (2010).
31) Southall, S. M., Doel, J. J., Richardson, D. J., Oubrie, A.: J. Biol. Chem., 281, 30650 (2006).
32) Bishop, B., Aricescu, A. R., Harlos, K., O’Callaghan, C. A., Jones, E. Y., Siebold, C.: Nat. Struct. Mol. Biol., 16, 698 (2009).