【トピックス】
ポリエチレンテレフタレート分解・代謝細菌の発見とそのメカニズムの解明
平賀和三、吉田昭介、宮本憲二、小田耕平
京工繊大 (当時) / RITE (現在)、京大院・工、慶大・理工、京工繊大
1.はじめに
石油を原料とするプラスチックの世界生産は現在3.1億トンに達している。これは50年前と比較して約20倍であり、さらに増加の一途を辿ると予想されている。翻って使用後のプラスチックの行方をみると、例えば容器包装用には7,800万トンが生産されているが、リサイクル用に収集されるのはその14%であり、他は焼却・エネルギー回収14%、埋め立て40%、環境への流出32%となっている1)。資源が有限である以上、現状のプラスチック経済は循環型・持続型への転換が急務である。また、環境に流出したプラスチックは微生物による分解を受けずに環境に蓄積し、景観の破壊や生物への物理的・化学的な悪影響をもたらしている。近年では、海洋におけるマイクロプラスチック (5 mm以下のプラスチック粒子と定義されることが多い) の小生物への影響も懸念されており2)、その回収技術の開発が求められている。
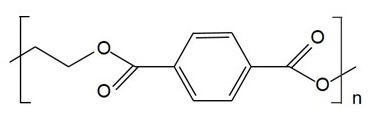
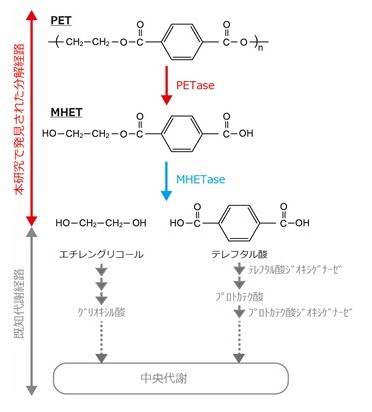
ペットボトルや衣類等に汎用されているポリエチレンテレフタレート (PET、世界生産量:約5,600万トン) は、ポリエチレン (PE)、ポリプロピレン (PP)、ポリビニルクロライド (PVC)、ポリスチレン (PS) と並ぶ主要なプラスチックの一つである。PETはテレフタル酸とエチレングリコールが縮重合した芳香族ポリエステルであり (図1)、生分解性が極めて低い3,4)。過去10年の研究では、PETは一部のリパーゼ、クチナーゼといった脂肪酸エステル加水分解酵素によって基質認識されるという報告がなされている5,6)。またFusarium属の真菌がPETを炭素源として分解、生育するという記述が認められる7,8)。これらの報告から、PETは「生分解されうる」ことがわかってきた。このような状況の中、筆者らはPETを分解・代謝する細菌を自然界より見出し、その能動的ともいえる分解メカニズムを解明した9,10)。本稿では、PET分解細菌の発見、分解酵素の同定・解析について解説し、PET生分解の進化的な成立について考察したい。
2.PET分解菌の探索
微生物分離源として、ペットボトルが大量に集積・加工されていた工場及びその周辺から、PET屑に接触した土壌や堆積物、処理水などの様々なサンプルを採取した。これらサンプルを非晶性のPETフィルム (以下、PETフィルム) を主炭素源とする培地に投入し、集積培養を試みた。数週間後、 PETフィルム上に特徴的な微生物塊を見出した (図2a)。顕微鏡で観察すると、細菌、酵母様細胞、原生動物など多種多様な微生物が存在していた。この微生物塊を取り除くと、顕著なPETフィルムの分解の進行を確認した (図2b)。当初、PET分解菌の分離には固体栄養培地上に形成するコロニーを対象に検索を行ったが、目的の菌を得ることができなかった。その原因として、使用した固体栄養培地上での増殖の速い微生物の優占など、目的外種の影響が考えられた。そこで、微生物塊を限界希釈して微生物群集の多様性を低減させ、その希釈菌液を用いてPETフィルムを主炭素源とする集積培養を行った。培養液における微生物の多様性を変性剤濃度勾配ゲル電気泳動 (DGGE) 法でモニタリングし、その顕著な低減を確認したのちに、低栄養固体培地上に菌液を播種した。このようにしてコロニーを形成した菌は、PETフィルムの薄片 (60 mg, 20×15×0.2 mm) を6週間で原型をほぼ消失させた。後に本菌は16S rRNA解析などからIdeonella属に分類される新種と判明した。そこで本菌が大阪府堺市で採取した環境サンプル由来であることに因み、Ideonella sakaiensis 201-F6株と命名した11)。これまでにIdeonella属のtype strainとしてIdeonella dechloratans CCUG 30898TとIdeonella azotifigens JCM 15503Tの2株が報告されているが、これらはI. sakaiensisと同様の条件では、PET分解性を示さなかった。興味深いことに、I. sakaiensisをPETフィルムと共に培養すると、繊維状の構造物を伸長し、PETフィルムと、あるいは菌同士で接着した (図3a,c)。栄養培地ではこのような細胞外構造物が確認できなかった。これらの観察から、繊維状構造物が炭素源であるPETへの菌体の近接効果をもたらしているのではないかと考えている。




3.I. sakaiensis のPET分解メカニズムの解明
3-1 PET分解酵素の探索と機能解析
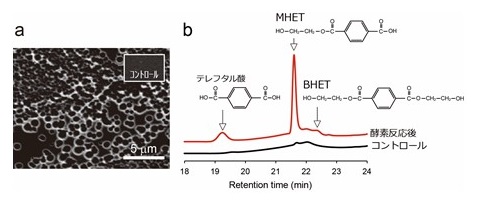
PET分解酵素に関する情報を得るため、I. sakaiensisのドラフトゲノム配列を決定し、5527個のORFを見出した。PET加水分解活性が報告されていた酵素の配列を用いて、それらのホモログをコードするORFを探索したところ、Thermobifida fusca由来の加水分解酵素TfH12)と51%の相同性を有する配列をコードするORF、ISF6_4831を見出した。そこで大腸菌を用いて組換えタンパク質を調製、精製し、PETフィルムとインキュベートしたところ、PETフィルム表面に無数のクレーター状分解痕が認められた (図4a)。さらにその反応溶液を逆相HPLCで分析したところ、分解産物と考えられるテレフタル酸、モノヒドロキシエチルテレフタレート (MHET)、ビスヒドロキシエチルテレフタレート (BHET) に対応するピークが認められた (図4b)。これらの結果から、ISF6_4831がPET加水分解活性を有する酵素をコードする遺伝子であることがわかった。次に、本酵素と既報のPET加水分解活性をもつ酵素との機能比較を試みた。まず、これら既報酵素のうちアミノ酸配列が公開されている11配列との進化系統樹を作製した (図5a)。本系統樹上でそれぞれ進化的に分岐した好熱性放線菌由来酵素TfH、枝葉コンポストのメタゲノム由来酵素LCC13)、真菌由来酵素FsC14)の3種を比較対象として選んだ。そして、これらの精製組換えタンパク質を調製し、ISF6_4831タンパク質と同条件下 (pH7.0、30℃) で活性測定を行った。まず、リパーゼやクチナーゼの基質となる脂肪酸エステルとして、炭素鎖長2〜8の直鎖脂肪族カルボン酸と発色団para-nitrophenolが脱水縮合した人工基質を用いた。その結果、これらの基質に対し、ISF6_4831タンパク質は3種の酵素と比べ、総じて低活性であった (図5b左)。一方、PETフィルムに対しては、TfHの120倍、LCCの5.5倍、FsCの88倍の活性を示した (図5b右)。

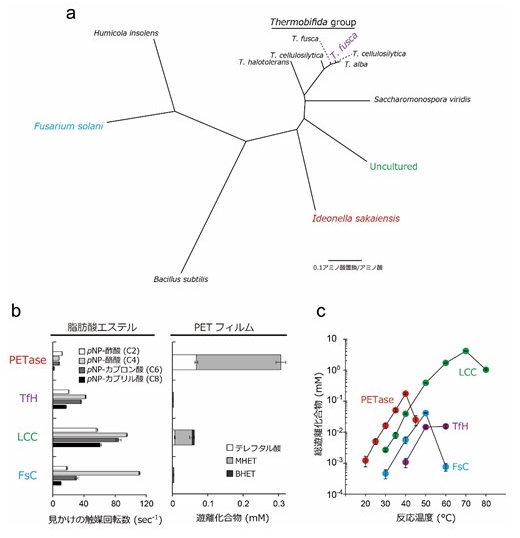


と定義すると、ISF6_4831タンパク質のそれはTfHの380倍、LCCの48倍、FsCの400倍であった。このように、ISF6_4831タンパク質はPETに対してより高い基質特異性を持つ酵素と考えられることから、PET加水分解酵素 (PETase) と命名した。次に、これら酵素のPET加水分解活性への温度の影響を調べたところ、PETaseは3種の酵素と比べ、40℃以下の低温で高PET分解活性を示したが、それ以上の温度では熱変性の影響が見られた (図5c)。ポリエステル鎖はその可動性が高いほど、酵素分解されやすいことが報告されている15)。PETは常温において流動性が低下したガラス状態 (PETのガラス転移点は約75℃) にある。このことから、PETaseはガラス状態のPETにも作用しやすい機構をもつものと考えられる。このようなPETaseのPETに対する高い基質特異性、及び常温域における高活性はI. sakaiensisが環境中でPETを炭素源として生存するために有利な性質と考えられる。
3-2 MHET加水分解酵素の探索と機能解析
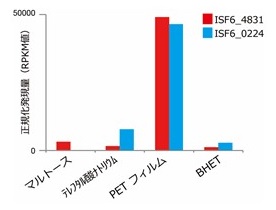
PETaseはPETフィルムを加水分解し、主にMHETとテレフタル酸を反応溶液中に遊離したが、MHETに対する加水分解活性を示さなかった。一方で、I. sakaiensisをPETフィルムとともに培養し、その培養上清中のMHET濃度を測定したところ、分解されたPET量に対応するMHETユニットと比べ、極めて低濃度であった。以上の事実から、I. sakaiensisは菌体内にMHETを取り込み、その代謝酵素としてMHET加水分解酵素を有していると推察した。これまでにPET加水分解活性を持つ一部の酵素においてMHET加水分解活性が報告されている16-18)。しかし、I. sakaiensisドラフトゲノム上で、それらのホモログをコードするORFはPETase遺伝子のみであるため、これらの既知酵素とは全く配列の異なるタンパク質がMHETの加水分解を担っていると考えられた。そこで、I. sakaiensisの網羅的な遺伝子発現解析から、MHET加水分解酵素遺伝子の予測を試みた。まず、I. sakaiensisをマルトース、テレフタル酸ナトリウム、BHET、PETフィルムをそれぞれ主炭素源として培養した。テレフタル酸、BHET はPET中間代謝物となりうる化合物である。マルトースはPET代謝の陰性対照として用いた。それぞれ対数増殖期中期にある菌体からmRNAを回収し、次世代シーケンサーにより網羅的に配列を解読後、I. sakaiensisドラフトゲノム上へのマッピングを行うことで各ORFの発現量を見積もった。次に、各ORFの4種の培地における発現パターンをクラスタリング解析により分類した。その結果、PETase遺伝子と同クラスターに分類されたORF、ISF6_0224を見出した。両ORFは、PETフィルムを主炭素源とする培地において最も高発現であった (図6)。これらのことから、ISF6_0224はPETase遺伝子と類似の発現制御を受けていると推測され、それと同時に代謝経路を共有している可能性が示唆された。また、ISF6_0224タンパク質の一次配列は没食子酸エステルやフェルラ酸エステル、クロロゲン酸といった芳香族化合物のエステル結合を加水分解する酵素の一群であるtannase familyに属することがわかった。本familyの触媒三残基 (Ser, His, Asp)、及び特徴的な二つのシステイン残基19)は、ISF6_0224タンパク質においても同様に認められる。以上の理由から、ISF6_0224をMHET加水分解酵素候補遺伝子として着目した。組換えタンパク質を作製、精製し、MHETに対する活性を測定したところ、明瞭な加水分解活性を示した。その反応速度論解析の結果は、kcat = 31 s-1、Km = 7.3μMであった。その一方で、ISF6_0224タンパク質はtannase familyに属する酵素が基質とする典型的な化合物や、PETフィルム、脂肪酸エステル人工基質には活性を示さなかった。以上のように、本酵素はMHETに対する基質特異性が極めて高いことから、MHET加水分解酵素 (MHETase) と命名した。
4.おわりに
本研究において、PET分解・代謝細菌I. sakaiensisを見出し、その酵素によるPET分解メカニズムを明らかにした。PETaseはPETを加水分解してMHETとテレフタル酸を主に遊離し、MHETaseはMHETを迅速に加水分解することで既に代謝経路が明らかとなっているテレフタル酸、エチレングリコールに変換した。筆者らは、これらの酵素が自然界には存在しない基質を効率よく分解することから、PET分解経路の成立について進化的な興味を持った。そこで、ゲノムが明らかになっている微生物を対象として、PET代謝に必須と考えられるPETase、MHETase、テレフタル酸ジオキシゲナーゼ、プロトカテク酸ジオキシゲナーゼ (図7) のホモログ遺伝子を探索したが、すべてを有する微生物は見出されなかった。一方でMHETaseホモログを持つ92の微生物のうち33はテレフタル酸ジオキシゲナーゼ、プロトカテク酸ジオキシゲナーゼを共に有することから、MHETアナログに対する代謝系の成立は古く、後にPETaseホモログがこれに加わることで、代謝系の拡張がなされたと考えられる。加えて、個々の酵素の基質特異性の向上が本経路の成立には欠かせない。自然界や試験管内でのPETが豊富且つ他の炭素源が乏しい環境が、これら代謝遺伝子セットの自然選択、酵素の分子進化を促進したのではないかと考えている。
謝辞
本稿で紹介した研究は、京都工芸繊維大学、帝人株式会社、株式会社 ADEKA、慶應義塾大学との共同で行われたものです。共同研究者の皆様に深く御礼申し上げます。
文献
1) Neufeld, L., Stassen, F., Sheppard, R., Gilman, T., Eds.: World Economic Forum, 2016, www3.weforum.org/docs/WEF_The_ New_Plastics_Economy.pdf. (2016).
2) Law, K. L., Thompson, R. C.: Science, 345, 144 (2014).
3) Muller, R. J., Kleeberg, I., Deckwer, W. D.: J. Biotechnol., 86, 87 (2001).
4) Kint, D., Munoz-Guerra, S.: Polym. Int., 48, 346 (1999).
5) Ribitsch, D., Acero, E. H., Greimel, K., Eiteljoerg, I., Trotscha, E., Fredd, G., Schwab, H., Guebitz, G. M.: Biocatal. Biotransform., 30, 2 (2012).
6) Zimmermann, W., Billig, S.: Adv. Biochem. Eng. Biotechnol., 125, 97 (2010).
7) Nimchua, T., Punnapayak, H., Zimmermann, W.: Biotechnol. J., 2, 361 (2007).
8) Nimchua, T., Eveleigh, D. E., Sangwatanaroj, U., Punnapayak, H.: J. Ind. Microbiol. Biot., 35, 843 (2008).
9) Yoshida, S., Hiraga, K., Takehana, T., Taniguchi, I., Yamaji, H., Maeda, Y., Toyohara, K., Miyamoto, K., Kimura, Y., Oda, K.: Science, 351, 1196 (2016).
10) Yoshida, S., Hiraga, K., Takehana, T., Taniguchi, I., Yamaji, H., Maeda, Y., Toyohara, K., Miyamoto, K., Kimura, Y., Oda, K.: Science, 353, 759 (2016).
11) Tanasupawat, S., Takehana, T., Yoshida, S., Hiraga, K., Oda, K.: Int. J. Syst. Evol. Microbiol., 66, 2813 (2016).
12) Muller, R. J., Schrader, H., Profe, J., Dresler, K., Deckwer, W. D.: Macromol. Rapid Commun., 26, 1400 (2005).
13) Sulaiman, S., Yamato, S., Kanaya, E., Kim, J. J., Koga, Y., Takano, K., Kanaya, S.: Appl. Environ. Microbiol., 78, 1556 (2012).
14) Silva, C. M., Carneiro, F., O'Neill, A., Fonseca, L. P., Cabral, J. S. M., Guebitz, G., Cavaco-Paulo, A.: J. Polym. Sci. Part A: Polym. Chem., 43, 2448 (2005).
15) Marten, E., Muller, R. J., Deckwer, W. D.: Polym. Degrad. Stab., 88, 371 (2005).
16) Eberl, A., Heumann, S., Bruckner, T., Araujo, R., Cavaco-Paulo, A., Kaufmann, F., Kroutil, W., Guebitz, G. M.: J. Biotechnol., 143, 207 (2009).
17) Acero, E. H., Ribitsch, D., Steinkellner, G., Gruber, K., Greimel, K., Eiteljoerg, I., Trotscha, E., Wei, R., Zimmermann, W., Zinn, M., Cavaco-Paulo, A., Freddi, G., Schwab, H., Guebitz, G.: Macromolecules, 44, 4632 (2011).
18) Ribitsch, D., Heumann, S., Trotscha, E., Herrero Acero, E., Greimel, K., Leber, R., Birner-Gruenberger, R., Deller, S., Eiteljoerg, I., Remler, P., Weber, T., Siegert, P., Maurer, K. H., Donelli, I., Freddi, G., Schwab, H., Guebitz, G. M.: Biotechnol. Prog., 27, 951 (2011).
19) Suzuki, K., Hori, A., Kawamoto, K., Thangudu, R. R., Ishida, T., Igarashi, K., Samejima, M., Yamada, C., Arakawa, T., Wakagi, T., Koseki, T., Fushinobu, S.: Proteins, 82, 2857 (2014).