【トピックス】
[Fe]-ヒドロゲナーゼの活性中心コファクターと立体構造解析に基づくその生合成酵素の同定
嶋 盛吾、藤城貴史
マックスプランク陸生微生物学研究所/JST・CREST、マックスプランク陸生微生物学研究所/埼玉大・理工
1.はじめに
ほとんどのメタン生成菌は炭酸ガス (CO2) を水素ガス (H2) からの電子で還元してメタンを生産することができる1)。H2を活性化して電子やヒドリドイオンを取り出す反応はヒドロゲナーゼと呼ばれる一群の酵素によって触媒される。メタン生成菌のヒドロゲナーゼとして[NiFe]-型と[Fe]-型が知られているが、培地中のニッケル濃度が制限された条件 (50 nM 以下) では[Fe]-ヒドロゲナーゼの生産量が通常の5-7倍に増加し、主要なヒドロゲナーゼとなる2)。[Fe]-ヒドロゲナーゼは、補欠分子族としてFe グアニリルピリジノール (FeGP) コファクターを活性中心に持つ (図1)3)。このコファクターは単核の鉄錯体を含み、低スピンの2価鉄がピリジノールの窒素とピリジノール環の6位のアシルメチル置換基のアシル炭素に固定され、2分子の一酸化炭素 (CO) 配位子が鉄に結合している。このコファクアーはシステイン残基のチオール基と鉄との共有結合によってタンパク質上に固定され、4,5)。またピリジノール環に結合しているグアノシン一リン酸 (GMP) 部位がタンパク質との水素結合や疎水性相互作用によって、FeGPコファクターのタンパク質への結合に寄与している。FeGPコファクターに見られるような鉄錯体構造は他に例は見られず、さらにこのピリジノールは二つのメチル基、アシルメチル基およびGMPによって修飾されているが、このような構造を持つピリジノールも自然界には知られていない。私たちはFeGPコファクターの生合成経路に興味を持ち研究に着手した。
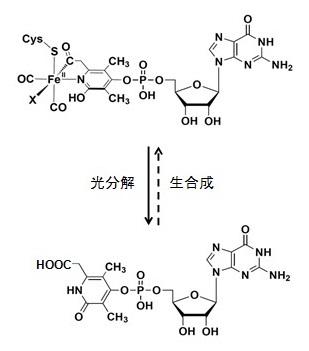
図1 FeGPコファクターとグアニリルピリジノール
2.FeGPコファクターの光分解産物
FeGPコファクターは短波長の光 (UV-Aから青色) の照射により鉄錯体部分が分解する。この光分解反応によりCOと鉄イオンが遊離し6)、グアニリルピリジノールのアシルメチル基は加水分解されカルボキシメチル基となる (図1)7)。FeGPコファクターの構造解析には、この光分解産物 (グアニリルピリジノール) のNMR解析結果が重要な役割を果たした8)。CO配位子とアシル基の存在は赤外分光分析9)とX線吸光分析10)およびガスクロマトグラフィー11)によって明らかとなった。最終的な錯体構造のジオメトリーは[Fe]-ヒドロゲナーゼの結晶構造解析を基にして決定された4,5)。
3.hcg 遺伝子クラスター
[Fe]-ヒドロゲナーゼを有するすべてのメタン生成菌のゲノムにはhmd-co-occuring (hcg) と呼ばれる遺伝子 (hcgA-G) が保存されている1)。多くのメタン生成菌で、これら七つのhcg遺伝子が遺伝子クラスターを形成している (図2)。hcg遺伝子は[Fe]-ヒドロゲナーゼの活性発現に必要なFeGPコファクター生合成に関与していることが予想されていた1)。HcgAはビオチン生合成酵素のひとつであるBioBや[FeFe]-ヒドロゲナーゼのコファクター生合成遺伝子であるHydEやHydGとの間に一次構造類似性が認められた。しかし他のHcgタンパク質に関しては、高い一次構造類似性を持つ機能既知のタンパク質の情報は得られなかった。そこで私たちはタンパク質立体構造の類似性から機能を推定することを目的として、Hcgタンパク質を大腸菌で異種生産し結晶構造解析を行った。

4.Hcgタンパク質の結晶構造と機能の同定
4-1 HcgBはグアニリル基転移酵素
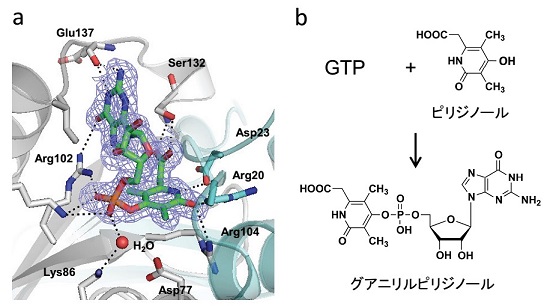
HcgBの立体構造はヌクレオチドトリホスファターゼとの間に立体構造の類似性が認められた。ヌクレオチドトリホスファターゼはヌクレオチド三リン酸を加水分解してピロリン酸とヌクレオチド一リン酸を生成する酵素である。FeGPコファクターの構造を眺めてみるとGMP部分がある。グアノシン三リン酸 (GTP) を前駆体としてFeGPコファクターの骨格を合成する場合、GTPトリホスファターゼに似た反応が必要である。構造を比較するとHcgBにはヌクレオチドトリホスファターゼの活性部位であるアスパラギン酸残基が欠如しており、実際にヌクレオチドトリホスファターゼ活性は検出されなかった。この結果から、HcgBがGTPからピリジノールにグアニリル基を転移する酵素であることが予想された。GTPとともに、数種のピリジン誘導体を第二の基質としてアッセイに使用した。質量分析によってグアニリルピリジノールの生成を確認し、さらにNMR分析で生産物の構造を解明した。数種のピリジン誘導体をテストした結果、2,4-ジヒドロキシピリジン誘導体のみがHcgB反応に利用されることが明らかとなった (図3)12)。
HcgB反応によって2,4-ジヒドロキシ3,6-ジメチルピリジンとGTPから人工的なグアニリルピリジノールを調製した12)。この人工的グアニリルピリジノールとHcgBの共結晶構造の解析ではピリジノール環の6位の位置に空間があり、その空間にはひとつの水分子が結合していた。次にFeGPコファクターの光分解によってグアニリルピリジノールを調製し、HcgBとグアニリルピリジノールを共結晶化したところ、その6位カルボキシメチル基がHcgBの構造にすっぽりと収まり、カルボキシル基がセリン残基と結合していた。この結果から、6-カルボキシメチル基を有するグアニリルピリジノールは、単にFeGPコファクターの光分解産物であるだけではなく、FeGPコファクター合成の前駆体である可能性が強く示唆された (図3)。

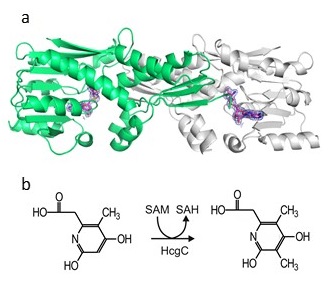
4-2 HcgCはメチル基転移酵素
一次構造比較では、HcgCと類似した機能既知のタンパク質は検出できなかった1)。しかしながら、立体構造はNAD (P) およびS-アデノシルメチオニン (SAM) を用いる酵素と相同性を示した13)。この結果よりHcgCがNAD (P) もしくはSAMを基質として利用する酵素タンパク質である可能性が考えられた。これらの補酵素とHcgCのアフィニティーを調べるために共結晶化を試みたところ、SAMが結合したHcgC構造のみが得られた。SAMを補酵素とする酵素としてはメチル基転移酵素などが知られている。かつて私たちの研究室で行なった安定同位体標識実験により、FeGPコファクターのピリジノールの3位メチル基がメチオニンのメチル基由来であることが明らかにされていた。この結果はFeGPコファクター生合成にSAM依存性のメチル基転移酵素が必要であることを示唆している。そこでHcgCはSAMからピリジノール3位へのメチル基転移反応を触媒する酵素であると予想した。基質との共結晶構造を用いたドッキングシミュレーション結果は、メチル基受容体は、6-カルボキシメチル-4-ヒドロキシ-5-メチル-2-ピリジノール (ピリジノール1) であることを示唆した14)。このピリジノールは2位と4位にヒドロキシル基が存在し、互変異性によって3位の酸性度が高まり、メチル化反応が容易になることが予想された (図4)。スイス連邦工科大学ローザンヌ校のXile Hu教授との共同研究でピリジノール1を化学合成し、HcgCの酵素活性測定に供したところ、ピリジノール1の3位がSAMによってメチル化されることが質量分析とNMRによって示され、HPLCを用いた実験により活性値 (kcat = 2.5 min-1) 得られた13)。
![]()
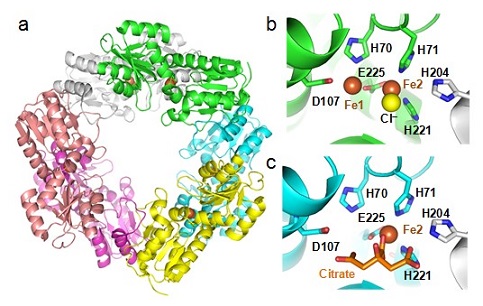
4-3 HcgDは2核鉄錯体を含む
一次構造解析によりHcgDはNif3ファミリーと呼ばれる機能未知のタンパク質群に類似性を示した1)。結晶構造解析により、HcgDはNif3ファミリータンパク質と同様に2核鉄錯体を含んでいることが明らかとなった14)。Nif3ファミリータンパク質の機能が知られていないため、立体構造の類似性から機能を類推することはできなかったが、HcgDをクエン酸などのキレーターを含む溶液中で結晶化すると2核鉄錯体の片方の鉄だけが特異的に除去されることが示され (図5)、この実験結果からHcgDは鉄をFeGPコファクターに供給する役目を果たしていると予想している14)。

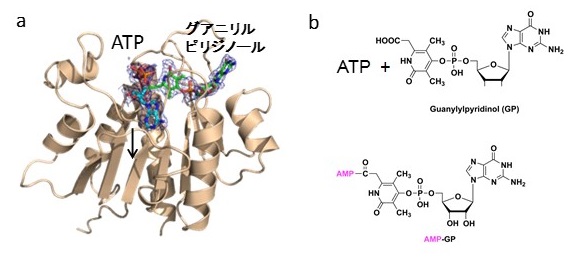
4-4 HcgEはアデニリル基転移酵素
HcgEの一次構造はユビキチン活性化酵素との間に相同性が認められた1)。しかし、FeGPコファクター生合成とユビキチン活性化にどのような類似点があるのか、当初は全く見当がつかなかった。HcgEの結晶構造を解析したところ、ユビキチン活性化酵素のATP結合ドメインの立体構造との間に高い類似性が示された15)。しかし、ユビキチン活性化酵素のユビキチン結合ドメインは、HcgEには保存されていなかった。ユビキチン活性化酵素とユビキチンの複合体結晶構造を眺めると、興味深い点が認められた。ユビキチン活性化系ではユビキチン様タンパク質のC末端カルボキシル基がATPと反応してアデニリル化される。この構造はHcgEがカルボキシル基にATPを作用させ、アデニリル化する酵素であることを示唆した。
上記のHcgBとグアニリルピリジノールとの共結晶化実験では6-カルボキシメチル-グアニリルピリジノールがHcgBの生産物である可能性を示唆した。私たちはグアニリルピリジノールのカルボキシル基がHcgEの触媒反応によってアデニリル化されることを予想し、先ず、ATPとグアニリルピリジノールをHcgEと共結晶させたところ、予想されたように、グアニリルピリジノールから伸びるカルボキシメチル基の先端の酸素原子がATPのα位のリン酸の近くに結合した構造が認められた (図6)15)。 (この共結晶化条件では、触媒活性に必要なマグネシウムが含まれないため反応が進行しない。) HcgE反応の生産物を質量分析で解析しアデニリル化されたグアニリルピリジノールが生成していることを確認し、アデニリル化に伴うピロリン酸の生成速度からHcgEの活性を明らかにした15)。

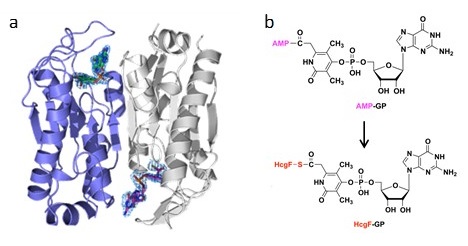
4-5 HcgFはアシル基合成シャペロンタンパク質か
HcgFの一次構造は機能が知られているタンパク質との類似性が検出されなかった。結晶構造を解析し、立体構造の類似性を調べたところ、ニコチンアミドモノヌクレオチドを結合するタンパク質との間に類似性が認められた14)。ニコチンアミドとピリジノールの構造類似性からグアニリルピリジノールを結合できるのではないかと考えた。グアニリルピリジノールとの共結晶化を行ったところ、実際にHcgFに結合することが判った (図7)。ホモ2量体を形成するHcgFには活性部位が2箇所存在する。活性部位の両方にグアニリルピリジノールが結合し、そのカルボキシメチル基部分は部分的にHcgFのCys9とチオエステル結合を作っていることが明らかになった。ユビキチンの活性化では同様にC末端のカルボキシル基がアデニリル化された後、システインのチオール基とチオエステル結合することが知られている。この結果から、私たちはHcgFにチオエステル結合したグアニリルピリジノールはアシル基合成の前駆体ではないかと考えている15)。

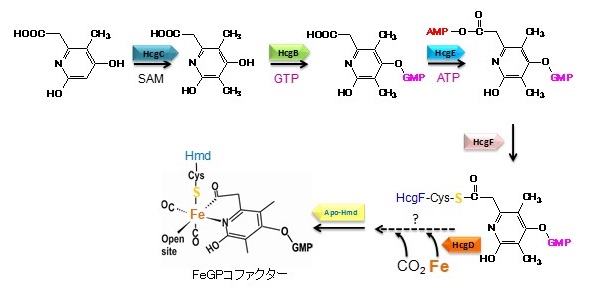
5.これまでに解明されたFeGPコファクター生合成経路
タンパク質結晶構造を基にした解析により、これまでにhcg遺伝子クラスターにコードされるタンパク質のうちHcgB、HcgCおよびHcgEの機能を解明できた (図8)。また、HcgDとHcgFの機能を推定した。ピリジノール環が形成された後に、HcgCの触媒反応によりその3位にメチル基が導入される。HcgBによってピリジノールにGMPが結合した後、カルボキシルメチル基はHcgEによってアデニリル化され、その後HcgFのシステイン残基に結合する。おそらく、この後HcgFにチオエステル結合したグアニリルピリジノールに鉄が導入されアシル基が形成されると考えられるが、アシル鉄結合反応がどのようにして進行するのかは今のところ不明である。HcgAとHcgGの機能はまだわかっていないが、これらのタンパク質に対しても「立体構造からの機能推定」を目指して研究を進めている。
![]()
6.おわりに
ゲノム解析によって得られた多くの遺伝子の機能を特定するには様々な手法がある。構造ゲノム学もそのひとつであるが、タンパク質の構造情報のみに頼って機能を推定することは容易なことではない。しかし、他の情報で機能を絞り込むことができる場合には、タンパク質構造が重要な示唆を与えてくれることがある。反応基質として候補となる化合物が特定できる場合には、共結晶法が有効である。Hcgタンパク質の一連の研究では、FeGPコファクターの光分解物としてグアニリルピリジノールを調製できたことが幸いして、有効な共結晶化実験が可能になった。現在は、3位のメチル基を持たないピリジノールを化学合成できるので、HcgCとHcgBの触媒反応によりグアニリルピリジノール前駆体を生合成できる。今後のFeGPコファクター生合成の生化学実験において大いに役立つものになるだろう。
謝辞
本研究はマックスプランク陸生微生物学研究所で実施された。結晶構造解析はマックスプランク生物物理学研究所のウルリッヒ=エルムラー博士との共同研究結果である。あわせて原著論文の共著者にも感謝の意を表する。本研究はマックスプランク財団および科学技術振興機構 (JST) 【さきがけ】 (嶋 盛吾) からの研究費助成を受けた。マックスプランク陸生微生物学研究所ロルフ=タウアー教授の援助に感謝する。
1) Thauer, R. K., Kaster, A. K., Goenrich, M., Schick, M., Hiromoto, T., Shima, S.: Annu. Rev. Biochem., 79, 507 (2010).
2) Afting, C., Hochheimer, A., Thauer, R. K.: Arch. Microbiol., 169, 206 (1998).
3) Shima, S., Ermler, U.: Eur. J. Inorg. Chem., 2011, 963 (2011).
4) Hiromoto, T., Ataka, K., Pilak, O., Vogt, S., Stagni, M. S., Meyer-Klaucke, W., Warkentin, E., Thauer, R. K., Shima, S., Ermler, U.: FEBS Lett., 583, 585 (2009).
5) Shima, S., Pilak, O., Vogt, S., Schick, M., Stagni, M. S., Meyer-Klaucke, W., Warkentin, E., Thauer, R. K., Ermler, U.: Science, 321, 572 (2008).
6) Lyon, E. J., Shima, S., Buurman, G., Chowdhuri, S., Batschauer, A., Steinbach, K., Thauer, R. K.: Eur. J. Biochem., 271, 195 (2004).
7) Shima, S., Schick, M., Ataka, K., Steinbach, K., Linne, U.: Dalton Trans., 41, 767 (2012).
8) Shima, S., Lyon, E. J., Sordel-Klippert, M. S., Kauss, M., Kahnt, J., Thauer, R. K., Steinbach, K., Xie, X. L., Verdier, L., Griesinger, C.: Angew. Chem. Int. Ed., 43, 2547 (2004).
9) Lyon, E. J., Shima, S., Boecher, R., Thauer, R. K., Grevels, F. W., Bill, E., Roseboom, W., Albracht, S. P. J.: J . Am. Chem. Soc., 126, 14239 (2004).
10) Korbas, M., Vogt, S., Meyer-Klaucke, W., Bill, E., Lyon, E. J., Thauer, R. K., Shima, S.: J. Biol. Chem., 281, 30804 (2006).
11) Lyon, E. J., Shima, S., Boecher, R., Thauer, R. K., Grevels, F. W., Bill, E., Roseboom, W., Albracht, S. P.: J. Am. Chem. Soc., 126, 14239 (2004).
12) Fujishiro, T., Tamura, H., Schick, M., Kahnt, J., Xie, X. L., Ermler, U., Shima, S.: Angew. Chem. Int. Ed., 52, 12555 (2013).
13) Fujishiro, T., Bai, L., Xu, T., Xie, X., Schick, M., Kahnt, K., Rother, M., Hu, X., Ermler, U., Shima, S.: Angew. Chem. Int. Ed.,55, 9648 (2016).
14) Fujishiro, T., Ermler, U., Shima, S.: FEBS Lett., 588, 2789 (2014).
15) Fujishiro, T., Kahnt, J., Ermler, U., Shima, S.: Nat. Commun., 6, 6895 (2015).