【トピックス】
ラクト-N-ビオシダーゼ (LnbX) の機能から考える母乳オリゴ糖を介したビフィズス菌と母乳栄養児の共生・共進化
後藤愛那、片山高嶺、山田千早、伏信進矢、櫻間晴子
京大院・生命、京大院・生命、東大院・農生、東大院・農生、京都学園大・バイオ
1.はじめに
腸内細菌とヒト (宿主) の相互作用については近年急速に研究が進み、同時に社会的関心も高まっている。多種多様なヒト腸内細菌のバランス (腸内細菌叢) は、宿主自身によって調節される一方で、逆に宿主の健康状態や疾病にも大きな影響を与え、最近ではヒトの健康にとって良い細菌叢や悪い細菌叢、あるいは良い菌や悪い菌が徐々に明らかとなってきた1,2)。ヒトが腸内細菌に与えているものは、嫌気的閉鎖環境、食餌由来成分、ムチン糖タンパク質および免疫関連物質などがあり3,4)、これらが腸内細菌叢の維持や調節を担う因子であると考えられるが、詳細なメカニズムについてはいまだ不明な点が多く残されている。
ヒトの生涯において腸内細菌叢が最も大きく変化するのは乳児期である。新生児腸管内では、母乳を飲み始めて数週間のうちにビフィズス菌が9割前後を占める“ビフィズスフローラ”が形成されることが分かっている (成人のビフィズス菌占有率は1割前後) 。世界保健機構 (WHO) が母乳育児を推奨していることや、ビフィズス菌が成人にとっても有益な菌であることを示す研究結果も数多くあることから、母乳栄養児腸管内におけるビフィズスフローラ形成は、乳児の健康的な成長と発達に重要であると考えられる5)。そのため、母乳中に含まれる「何が」ビフィズス菌を特異的に増殖させているかは、非常に興味深い研究課題であり、我々は母乳中に含まれるオリゴ糖HMOs (human milk oligosaccharides) と、それを分解資化するビフィズス菌の特徴的な酵素の関係からそれを理解することを目指してきた6)。
HMOsは母乳中に10〜20 g/L含まれ、ラクトースおよび脂質に継いで3番目に多い固形成分であるが、ヒトの消化酵素に耐性を持っているため分解吸収されず、乳児自身の栄養にはならない7-9)。HMOsはこれまでに100種以上の構造が報告されているが、哺乳類の中でもヒトに特徴的な構造はラクト-N-ビオースI (LNB, Galβ1-3GlcNAc) 構造である。実際の母乳中では、LNBそのものは含まれておらず、ラクトース (Lac, Galβ1-4Glc) の非還元末端側にβ-1,3結合した4糖の基本骨格ラクト-N-テトラオース (LNT, Galβ1-3GlcNAcβ1-3Galβ1-4Glc) として、またLNTにフコース (Fuc) やシアル酸 (Neu5Ac) が付加した構造として含まれている。これらLNB構造を末端に持つオリゴ糖はI型HMOsと総称され、上述した通りヒトに特徴的な構造である10,11)。
乳児腸管内においてはBifidobacterium属の中でも、B. breve、B. bifidum、B. longum subsp. longum (以降、B. longumとして表記) 、B. longum subsp. infantisの4種が優占種として知られる12,13)。我々は、これら乳児型ビフィズス菌のI型HMOs分解酵素に特に注目して解析を行ってきた。その過程で、乳児腸管という共通のニッチに適応したこれら4種のビフィズス菌が、それぞれまったく異なる戦略によってHMOsを資化することを明らかとした6)。
前述した通り、LNBは基本骨格であるLNTの一部として含まれているわけであるが、4種の乳児型ビフィズス菌はLNBを単一炭素源とした培地中で生育することが可能である。その資化過程では、LNBは二糖特異的トランスポーターで細胞内に取り込まれた後、二糖特異的な細胞内酵素 (ホスホリラーゼ) で分解される14,15)。この二糖特異的トランスポーターと二糖特異的ホスホリラーゼがビフィズス菌に極めて特異的であることから、LNBは乳児型ビフィズス菌4種に特化したビフィズス因子であると考えられた16)。またこの事実は、LNTをLNBとLacに分解する酵素の存在を示唆していることから、我々はまずB. bifidumから菌体外酵素ラクト-N-ビオシダーゼ (LNBase) を単離した17)。B. bifidumはLNBase以外にもHMOsからFucやNeu5Acを遊離する1,2-α-L-フコシダーゼ、1,3-1,4-α-L-フコシダーゼ、シアリダーゼを有しており18-20)、これら菌体外酵素で分解されたHMOs分解産物は、B. bifidum自身だけでなく他の3種のビフィズス菌によっても利用されるチャンスを得ることになる。特に、LNBが優れたビフィズ因子であるという事実を考慮すると、ここで発見された「菌体外でLNTからLNBを遊離するLNBase」は、ビフィズス菌種間をまたぐビフィズスフローラ形成における重要な鍵となる酵素であると考えられる。
ところで、LNBase活性はB. bifidumだけでなくB. longumにも検出されるが、B. bifidum由来のLNBase (LnbBと命名) の配列を使用して相同性検索しても、B. longumのゲノム上にはホモログが存在しなかった21)。 LnbBは糖質加水分解酵素ファミリー20 (GH20) に属しているが、 B. longumゲノム上にあるGH20ホモログにはLNBase活性は確認できなかったのである22)。そのため、B. longumはLnbBとは全く異なる酵素遺伝子を有している可能性が示唆された。本トピックスでは、B. longum由来の新規なLNBaseの遺伝子単離から構造機能解析、また乳児糞便解析を通した生理機能評価までの一連の研究成果を報告する。
2.LnbXの遺伝子クローニングと性状解析
2-1 ラクト-N-ビオシダーゼ遺伝子の同定23)
B. longum JCM1217株のゲノムライブラリーを作製し、p-ニトロフェニル-β-ラクト-N-ビオシドI (LNB-β-pNP) の加水分解活性を指標にしたスクリーニングを行った。その結果、陽性クローンには機能未知の2つのORF (BLLJ_1505ならびにBLLJ_1506) が共通して存在していた (図1)。以降、BLLJ_1505をLnbX、BLLJ_1506をLnbYと表す。
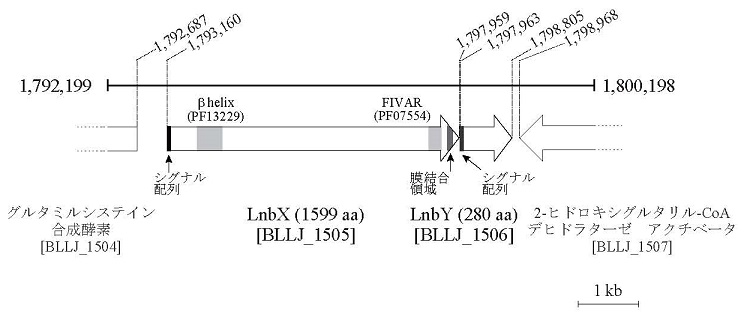
図1 ラクト- N -ビオシダーゼ(lnbXY)の遺伝子構造。B. longum JCM1217のゲノム配列をもとに表記 (文献23より改変)
次に、B. longumにおけるlnbX遺伝子破壊株を相同組換えにより作製したところ、予想通りLNBase活性が失われた。相補実験を行ったところ、lnbXやlnbY遺伝子を単独で導入しても活性は回復しなかったが、lnbXY両遺伝子を導入すると活性が回復した。これらのことから、B. longumのLNBaseはlnbXY遺伝子で構成されることが明らかとなった。
2-2 組換え酵素の精製と諸性質の決定
B. longumのLNBaseは細胞表層に発現する分泌型酵素である。lnbXならびにlnbYのシグナル配列と膜結合領域を除いた領域を大腸菌でそれぞれ単独で発現させた場合には活性が検出されなかったが、共発現させた場合には高い活性が検出された。LnbXとLnbYを共発現させた大腸菌からLNBaseを精製したところ、LnbXのみとなった。これらの結果から、酵素の本体はLnbXとLnbYのヘテロ2量体ではなく、LnbX単独であることが強く示唆された (後述)。また、本酵素は二量体構造であり、Mg2+およびCa2+を含有していた。
2-3 基質特異性の解析
本酵素の基質特異性を、B. bifidum由来のLnbBと比較した (表1)。その結果、本酵素は非還元末端側のLNB構造を厳密に認識するLnbB と異なりLNB構造のGal部分の認識にある程度の寛容さを有しており、GalにFucがα- (1→2) 結合したラクト-N-フコペンタオース I (LNFP I) やNeu5Acがα- (2→3) 結合したシアリルラクト-N-テトラオースa (LSTa) に対しても作用した。しかしLnbB と同様、N-アセチルグルコサミン (GlcNAc) にFucやNeu5Acが結合したラクト-N-フコペンタオースIIやシアリルラクト-N-テトラオースb (LSTb) には全く作用しなかった。一方で、どちらの酵素もラクトース部分から枝分かれしているラクト-N-ヘキサオース (LNH) に対しては作用した。LNB構造を持たないラクト-N-ネオテトラオースや非還元末端側にGalを持たないラクト-N-トリオースIIには作用しなかった。興味深いことに、本酵素はLNB-β-pNPと比べても遜色のない程度に、GalNAcβ1-3GlcNAc-β-pNPを加水分解する活性を示した。我々の知る限り、このような二糖構造を遊離できる酵素というのはこれまでに知られていない。この二糖構造はα-ジストログリカンのラミニン結合のリガンドの一つであることが知られていることから、α-ジストログリカンのO-マンノシル型糖鎖などの構造解析において有用なツールとして役立つ可能性がある。
表1 ラクト-N-ビオシダーゼの基質特異性

本酵素の速度論的パラメータを算出したところ、LNFP Iに対する触媒効率はLNTより300倍低い値を示したことから、本酵素の生理的な機能は専らLNTの加水分解であることが示唆された (Km and kcat: 0.40 mM and 110 s-1 for LNT; 15 mM and 14 s-1 for LNFP I)。これは、B. longumをHMOs培地で培養した際に主にLNTのみを利用することと一致した。
2-4 LnbYのLnbXシャペロンとしての役割
His-tag付LnbXを単独発現する大腸菌と、His-tag付LnbXとLnbYを共発現する大腸菌の無細胞抽出液を用いてゲル濾過分析を行った (図2)。その結果、共発現させた方では分子サイズ356 kDa付近にLnbXが溶出され酵素活性も検出されたが、単独発現させた方では酵素活性は検出されず、LnbXタンパク質はボイド画分に可溶性封入体として溶出された。次にグアニジン塩酸塩で変性させたLnbXを用いたin vitroのリフォールディング実験を行った。変性LnbXはLnbYの存在下で活性が回復した (図3a)。また金属イオン (Ca2+,Mg2+) を同時に加えるとより回復率が上昇した。金属イオン濃度を0.1 mM、変性LnbXに対するLnbYの量を20倍とした時に回復率が最大となった (図3b)。これらのことから、LnbYはLnbXのシャペロンであること、またリフォールディングには金属イオンも必要であることが示された。
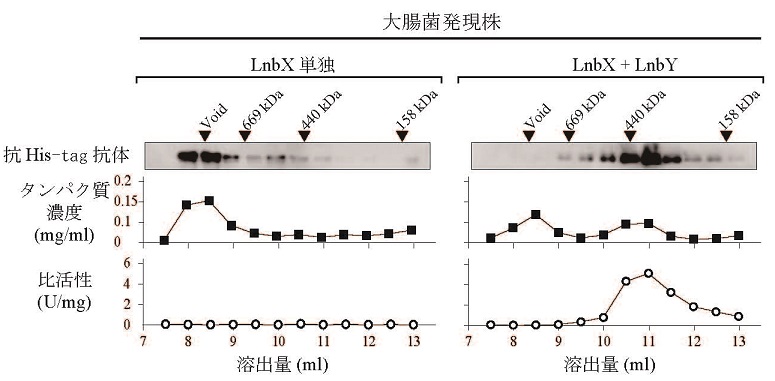
図2 ゲル濾過によるLnbXのフォールディング状態の解析と活性測定 (文献23より改変)
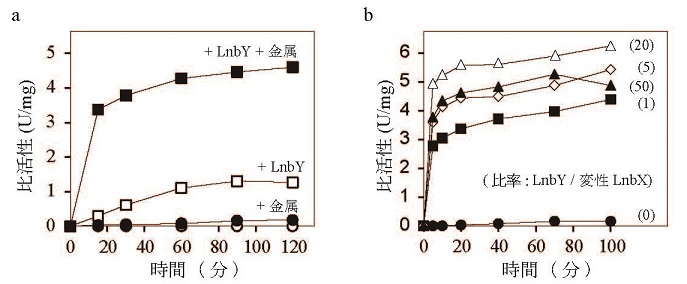
![]()
3.LnbXの構造解析24)
3-1 LnbX触媒ドメイン (31-625) のX線結晶構造解析
結晶化に先立ち、LnbXのN末端およびC末端を様々な長さに欠失した変異体を作製して比活性を測定した。その結果、高い比活性を保つ最小単位 (触媒ドメイン) としてLnbX (31-625) を同定した。この変異体のC 末端にヒスチジンタグ配列 (His-tag) を付け、シャペロンタンパク質LnbY (30-280) にはHis-tagを付加せず、複製起点の異なる発現ベクターに組み込んで、大腸菌内で共発現させた。触媒ドメインはニッケルカラムおよび他の2 種類のカラムクロマトグラフィーを用いて精製した。精製酵素を用いて結晶化条件をスクリーニングした結果、フッ化アンモニウムとポリエチレングリコール3350、MES-NaOHバッファー (pH 6.0) 存在下で良質な結晶が得られた。高エネルギー加速器研究機構放射光科学研究施設およびSPring-8でX 線回折実験を行い、位相決定はセレノメチオニンを用いた単波長異常分散法で行った。その結果、リガンドフリーおよびLNB複合体構造をそれぞれ1.82 Åおよび2.36 Åで決定することに成功した。LnbXの触媒ドメインは図4に示すようにオリゴ糖分解酵素ではめずらしいβ-へリックス構造を有しており、LNBの結合による構造変化はみられなかった。LnbX触媒ドメインのホモログはビフィズス菌以外の数種のヒト腸内細菌や、ヒト以外の哺乳動物から単離されたビフィズス菌にも見られた。以上の結果から、LnbXは新規な糖質加水分解酵素ファミリー (GH136) として登録された (http://www.cazy.org/GH136.html)。
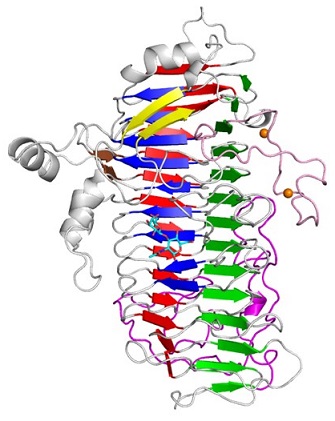
図4 LnbX触媒ドメイン (31-625) のLNB複合体構造。LNBはシアンで示した (文献24より改変)
3-2 LnbXとLnbBの構造比較
LnbX触媒ドメインはβ-へリックス構造を有する一方で、上述したB. bifidum由来のLnbBは (β/α)8バレル構造をとっている。基質結合ポケットを比較したところ、LnbBの活性ポケットにはFucやNeu5Acが結合したLNB構造を受け入るスペースがないが、LnbXの活性ポケットでは-2サブサイトに結合したGalのO2およびO3位が溶媒側に露出していることが明らかとなった (図5)。このことは、LnbXがLNFP IやLST aに作用可能なことを構造上うまく説明していた。LnbBは基質のGlcNAcのN-アセチル基が求核触媒残基となる基質補助型のグリコシダーゼであることが分かっているが、LnbXは一般的なグリコシダーゼと同様に2つの酸性残基を触媒残基として有し、そのうち酸塩基触媒は水を介してプロトンを授受するGrotthus機構をとることが示唆された。以上のことから、LNTの加水分解という全く同じ反応を担う2つの酵素は全体構造および反応機構も大きく異なっていることが明らかとなった。同じ環境に生息し、HMOsという同じ基質による選択圧を受けながらも、それぞれのビフィズス菌が2つの酵素を別々に進化させてきたことは非常に興味深い。
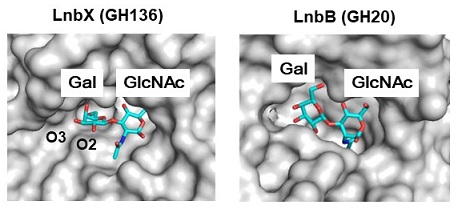
図5 LnbX (GH136) とLnbB (GH20) の基質結合ポケットの比較 (文献24より改変)
4.乳児の糞便解析24)
乳児腸管内でのビフィズス菌によるHMOs資化におけるLnbXの重要性と寄与を理解することを目的に、助産院の協力を得て月齢1〜6ヶ月の健康な乳児16名の糞便を回収した。16名中の10名は完全母乳栄養児で、他6名は母乳と人工乳の混合栄養児であった。各糞便サンプルから腸内細菌DNAを抽出し、定量PCRによりBifidobacteirum (属レベル)、B. longum (種レベル)、lnbX遺伝子それぞれのコピー数を定量した。属レベルではBifidobacterium属が完全母乳栄養児群および混合栄養児群いずれの群糞便中でも検出された。しかし、群間で比較すると、混合栄養児群より完全母乳栄養児群で有意にBifidobacterium属の占有率が高いことがわかった (図6a)。種レベルで解析すると、B. longum以外の種では両群に差がないものの、B. longumの占有率に有意な差が検出された (図6b)。lnbX遺伝子は、完全母乳栄養児群では5名 (50%) で検出されたが、混合栄養児群では1名 (17%) でのみ検出された。lnbX遺伝子の占有率も母乳栄養児群で有意に高い結果となった (図6c)。さらに、lnbX遺伝子とB. longumの占有率の相関を検討したところ、完全母乳栄養児群において有意ではないものの正の相関 (ピアソンの相関係数 r = 0.62、p = 0.058) が見られた。これとは対照的に、混合栄養児群では相関は全く見られなかった。なお、lnbX遺伝子は全てのB. longum株が有しているわけではなく、データベース上では半数程度のB. longumゲノム上にしか見られない。これらの結果は、HMOsがビフィズスフローラ形成に重要な役割を果たしていることを示すと同時に、1型HMOsがビフィズス菌分解酵素 (少なくともlnbX遺伝子) の選択圧となっていることを示唆しており、HMOsを介したヒトとビフィズス菌の共生・共進化を考える上で非常に興味深い。

![]()
5.おわりに
乳児腸管内におけるビフィズスフローラ形成について、特にI型HMOsとその分解酵素LNBaseに着目した研究を紹介した。しかしLNBase以外にも、ビフィズス菌の持つ特徴的なHMOs資化関連遺伝子が他グループによって報告されている。そのひとつが、LacにFucが付加したフコシルラクトース (FL) 利用に関連する遺伝子である。すでに報告されている通り、I型HMOsに次いでFLも主要なHMO分子種であり、特に2’-FL (Fucα1-2Galβ1-4Glc) と3-FL (Galβ1-4 (Fucα1-3) Glc) が母乳中には多く含まれている25)。
2015年にMatsukiらは、27名の乳児について生後1日から29日までの経時的な腸内細菌叢変化を分析し、ビフィズスフローラが形成される時期には乳児間で差があることを報告した26)。この差は乳児糞便中のHMOsの減少率と相関しており、単離した菌株においてFLの資化が見られた。さらに筆者らは単離したビフィズス菌のゲノム解析と合わせ、B. breveが持つFLトランスポーター遺伝子を見出した。さらに2016年の報告では、乳児由来 B. longum のHMOs資化能力は株ごとにバリエーションがあることが示された27)。グライコーム解析からB. longumがLNTを有効に利用するという点は共通していたが、一部の株がFLも効率的に資化することが明らかとなった。このFLの利用は、上記と同じFLトランスポーターと1,2-α-L-フコシダーゼ遺伝子によることが示された。
以上の2報によって示唆されたFLとFLトランスポーターの関連、I型HMOsとLNBaseの関連は、いずれもビフィズスフローラ形成において重要である点が共通している。しかしながら、FLトランスポーターは保有株をニッチにおける生存競争において有利にするのみである一方、LNBase発現株はビフィズス菌種間のクロスフィーディングというさらに複雑な関係性を形作る可能性がある28)。また、FLの利用性には菌種間や株間で違いがあるが、LNTの利用性は乳児型ビフィズス菌に普遍的である。実際の乳児腸管内、あるいは複数のビフィズス菌株が混在する際のHMOs分子の取り合いや分配の詳細についてはまだほとんど解析が進んでいないのが現状であり、今後より実際の環境に近い状態でのHMOs分子の挙動を解析することを通し、ビフィズスフローラ形成機構がより深く理解されることが期待される。
謝辞
乳児糞便は長尾助産院 (京都府向日市) にて回収しました。サンプル採取にご協力下さいました廣瀬潤子先生 (滋賀県大)、長尾早枝子先生 (長尾助産院) およびお母様方とそのお子様方に感謝いたします。また、本研究に対して有益なご助言を下さいました北岡本光先生 (農研機構) に深謝します。本研究の一部は発酵研究所のご寄付によって開設された石川県立大学腸内細菌共生機構学分野において行われ、特任助教の阪中幹祥先生にご協力を頂きました。
文献
1) Kau, A. L., Ahern, P. P., Griffin, N. W., Goodman, A. L., Gordon, J. I.: Nature, 474, 327 (2011).
2) Turnbaugh, P. J., Ley, R. E., Mahowald, M. A., Magrini, V., Mardis, E. R., Gordon, J. I.: Nature, 444, 1027 (2006).
3) David, L. A., Maurice, C. F., Carmody, R. N., Gootenberg, D. B., Button, J. E., Wolfe, B. E., Ling, A. V., Devlin, A. S., Varma, Y., Fischbach, M. A., Biddinger, S. B., Dutton, R. J., Turnbaugh, P. J.: Nature, 505, 559 (2014).
4) Okai, S., Usui, F., Yokota, S., Hori-I, Y., Hasegawa, M., Nakamura, T., Kurosawa, M., Okada, S., Yamamoto, K., Nishiyama, E., Mori, H., Yamada, T., Kurokawa, K., Matsumoto, S., Nanno, M., Naito, T., Watanabe, Y., Kato, T., Miyauchi, E., Ohno, H., Shinkura, R.: Nat. Microbiol., 1, 16103 (2016).
5) Olszak, T., An, D., Zeissig, S., Pinilla Vera, M., Richter, J., Franke, A., Glickman, J. N., Siebert, R., Baron, R. M., Kasper, D. L., Blumberg, R. S. : Science, 336, 489 (2012).
6) Katayama, T.: Biosci. Biotechnol. Biochem., 80, 621 (2016).
7) Brand-Miller, J. C., McVeagh, P., McNeil, Y., Messer, M.: J. Pediatr., 133, 95 (1998).
8) Gnoth, M. J., Kunz, C., Kinne-Saffran, E., Rudloff, S.: J. Nutr., 130, 3014 (2000).
9) Kunz, C., Rudloff, S., Baier, W., Klein, N., Strobel, S.: Annu. Rev. Nutr., 20, 699 (2000).
10) Kobata, A.: Proc. Jpn. Acad. Ser. B. Phys. Biol. Sci., 86, 731 (2010).
11) Urashima, T., Asakuma, S., Leo, F., Fukuda, K., Messer, M., Oftedal, O. T.: Adv. Nutr., 3, 473S (2012).
12) Mitsuoka, T.: Asian Med. J., 31, 400 (1988).
13) Sakata, S., Tonooka, T., Ishizeki, S., Takada, M., Sakamoto, M., Fukuyama, M., Benno, Y.: FEMS Microbiol. Lett., 243, 417 (2005).
14) Kitaoka, M., Tian, J., Nishimoto, M.: Appl. Environ. Microbiol., 71, 3158 (2005).
15) Suzuki, R., Wada, J., Katayama, T., Fushinobu, S., Wakagi, T., Shoun, H., Sugimoto, H., Tanaka, A., Kumagai, H., Ashida, H., Kitaoka, M., Yamamoto, K.: J. Biol. Chem., 283, 13165 (2008).
16) Xiao, J. Z., Takahashi, S., Nishimoto, M., Odamaki, T., Yaeshima, T., Iwatsuki, K., Kitaoka, M.: Appl. Environ. Microbiol., 76, 54 (2010).
17) Wada, J., Ando, T., Kiyohara, M., Ashida, H., Kitaoka, M., Yamaguchi, M., Kumagai, H., Katayama, T., Yamamoto, K.: Appl. Environ. Microbiol., 74, 3996 (2008).
18) Katayama, T., Sakuma, A., Kimura, T., Makimura, Y., Hiratake, J., Sakata, K., Yamanoi, T., Kumagai, H., Yamamoto, K.: J. Bacteriol., 186, 4885 (2004).
19) Kiyohara, M., Tanigawa, K., Chaiwangsri, T., Katayama, T., Ashida, H., Yamamoto, K.: Glycobiol., 21, 437 (2011).
20) Sakurama, H., Fushinobu, S., Hidaka, M., Yoshida, E., Honda, Y., Ashida, H., Kitaoka, M., Kumagai, H., Yamamoto, K., Katayama, T.: J. Biol. Chem., 287, 16709 (2012).
21) Fukuda, S., Toh, H., Hase, K., Oshima, K., Nakanishi, Y., Yoshimura, K., Tobe, T., Clarke, J. M., Topping, D. L., Suzuki, T., Taylor, T. D., Itoh, K., Kikuchi, J., Morita, H., Hattori, M., Ohno, H.: Nature, 469, 543 (2011).
22) Honda, Y., Nishimoto, M., Katayama, T., Kitaoka, M.: J. Appl. Glycosci., 60, 141 (2013).
23) Sakurama, H., Kiyohara, M., Wada, J., Honda, Y., Yamaguchi, M., Fukiya, S., Yokota, A., Ashida, H., Kumagai, H., Kitaoka, M., Yamamoto, K., Katayama, T.: J. Biol. Chem., 288, 25194 (2013).
24) Yamada, C., Gotoh, A., Sakanaka, M., Hattie, M., Stubbs, K. A., Katayama-Ikegami, A., Hirose, J., Kurihara, S., Arakawa, T., Kitaoka, M., Okuda, S., Katayama, T., Fushinobu, S.: Cell Chem. Biol., 24, 515-524 (2017).
25) Asakuma, S., Hatakeyama, E., Urashima, T., Yoshida, E., Katayama, T., Yamamoto, K., Kumagai, H., Ashida, H., Hirose, J., Kitaoka, M.: J. Biol. Chem., 286, 34583 (2011).
26) Matsuki, T., Yahagi, K., Mori, H., Matsumoto, H., Hara, T., Tajima, S., Ogawa, E., Kodama, H., Yamamoto, K., Yamada, T., Matsumoto, S., Kurokawa, K.: Nat. Commun., 7, 11939 (2016).
27) Garrido, D., Ruiz-Moyano, S., Kirmiz, N., Davis, J. C., Totten, S. M., Lemay, D. G., Ugalde, J. A., German, J. B., Lebrilla, C. B., Mills, D. A.: Sci. Rep., 6, 35045 (2016).
28) Tannock, G. W., Lawley, B., Munro, K., Gowri Pathmanathan, S., Zhou, S. J., Makrides, M., Gibson, R. A., Sullivan, T., Prosser, C. G., Lowry, D., Hodgkinson, A. J.: Appl. Environ. Microbiol., 79, 3040 (2013).