【トピックス】
Nonribosomal peptide synthetase における内部アデニレーションドメインを用いたジペプチド合成
安部智子、小林逹彦
東京電機大・理工、筑波大院・生命環境
1.はじめに
機能性オリゴペプチドの有益性は製薬業界や健康食品、化粧品業界など多様な分野で研究されており1-3)、また、オリゴペプチドを利用することは従来のアミノ酸利用を拡大させる可能性や新たなアミノ酸用途を生む可能性をもつ。ゆえに新たな酵素法によるオリゴペプチド合成法が注目されている4,5)。本トピックスでは、我々が提案した、nonribosomal peptide synthetases (NRPS) の1つで複数のドメインからなるマルチドメイン酵素内の “internal (内部の) ” アデニレーションドメインのみを用いたオリゴペプチド合成法を紹介する。
2.アデニレーションドメインの働き
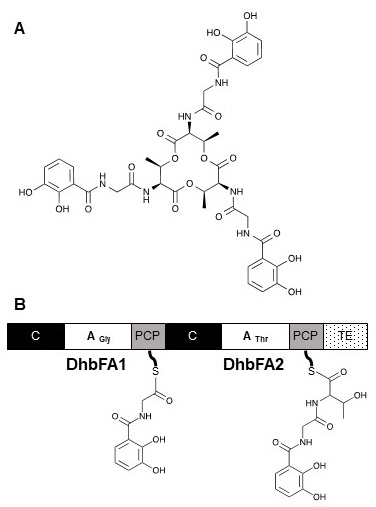
バシリバクチン (2,3-dihydroxybenzoate-glycine-l-threonine trimeric ester) (図1A) は鉄が乏しい環境下で鉄イオンを集めるためにバクテリアによって合成されるシデロフォアと呼ばれる化合物である6,7)。バシリバクチンはnonribosomal peptide synthetases (NRPSs) 8) によって合成される。NRPSはバクテリアの二次代謝産物の、主に小さなペプチドの合成に関与しており、これらのペプチドのいくつかは、抗生物質や抗ウイルス薬、免疫抑制剤、細胞分裂阻害剤などとしての利用が期待されている9,10)。一般的なNRPSは、基質となるアミノ酸を選択して活性化し、アミノ酸同士を結合してペプチドを組み立てるラインとして機能するタンパク質ユニットのいくつかのセットからなる。はじめにアデニレーションドメインがaminoacyl-O-AMPを合成し、その後、peptidyl carrier protein (PCP) ドメインがもつ4’-phosphopantetheine末端のチオール基へアミノ酸を共有結合させる。Condensationドメインはペプチド鎖伸長の過程でペプチド結合形成を触媒する。バシリバクチンの生合成においてDhbF (図1B) というマルチドメイン酵素に含まれる2つのアデニレーションドメインは、それぞれグリシンまたは L-スレオニンを活性化し、PCPドメインに結合した4’-phosphopantetheineに結合させる役割をもつ11)。本研究では、このDhbFがもつ2つの内部アデニレーションドメインをDhbFA1あるいはDhbFA2と命名し、実験に用いた。また、NRPSのアデニレーションドメインはsuperfamily of adenylate-forming enzymeと呼ばれるスーパーファミリーに属しており、このスーパーファミリーには、アシルCoA合成酵素やアセチルCoA合成酵素、リグニンの形成に関与する4-クマル酸:CoAリガーゼ、さらにはホタルルシフェラーゼ等が属している12-14)。

3.内部アデニレーションドメイン単体での発現および精製
DhbFの内部のアデニレーションドメインだけを発現させて調べた報告はなかったため、これらが機能するために必要な遺伝子領域は不明であった。そのため、adenylate-forming enzymeのスーパーファミリーに属する他の酵素や、独立型のアデニレーションドメインのアミノ酸配列と共に DhbFのアミノ酸配列を用いてシーケンスアライメントを行い、それをもとにDhbFの内部アデニレーションドメインだけを発現させるための領域を決定した。その領域のDNAをPCRで増幅した後に、コールドショック発現ベクターに組み込み、大腸菌を用いての大量発現および遺伝子産物の精製を検討した結果、DhbFA1あるいはDhbFA2のドメインのみのタンパク質を得ることに初めて成功した (図2)。


4.内部アデニレーションドメインを用いたジペプチド合成
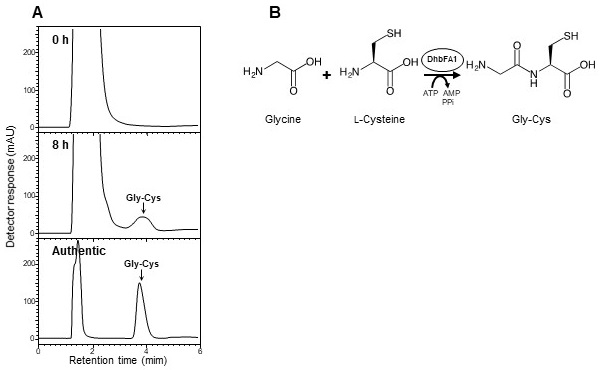
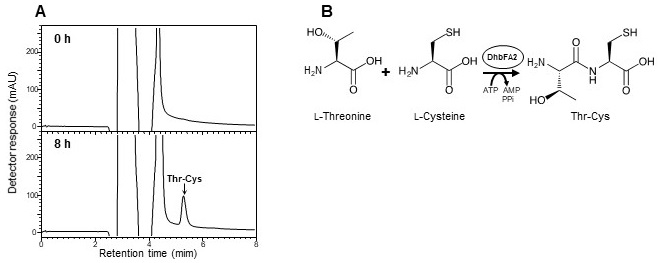
バシリバクチンの生合成においては、DhbFA1はATPを用いてグリシンを活性化し、PCPドメインに受け渡す役割をもつドメインである。単独精製したDhbFA1にグリシンと、PCPドメインの代わりに L-システインを基質として加えて反応させたところ、反応の進行を示すAMP量の増加が確認され、また、AMP以外の産物のピークもHPLC分析 (CROWNPAK CR (+) column, 4.0×150 mm, 移動相溶媒 過塩素酸pH1.5) により確認された (図3A)。この産物は、分取・精製後、質量分析によって、分子量が178であることがわかった。この分子量はグリシンとL-システインが脱水して結合した分子量と一致するが、分子量から予想される反応産物として3つの化合物 (グリシンのカルボキシル基とL-システインのチオール基がチオエステル結合した化合物S-glycyl-l-cysteine、グリシンのカルボキシル基とL-システインのアミノ基がペプチド結合したジペプチドGly-Cys、あるいはその逆のL-システインのカルボキシル基とグリシンのアミノ基がペプチド結合したジペプチドCys-Gly) が考えられたため、LC-MS/MSによる産物の同定を行った。その結果、グリシンのカルボキシル基とL-システインのアミノ基がペプチド結合したGly-Cysにしか確認されないフラグメンテーション (m/z 99) が検出され、内部アデニレーションドメインであるDhbFA1がグリシンとL-システインを結合してジペプチドを合成することを初めて明らかにした (図3B)。同様に、単独精製したDhbFA2にL-スレオニンとL-システインを基質として加えて反応させた場合にも、L-スレオニンとL-システインが脱水縮合した化合物と同じ分子量をもつ産物のピークがHPLC分析 (Develosil RPAQUEOUS column, 4.6×250 mm, 移動相溶媒5%アセトニトリル含有0.1%ギ酸) で確認された (図4A)。LC-MS/MS 分析の結果、L-スレオニンのカルボキシル基とL-システインのアミノ基がペプチド結合したThr-Cysにしか確認されないフラグメンテーション (m/z 143) が検出され、DhbFA2もDhbFA1同様にジペプチドを合成することを明らかにした (図4B)。またDhbFA1では、グリシンの代わりにL-セリン、あるいはL-アスパラギン酸とL-システインを基質として用いた場合にも活性が確認され、Ser-Cysおよび Asp-Cysの生成が示唆された。


ジペプチド合成におけるDhbFA1あるいはDhbFA2のL-システインに対するKm値を解析したところ、DhbFA1のL-システインに対するKm 値は22mMと高かったが、DhbFA2のL-システインに対するKm 値は0.016mMと低いことがわかった。基質アミノ酸に対する親和性が高いことは、工業的なペプチド生産において重要である。
5.オリゴペプチド合成とその機構
DhbFA2はThr-Cysの1種類のジペプチドしか合成できなかったが、DhbFA1はGly-Cys以外にもL-セリンやL-アスパラギン酸を含むSer-CysやAsp-Cysを合成できることが示された。さらに、DhbFA1もDhbFA2も、L-システインの代わりの基質としてN末端にL-システインをもつオリゴペプチド (Cys-GlyやCys-Gly-Gly-Arg-Glu、Cys-Gly-Gly-Arg-Glu-Ser-Gly-Ser-Gly-Ser) を用いた場合にトリペプチドやヘキサペプチド、ウンデカペプチドなどのオリゴペプチドを合成できることが示された。これらのオリゴペプチドの合成活性は、L-システインを基質としたジペプチドの合成活性とあまり変わらなかった。また、D-システインも L-システインと同様に基質になりうることがわかった。これらのことから、N末端がシステインであれば、どのようなアミノ酸配列のペプチドでも基質にすることができると考えられる。
近年、アデニレーションドメインと同じくadenylate-forming enzymeのスーパーファミリーに属するD-alanine:D-alanyl carrier protein ligaseであるDltAという酵素もペプチドを合成できることを我々は明らかにし、そのメカニズムを提唱するまでに至った16-18)。DhbFA1とDhbFA2もadenylate-forming enzymeのスーパーファミリーに属する酵素であるため、DltAのペプチド合成機構をもとに我々が提案するDhbFA1あるいはDhbFA2が関与する新規ペプチド合成反応の機構は次のようなものである。 (i) 本来のアデニレーションドメインの活性 (adenylation) によりS-aminoacyl-cysteineが中間体として形成され、続いて (ii) 化学的なS→Nアシル転移反応によりN-aminoacyl-cysteine (ペプチド) が合成される (図5)。つまり、酵素的な反応と化学的な反応の二段階の連続した反応を経るペプチド結合の形成である。DhbFA1とDhbFA2が上記のメカニズムでペプチドを合成することは、DhbFA1とDhbFA2のどちらの酵素でも、システインの代わりにアミノ基を持たずにチオール基のみをもつシステインアナログである 3-メルカプトプロピオン酸を基質として用いた場合には反応が進み、チオール基を持たないL-アラニンやL-セリンなどのアミノ酸を基質に用いた場合、あるいはS-methyl-cysteineなどの保護されたチオール基をもつ化合物を基質に用いた場合には反応が進まなかったことからも考察される。反応には遊離のチオール基が必要であり、カルボキシル基が直接システインのアミノ基と結合する訳ではないことを示す。この反応メカニズムはNative chemical ligation (NCL) 法19,20) と呼ばれるペプチドの化学合成法の反応機構 (可逆的なチオール-チオエステル交換反応の後に続いてS→Nアシル転移反応が起こる) と類似した点があるが、我々が提案する方法の第1段階の反応は酵素反応であるため、NCL法では必要な基質のチオエステル化が不要という利点がある。しかしながら、第2段階目の反応はNCL法と同様であるため、NCL法の応用反応を参考にオリゴペプチド同士を連結する反応や、連結部をシステイン以外のアミノ酸に変換する方法21)などを現在、検討中である。
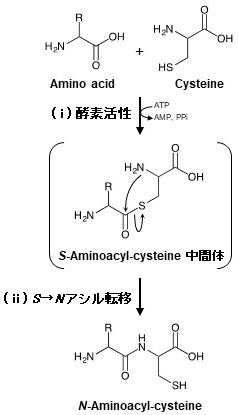
図5 アデニレーションドメインによる新規ペプチド合成反応機構
6.おわりに
本研究で我々はNRPSのマルチドメイン酵素内の内部アデニレーションドメインを用いてもペプチドを合成できることを証明し22)、これはadenylate-forming enzymesのスーパーファミリーに含まれる酵素全てが普遍的にもつ活性であることを強く示唆するものである。本スーパーファミリーには様々な基質特異性をもった酵素が含まれるため、本スーパーファミリーに属する酵素を用いることで有用なペプチドを容易に合成することができるだろう。
謝辞
本研究は、東京電機大学理工学部生命理工学系 環境微生物学研究室を中心に行われた研究の成果であり、研究室の皆様に心から感謝いたします。また、本研究を行うにあたり、Bacillus subtilis strain168の染色体DNAを分与頂きました筑波大学 中村顕先生、研究に関してご助言を頂きました筑波大学 橋本義輝先生、東京電機大学 山名昌男先生に感謝申し上げます。本研究は文部科学省からの科学研究補助金の助成の一部を受けたものです。また、東京電機大学総合研究所研究 (Q17L-02) として行ったものです。
文献
1) Mills, S., Stanton, C., Hill, C., Ross, R. P.: Annu. Rev. Food Sci. Technol., 2, 299 (2011).
2) Santos, S., Torcato, I., Castanho, M. A.: Biopolymers, 98, 288 (2012).
3) Grottelli, S., Ferrari, I., Pietrini, G., Peirce, M. J., Minelli, A., Bellezza, I.: Int. J. Mol. Sci., 17, 1332 (2016).
4) Tanaka, T., Takagi, K., Saddam, H. M., Takeda, Y., Wakayama, M.: Appl. Biochem. Biotechnol., 183, 362 (2017).
5) Kino, H., Kino, K.: Biosci Biotechnol Biochem., 79, 1827 (2015).
6) Pi, H., Helmann, J. D.: Proc. Natl. Acad. Sci. USA, 114, 12785 (2017).
7) Abergel, R. J., Zawadzka, A. M., Hoette, T. M., Raymond, K. N.: J. Am. Chem. Soc., 131, 12682 (2009).
8) Finking, R., Marahiel, M. A.: Annu. Rev. Microbiol., 58, 453 (2004).
9) Labby, K. J., Watsula, S. G., Garneau-Tsodikova, S.: Nat. Prod. Rep., 32, 641 (2015).
10) Nguyen, K. T., Ritz, D., Gu, J. Q., Alexander, D., Chu, M., Miao, V., Brian, P., Baltz, R. H.: Proc. Natl. Acad. Sci. USA, 103, 17462 (2006).
11) May, J. J., Wendrich, T. M., Marahiel, M. A.: J. Biol. Chem., 276, 7209 (2001).
12) Gulick, A. M.: ACS Chem. Biol., 4, 811 (2009).
13) Ehmann, D. E., Shaw-Reid, C. A., Losey, H. C., Walsh, C. T.: Proc. Natl. Acad. Sci. USA, 97, 2509 (2000).
14) Conti, E., Franks, N. P., Brick, P.: Structure, 4, 287 (1996).
15) Röttig, M., Medema, M. H., Blin, K., Weber, T., Rausch, C., Kohlbacher, O.: Nucleic Acids Res., 39, W362 (2011).
16) Abe, T., Hashimoto, Y., Zhuang, Y., Ge, Y., Kumano, T., Kobayashi, M.: J. Biol. Chem., 291, 1735 (2016).
17) 安部智子, 橋本義輝, 小林達彦: バイオサイエンスとインダストリー, 74, 318 (2016).
18) Abe, T., Hashimoto, Y., Sugimoto, S., Kobayashi, K., Kumano, T., Kobayashi, M.: J. Antibiot., 70, 435 (2017).
19) Dawson, P. E., Muir, T. W., Clark-Lewis, I., Kent, S. B.: Science, 266, 776 (1994).
20) Johnson, E. C., Kent, S. B.: J. Am. Chem. Soc., 128, 6640 (2006).
21) Shang, S., Tan, Z., Danishefsky, S. J.: Proc. Natl. Acad. Sci. USA, 108, 5986 (2011).
22) Abe, T., Kobayashi, K., Kawamura, S., Sakaguchi, T., Shiiba, K., Kobayashi, M.: J. Gen. Appl. Microbiol. (in press).