【トピックス】
デジタルバイオアッセイとその応用
田端和仁、野地博行
東大院・工、内閣府・ImPACT
1.はじめに
近年、1分子検出法を元にしたデジタルバイオアッセイが勃興している。デジタルバイオアッセイでは、μmサイズの微小な容器を大量に準備し、検出したい分子を確率的に1分子以下になるように各容器に閉じ込める。すると、検出したい分子が入っている容器と入っていない容器が存在するため、検出したい分子のシグナルを検出することができれば,シグナルを発する容器を「1」とし、発しない容器を「0」としてデーターを得ることができる。この方法の最も特徴的な点は、1分子を検出しているため、シグナルの数を数え上げることで分子の数を知ることができる点である。また、微小容器の体積がわかっていればその数を濃度に変換することができ、検量線を必要としない絶対定量が可能になる。そのほかにも、検出のデジタル化によるs/nの向上に伴う高感度化や、分子集団のヘテロジェネリティの検出が可能になるといった特徴がある。
このような有用性から、デジタルバイオアッセイは、最近注目されている分析手法の一つである1,2)。デジタルバイオアッセイにおいて最も有名な例はデジタルPCRであろう。これは装置も販売されており、一般に普及しつつある。そのほかにも、Agresti J. J.らのドロップレット中に酵母1細胞を閉じ込めその発現タンパク質のスクリーニングをする方法や3)、Rondelez Y.らのドロップレット内に閉じこめた1分子の酵素活性を検出する方法4)、ドロップレット中に閉じこめた1細胞で行うsingle cell transcriptomeなど5,6)、様々な検出方法のデジタル化が報告されている。そこで、本稿では酵素のデジタルバイオアッセイを中心にその応用について報告したい。
1-1 デジタルバイオアッセイのためのデバイス
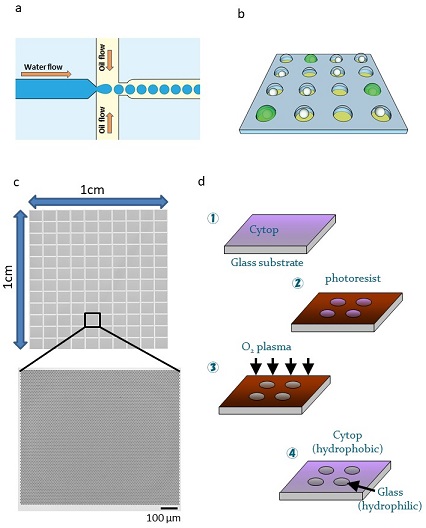
上記でも触れたが、デジタルバイオアッセイを実現するためには1分子を閉じこめるための微小な容器を大量に準備する必要がある。そのための方法としてよく用いられるのが油中水滴 (W/O droplet) を用いる方法である (図1a)。この方法ではマイクロ流体デバイスを利用することで、10μmから100μm程度の均一径の液滴を短時間で数百万のオーダーで調製することが可能である。一方、酵素の評価においては、活性の経時変化を知ることが重要である。しかし、この油中水滴を利用したシステムは、マイクロ流体デバイス中で行われるため、測定対象が観察面にとどまることなく流出してしまう。そのため、一つの液滴に注目してその時間変化を追うことは難しい。この問題に対処するため、生成後の液滴を回収して、観察のための別デバイスに導入するという手法もとられているが、手順や手間が増えるため、簡便性を失ってしまう。そこで我々は、基板上に液滴のアレイ構造を作る方法を考案した (図1b)。この方法では、液滴の個数は基盤のサイズやパターンによる制限を受けるため、マイクロ流体デバイスを利用した方法に比べ、自由度の点では劣る。しかし、個々の液滴が整列しているために各液滴のアドレッシングが可能となり、注目する液滴の経時的観察や回収が容易になるというメリットがある。我々が開発したドロップレットアレイデバイス (人工細胞デバイス)は、直径5μm程度、深さ3μm程度の微小なリアクターを1 cm2に100万個配置したデバイスである (図1c)。この人工細胞デバイスの作成は、ガラス基板にフッ素樹脂 (CYTOP) をコーティングし、光感光性であるレジストをその上にコートする。フォトリソグラフィーによって、微小容器のパターンを作成し、ドライエッチングによってパターン部分のフッ素樹脂を除去し、ガラス面を露出させる。最後に洗浄を行い完成する (図1d)。リアクター部分は底面にガラスが露出しており親水的になる。一方それ以外の部分はフッ素樹脂に覆われているため、疎水的である。このデバイスを用いて、フローセル構造を作成し内部を水溶液で満たした後、オイルなどの溶媒を連続的に流し込むと、リアクター部分は親水的であるため水溶液が残り、それ以外は溶媒で覆われてしまう。反応溶液部分にとどまった水滴は数十fL (10-15 L) となり、非常に微少な水滴のアレイを作成することができる。このような微小体積の液滴であれば、酵素反応産物の濃度上昇を容易に検出可能となる。例えば、1 fLの液滴に1秒間で10回反応する酵素が1分子閉じこめられているとする。そうすると、1分間で600分子の反応産物が生成されることになり、これは濃度に変換すると1μMもの濃度に達する。つまり人工細胞デバイスを利用すれば1分子酵素の活性を検出することが可能となる。

2.人工細胞デバイス内での1分子酵素アッセイ
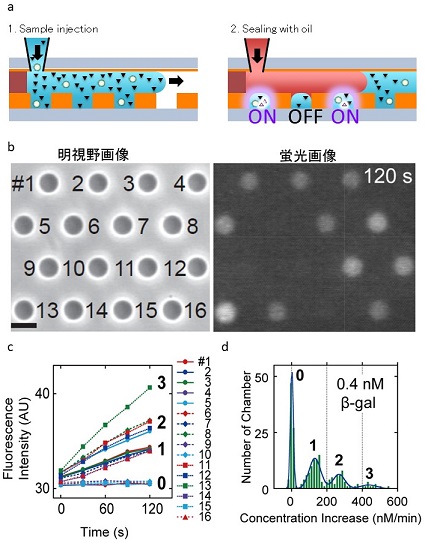
人工細胞デバイスを用いて、1分子酵素アッセイの可能性を検証した7)。酵素としてβ-galactosidase (βgal) を用い、その活性を可視化するための基質としてfluorogenicな基質であるFDG (Fluorescein di-D-Galactopyranoside) を用いた。これら2つを適当なバッファーに懸濁してデバイス内に導入するが、βgalに関しては、1分子アッセイを実現するため、1リアクター当たり、0.1個程度となるように加えている。この溶液をデバイスに導入した後、デバイスを氷上に置くことで気体の水に対する溶解度を上げる。こうすることにより、各リアクターに入っている空気がバッファーに溶解し、リアクター内部はバッファーで満たされる。続いて、フッ素系のオイルである、FC40を導入する (図2a)。すると、デバイス上の親水・疎水パターンに従い、オイルがリアクター周囲を満たすため、バッファーが入ったリアクターが孤立化される (図2b 明視野画像)。この状態のデバイスを顕微鏡上にセットし、各リアクターの蛍光強度をモニターする。一定時間後、図2bに示すように、蛍光を発するリアクターと発しないリアクターが観察される。これは、リアクター内のβgalの存在確率が1以下となっていることを示し、1分子のβgal活性を検出できている可能性を示している。また、各リアクターの蛍光強度を時間に対してプロットすると図2cのような直線関係が得られる。直線の傾きが離散的になっており、リアクター内のβgalが1分子存在する場合、2分子存在する場合となっていることを強く示唆している。これらの傾きの値から蛍光色素の濃度変化を計算し、ヒストグラム化したものが図2dである。このヒストグラムも複数のピークを示し、そのピークの間隔もほぼ一定である。このヒストグラムをポアソン分布に従うとしてフィッティングを行った所、大変よく一致した。これらのことから、得られたピークはリアクター内にβgalが0分子、1分子、2分子、3分子入っている場合の酵素活性を示していることが示された。つまり、本人工細胞デバイスにて酵素1分子の活性を測定可能であることが明らかとなった。

3.人工細胞デバイスを用いた1分子酵素アッセイの応用
3-1 Digital ELISA
冒頭でも述べたが、近年デジタルバイオアッセイが勃興している。これは、微小空間内に目的の1分子を閉じこめて検出する新しい高感度アッセイの方法である。原理が1分子の検出に基づいているため、検出した回数や個数をカウントすることで、系の中に存在する目的物質の絶対数が計測可能であり、新たな絶対定量の方法としても注目されている。
そこで、私たちは医療現場において頻繁に行われるELISA法のデジタル化に取り組んだ8)。ELISAはEnzyme-Linked ImmunoSorbent Assayの略であり、抗原抗体反応の検出を酵素活性を利用して行うアッセイである。一般的な方法では、まず固体表面上に固定した抗体をサンプルと混合して抗原と結合させる。その後、B/F分離を行い、酵素を結合させた検出用の抗体を加え、固体表面上の抗原-抗体複合体に結合させる。すると、抗体-抗原-検出用抗体という3者複合体が完成する。さらにB/F分離を行い、検出用抗体が持つ酵素の蛍光、発光基質を加えることで3者複合体を検出し、抗原の検出を行っている。この3者複合体1分子をリアクター内に閉じこめることができれば、先ほどの酵素活性の検出同様に、1分子レベルでのELISAが可能になるのでは無いかと考えた (図3a)。
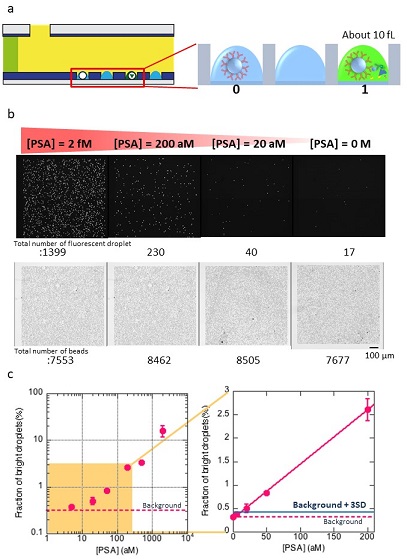

そこで、前立腺癌のマーカー分子であるPSAのELISAを目的にデジタルアッセイの開発を試みた。まずは、直径3μmのプラスチックビーズの表面にPSAに対する抗体を結合させ、βgalを結合した検出用抗体を用いて、一般的な方法に基づくELISAアッセイを行った所、その検出感度はpMオーダーとなり、通常のELISAと同等の感度が得られることが分かった。そこで、この抗体結合ビーズを使ってデジタル化を行った。抗体結合ビーズに対して様々な濃度のPSA溶液を作用させ、抗原抗体反応を行った。検出抗体を加えた後、B/F分離を行ったものをFDGを含むバッファー溶液と共にデバイス内に導入し、FC40を用いて各リアクターを孤立化したものを顕微鏡で観察した。まずは明視野で観察を行い、リアクターに補足されたビーズを確認しその個数を確認した (図3b)。続いて、蛍光観察を行い、蛍光を発しているリアクターを確認した。ここでも、蛍光を発しているリアクターと発していないリアクターが観察されたため、ビーズ上の3者複合体は1分子であることが強く示唆された。また、PSA濃度依存的に蛍光を発しているリアクターの数が減少していることからもPSAを含む3者複合体を検出できていることが分かった。そこで、PSAの濃度を横軸にとり、補足されたビーズの総数に対して、蛍光を発するビーズが含まれているリアクターの割合を縦軸にとって結果をプロットした (図3c)。すると、非常に直線性の良いプロットが得られ、aM (10-18M) レベルの極低濃度においてもシグナルが検出できていることが分かった。本アッセイの検出感度を決定するため、PSAを含まないネガティブコントロールの実験からその値の標準偏差の3倍 (3S. D.) をLOD (Limit Of Detection) として定義したところ、その値は15aMであった。この結果は、従来のELISA法より100万倍程度の高感度化に成功したことを示すものである。
3-2 Digital インフルエンザ診断
つづいて、インフルエンザウイルスの検出のデジタルバイオアッセイ化に取り組んだ。今冬は、インフルエンザが大流行し、患者数は過去最高を更新して2月初頭で280万人が罹患している。この大流行の原因は、隠れインフルと呼ばれる微熱程度で発熱も1日ぐらいのインフルエンザ患者が多く、ほとんどの人がインフルエンザだと気づかないためである。一方、インフルエンザは感染初期に投薬すればウイルス放出量も少なくなることが分かっており、重症化も抑えられることが知られている。しかしながら、現在のインフルエンザ診断は、イムノクロマト法をベースにしており、それほど感度が高くない。そのため、陰性率は60%程度とかなり多く陽性の取りこぼしが出てしまう。また、検査方法も鼻の奥に検体採取用の綿棒を突っ込むため、痛みを伴い患者にとって気軽な検査とは言えない。
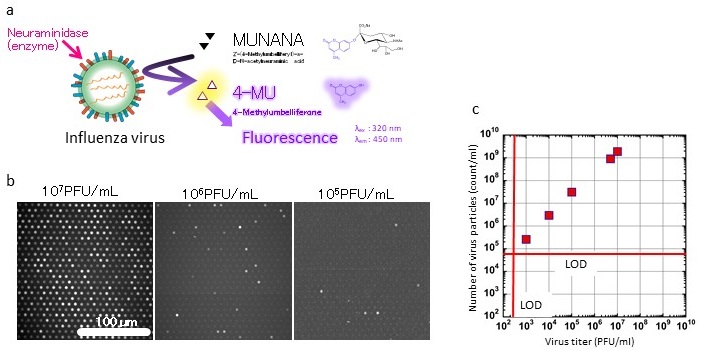
そこで、より高感度かつ簡単なインフルエンザウイルス検査方法開発を目指して、デジタルアッセイの開発を開始した。インフルエンザウイルスには、その表面にヘマグルチニン (HA) とノイラニミニダーゼ (NA) と呼ばれるタンパク質が露出している (図4a)。このノイラミニダーゼは末端シアル酸残基のα- (2→3) (鳥インフルエンザノイラミニダーゼ) , α- (2→6) (ヒトインフルエンザノイラミニダーゼ) 結合を認識して切断するグリコシダーゼの一種である。つまり、インフルエンザウイルスはその表面に酵素をたくさん提示しており、この活性を検出することができればウイルス検出を実現できる。そこで、市販されているノイラミニダーゼの蛍光性基質であるMU-NANAを用いて、デジタルバイオアッセイを試みた。MU-NANAを含むバッファーにインフルエンザウイルスを加えて人工細胞デバイスに導入し、他の実験と同様にオイルで各リアクターを孤立化させた。ここで、使用したウイルスの力価はPFU (Plaque Formation Unit) /mlという単位を用いて計測している。コンフルエントになった培養細胞にウイルス溶液を加えると、感染が成立した細胞周辺はウイルス放出によって死滅するため、染色によって、透明なプラークが形成される。この個数を数えて1 ml当たりに規格化した値が上記の数値になる。この値は1 mlあたりの感染できるウイルスの個数として考えられている。
109 PFU/mlのウイルスサンプルから連続希釈で様々な濃度のサンプルを調製し、デジタルバイオアッセイを行った所、蛍光を発するリアクターが確認できた。また、蛍光を発しないリアクターも確認できたこと、さらには濃度依存性も見られたことから1ウイルス粒子が検出できていることが分かった (図4b)。ウイルス濃度 (PFU/ml) に対して、蛍光を発したリアクターを1ウイルスとしてカウントし1 ml当たりの個数としてプロットしたところ、PFU/mlの値に対して、ウイルス数は200倍程度 (インフルエンザA型1H1N PR8において) 多いことが分かった (図4c)。これは、ウイルス全体のうちほとんどが感染できないウイルスであることがわかった。さらに、この方法の検出感度を決定するため、ウイルスを含まないサンプルの計測を行ったが、180万リアクターを観察しても擬陽性シグナルが得られなかった。これは、本デジタルインフルエンザ検出法の検出感度が、実験的には決定できないほど低いということを示している。一方、現状の計測システムで、1リアクターだけ蛍光を発したと仮定し、検出感度を計算すると、その値はおよそ6.2×104 count/ml (=3.3×102 PFU /ml) 以下となった。これは、一般的なRT-PCR法による検出感度とほぼ同等の値であった。

一方、市販されているインフルエンザ迅速診断キットを購入しその検出感度を同じウイルスを使って調べたところ、109 count/ml (=106PFU/ml) 程度であった。これは、従来の診断法と比べ、デジタルアッセイでは1万倍程度の高感度化を達成できたことになる。これだけの高感度化が達成されれば、診断の現場においても発熱前診断などが可能になることが考えられる。また、うがい液や唾液などのウイルスが少ないサンプルからの診断も考えられ、患者負担の少ない検査法の確立も期待できる。
3-3 1分子DNAからの酵素発現とスクリーニング
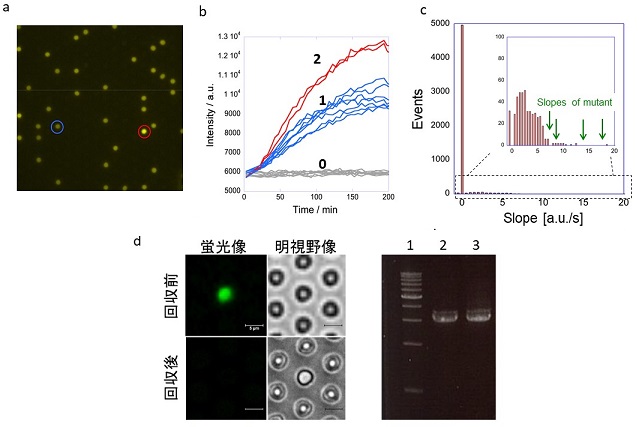
今回開発した人工細胞デバイスは、およそ100万個の微小なリアクターの集まりという非常に単純なものである。ゆえに、これらを単なるリアクターであると見なし、チューブ内や試験管内で行う反応を再構成することを試みた。今回の試みでは、PUREシステムとして知られる無細胞転写翻訳系をDNA1分子から反応させ、タンパク質合成が可能かを検証した。蛍光タンパク質であるVenusをコードしたDNAを準備し、PUREシステムの溶液と混合し、各リアクター中にDNAが1分子以下となるような濃度でリアクターに封入し、顕微鏡下でVenusの蛍光を経時的に観察した。すると、数時間の後に蛍光を発するリアクターが観察できた (図5a)。各リアクターからの蛍光強度解析を行ったところ、蛍光強度増加の傾きが離散的であるなど、1分子検出の特徴を備えていた (図5b)。このことより、1分子DNAからのタンパク質発現が人工細胞デバイス内で可能なことが分かった。
1分子DNAからのタンパク質発現を行い、その機能を解析できるならば、酵素の進化分子工学的アプローチが本人工細胞デバイスを利用して可能ではないかと考えた。そこで私たちは、βgalの遺伝子をエラープローンPCRによってライブラリ化し、そこから高活性なβgalのスクリーニングを試みた。今回のライブラリはβgalの遺伝子当たり平均3.6個の変異を含んでいる。このライブラリをPURE溶液とFDGとともに混合し、先ほどと同様に1リアクター当たり1DNA以下となる濃度で封入したため、およそ104〜105のスクリーニングサイズとなった。また、今回は変異の入っていないDNA (WT) も対照として同時に実験を行った。すると、この実験においても数時間後からβgal活性に由来する蛍光がいくつかのリアクターで観察された。そこで、この蛍光強度上昇の傾きをヒストグラムとしてプロットした (図5c)。WTのヒストグラムに対して明らかに傾きの大きいリアクターがいくつか見つかったため、これらのリアクターに含まれるDNAをガラスキャピラリーによって回収し、PCRによって増幅したところ、βgal由来のバンドが明瞭に観察された (図5d)。そのうちの一つのDNAの配列を決定したところ、I850Fの変異が入っていることが分かった。さらに、この配列を大腸菌に導入し、発現させたタンパク質を精製してWTと比較したところ、約2倍の活性増加が確認された。このように、104から105のスクリーニングサイズでの酵素機能をベースとしたスクリーニングが1日から2日程度で完了できることが示された。これは、微小リアクターアレイ内での1分子DNAからのタンパク発現とその機能解析を超並列化することによって実現されており、酵素の人工進化実験を大幅に効率化できることを意味している。

4.おわりに
今回開発した人工細胞デバイスを利用して、酵素1分子アッセイからそれを応用した様々な計測、実験手法を開発した。これ以外にも、微小リアクターを細胞そのものと見立てて、その内部に細胞を再構成する試みなども行っている。これは、人工細胞デバイスの基本コンセプトが小さな試験管の集まりという単純なものであるため、様々な反応をリアクター内で行うことができるためである。今後も、様々なデジタルバイオアッセイの開発や、細胞機能の再構成などに取り組んでいきたいと思っている。
謝辞
本研究を実施するに当たり、野地研究室のメンバーの努力に感謝する。また、デバイス開発に当たっては東京大学の竹内昌治教授にご協力を頂いた。さらに、インフルエンザウイルスに関しては、東京大学の河岡義裕教授、北海道大学の大場雄介教授に分与していただいた。これ以外にもご協力いただいた方々に感謝する。
文献
1) Basu, A. S.: SLAS Technol., 22, 369 (2017).
2) Basu, A. S.: SLAS Technol., 22, 387 (2017).
3) Agresti, J. J., Antipov, E., Abate, A. R., Ahn, K., Rowat, A. C., Baret, J. C., Marquez, M., Klibanov, A. M., Griffiths, A. D., Weitz, D. A.: Proc. Natl. Acad. Sci. USA, 107, 4004 (2010).
4) Rondelez, Y., Tresset, G., Tabata, K. V., Arata, H., Fujita, H., Takeuchi, S., Noji, H.: Nat. Biotechnol., 23, 361 (2005).
5) Macosko, E. Z., Basu, A., Satija, R., Nemesh, J., Shekhar, K., Goldman, M., Tirosh, I., Bialas, A. R., Kamitaki, N., Martersteck, E. M., Trombetta, J. J., Weitz, D. A., Sanes, J. R., Shalek, A. K., Regev, A., McCarroll, S. A.: Cell, 161, 1202 (2015).
6) Klein, A. M., Mazutis, L., Akartuna, I., Tallapragada, N., Veres, A., Li, V., Peshkin, L., Weitz, D. A., Kirschner, M. W.: Cell, 161, 1187 (2015).
7) Sakakihara, S., Araki, S., Iino, R., Noji, H.: Lab Chip, 10, 3355 (2010).
8) Kim, S. H., Iwai, S., Araki, S., Sakakihara, S., Iino, R., Noji, H.: Lab Chip, 12, 4986 (2012).