【トピックス】
低酸素条件下で形成される代謝酵素集合体G-bodyの発見
三浦夏子
阪府大院・生命環境
1.はじめに
細胞質におけるタンパク質集合体の形成は、膜に依存せず細胞区画を形成する新たな機構として世界的に注目を集めている1)。我々は以前の研究で、酵母Saccharomyces cerevisiae BY4741株において、15以上の代謝酵素群が低酸素条件下で集合体を形成することを見出した2)。さらに、集合体形成によって、グルコース代謝の効率が上昇している可能性を示唆した2)。2017年、アメリカの他グループにより、同様の現象が他の酵母株やヒト培養細胞株でも観察された3)ことから、我々が酵母で見出した現象が、真核生物に広く存在する可能性が示唆された。代謝酵素、特に解糖系酵素を多く含むこの集合体は、“Glycolytic body” または “G-body” と名付けられ3)、新規な代謝調節機構として注目を集めつつある1)。
低酸素条件下におけるG-body形成は、京都大学大学院農学研究科応用生命科学専攻生体高分子化学研究室 (植田充美教授、以下、生体高分子化学研究室) において、2000年代当時ほとんど不明であった、解糖系酵素の分泌機構を調べる過程で偶然見出した現象である。本稿ではまず、G-body発見に至るまでの経緯と周辺状況について振り返りたい。次に、G-bodyの制御機構とその生理的意義について、Jinら (2017) による最新の報告を交えながらまとめて述べる。最後に、研究の現状と今後の展開についてディスカッションしたい。
2.Moonlighting proteins -解糖系酵素の新たな側面
解糖系酵素は細胞の中心代謝を担うタンパク質として知られるが、その働きには代謝酵素として以外の機能も含まれることが明らかとなっており、こうした副業としてのタンパク質機能を “Moonlighting機能” と呼ぶ4,5)。“Moonlighting機能” を発揮するMoonlighting proteinsとして最もよく知られるのが解糖系酵素である6,7)。中でも解糖系酵素エノラーゼは、亀の眼のレンズを構成するクリスタリンから単離され、その状態で酵素活性を保持していた8)ことから、乳酸デヒドロゲナーゼとともに、初めて発見されたMoonlighting proteinsの一つとして知られている。解糖系酵素のMoonlighting 機能には、細胞外で接着や細胞間コミュニケーション等にはたらく機能も多く含まれる9-12)が、なぜ通常は細胞内に存在すると考えられる解糖系酵素が細胞外で機能するのかは明らかになっていなかった。
3.細胞外に存在する解糖系酵素
真核生物において、解糖系酵素は中心代謝を司るタンパク質として細胞質に存在する。我々が後にG-bodyにつながる研究を開始した2006年から2010年代前半にかけては、こうした認識が一般的であった。従来よく知られている、ERからゴルジ体、分泌小胞を介した分泌 (Conventional protein secretion, 以下CPS) 経路に対して、通常の分泌経路を通らない経路をUnconventional protein secretion (UPS)13)経路と呼ぶ。現在ではUPSも広く認識されるようになったが、Pubmed上でこの単語が初めてみられたのが2003年である13)ように、当時CPSに必須な分泌シグナルを持たないタンパク質が分泌される例は、ほとんど報告されていなかった。また、プロテオーム解析などで細胞外タンパク質として解糖系酵素が検出されても、サンプルを扱う途中で細胞が破裂したり、細胞膜が傷ついて中身が漏れ出したりした可能性を否定することは難しかった。実際、解糖系酵素は平均的なタンパク質に比べて、細胞内での存在量が多い。酵母S. cerevisiaeでは、一細胞内における総タンパク質分子数がおよそ5.0 × 107分子存在する14)のに対して、Kulakらによる報告 (2014) では解糖系酵素エノラーゼ (Eno2p) 単体の存在数は一細胞当たりおよそ9.3 × 105分子15)と、全タンパク質分子数の2%に迫る。ちなみにアクチン (Act1p) の存在数は一細胞当たりおよそ1.2 × 105分子15)であり、エノラーゼの7分の1に満たない (以上、Saccharomyces Genome Database [https://www.yeastgenome.org/] 参照)。従って、細胞質に存在する解糖系酵素がわずかに漏れ出しただけでも、分泌量の低い細胞外タンパク質に相当する分子数が検出されてしまう可能性がある。また、当時は解糖系酵素の分泌を制御する因子が見つかっていなかったこともあり、細胞外での検出イコール未知の機構による分泌とは判定できないという問題があった。
一方で既に、解糖系酵素が細胞外に存在することを指摘する報告は蓄積されつつあった。特に酵母S. cerevisiaeでは、Edwardsら (1999)16)やLopez-Villarら (2006)17)がいち早く細胞外に存在する解糖系酵素を見出していた18)ほか、Oliveiraら (2010) は細胞外小胞とその中にある解糖系酵素の存在を指摘していた19)。他の生物でも、細胞外タンパク質のプロテオーム解析により、ヒト間葉系幹細胞20)や、Bacillus subtilis21-22)等でも、細胞外における解糖系酵素の存在が認められた。しかしながら、解糖系酵素の分泌機構は依然として不明であったため、その検証は困難であった。
当時生体高分子化学研究室では、プロテオーム解析に資するモノリスカラムの開発により、2012年には酵母S. cerevisiaeの表層タンパク質解析技術が確立していた23)。こうした中で、生体高分子化学研究室でも、線虫24,25)や酵母26)の細胞外に存在する解糖系酵素を検出した。そこで疑問となるのは、やはり、果たして解糖系酵素が実際に分泌されているのか、それとも単に細胞から漏れ出した解糖系酵素が検出されたのか、ということであった。一方で、特に酵母の表層タンパク質解析法は、漏れ出したタンパク質の有無を検定する方法を組み込んで慎重に構築されており23)、後者については考えにくかった。幸いなことに、酵母では2013年にノーベル医学生理学賞を受賞したRandy Schekman らにより分泌経路を阻害する各種変異株が構築されており27-30)、特定のタンパク質が既知の分泌経路を通るか否かの判定が可能であった。酵母にはまた加えて、遺伝子組み換えが容易で扱いやすいという利点があった。そこで酵母S. cerevisiaeを用いて、解糖系酵素分泌を検証するための再構成系を考案した。
解糖系酵素の分泌を検証する上で我々がとった手法は、プラスミドを用いてタグ付けした解糖系酵素を細胞内で発現させ、定量ウエスタンブロッティングにより細胞外への分泌の有無を判定する31)というシンプルなものであった。細胞内発現する解糖系酵素には、その分泌経路を可視化する目的で蛍光タンパク質EGFPを融合して発現させた。詳細な手法31)及び結果26)については本稿では割愛するが、この手法により、エノラーゼを含む複数の解糖系酵素の分泌を再構成することができた。また、この手法を酵母遺伝子欠損株ライブラリに拡張したところ、既知のUPS により分泌されるタンパク質とは異なり、少なくともエノラーゼの分泌は、主要なオートファジー関連遺伝子の欠損に影響されないことが判明した。一方で、SNAREタンパク質をコードするTLG2の欠損株では、エノラーゼの分泌が有意に阻害されることが分かった。またセントロメア型プラスミドを用いてTLG2欠損株にTLG2の再導入を行ったところ、エノラーゼの分泌が回復した。以上の結果から、解糖系酵素エノラーゼがTLG2依存的に分泌されることが示唆できた26)。
解糖系酵素の分泌経路を探る中で、エノラーゼに含まれる特定のアミノ酸配列がUPS経路を介した分泌に寄与しているのではないかと考え、エノラーゼの断片化も行った。N-末端を残してC-末端側から削ったエノラーゼ断片に、タグ配列とEGFPを融合させたところ、エノラーゼのN-末端 (1-28) アミノ酸をEGFPに融合発現させたタンパク質 (Eno2p(1-28)-EGFP) も、細胞外に分泌されることが分かった26)。また、興味深いことに、この融合タンパク質は細胞内でドットのような局在を示した (図1)。その局在は核膜タンパク質や細胞膜タンパク質と一部オーバーラップするものの、核やその他の細胞小器官とは明確に異なっていた26)。こうした観察結果から、エノラーゼ全長も特定の条件下では細胞質において同様の局在を示すのではないかと考えた。
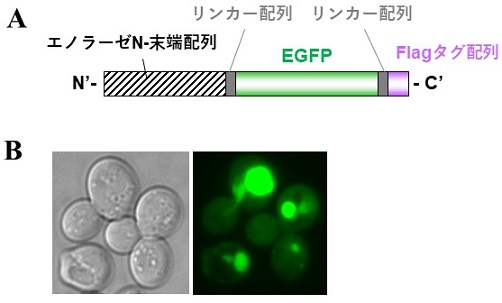
![]()
4.低酸素条件下における解糖系酵素の細胞内局在変化
4-1 エノラーゼが低酸素条件下で形成する “Foci” の発見
タンパク質の新規な局在性の発見はしばしば、未知なタンパク質機能の発見につながる。特に、数々のMoonlighting機能を持つエノラーゼであれば、未知の機能が存在しても不思議ではない。こうした考えから、エノラーゼN-末端配列で見られた細胞内局在性がエノラーゼ全長でも見られる条件の探索を考えた。一方で、起こるかどうかわからない現象に対して、膨大な数の条件検討を当てもなく行うことは困難であるとも考えられた。そんな中、分泌経路探索用に構築した、エノラーゼ全長にEGFPを融合発現する酵母を培養した試験管を、培養後、試験管立てに立てたまま常温でしばらく放置してしまい、酵母が試験管の底に沈んで溜まってしまった。これを蛍光顕微鏡で観察したところ、細胞質に不均一に存在する蛍光を見出した。当時、廃棄すべきサンプルを顕微鏡で観察しようとした理由は記憶にないが、その後何度か静置培養を繰り返し、振とう培養と静置培養で、エノラーゼ全長とEGFPの融合タンパク質 (Eno2p-EGFP) の細胞内局在性が異なることが分かった (図2)。つまり、静置培養で生じる何らかの条件が、Eno2p-EGFPの局在変化を引き起こしている可能性が考えられた。静置培養時のEno2p-EGFPの局在は、ドットのように見えたため、こうした局在を単純に “foci” もしくは “enolase foci” と呼ぶことにした。この “foci” が後に “G-body” と名付けられた細胞内タンパク質の集合体であった。
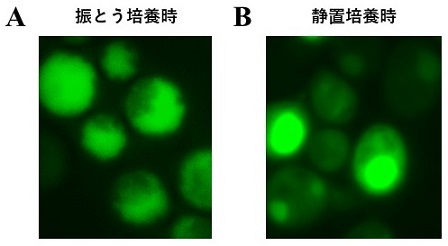

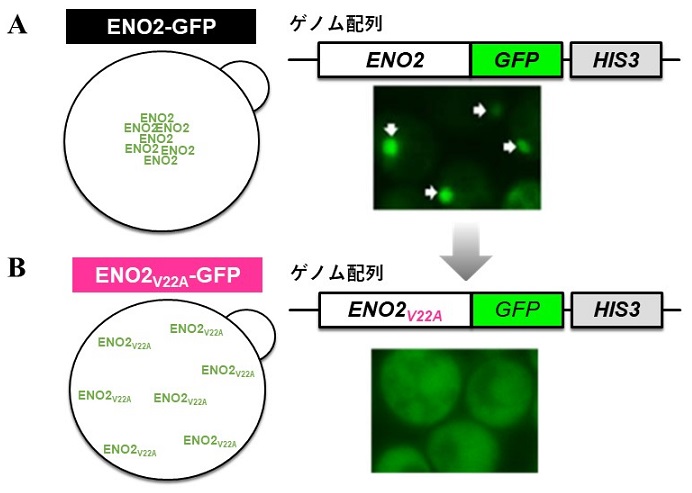
エノラーゼの局在変化は観察できたものの、静置培養条件で具体的に何がfoci形成の引き金となっているのかは不明であった。通常の培養条件では培地の栄養条件等を変更したり、いくつかのストレスを加えたりしても、foci形成を再現することはできなかった。そこで、当時別グループで使用されていた、微嫌気培養容器32)を利用して、酸素濃度が変化した条件での局在を調べることにした。この培養容器には、サンプリング用と空気抜き用に、上部から二か所金属の筒が差し込まれている。低酸素培養を行う場合は、事前に二酸化炭素を吹き込んで溶存酸素量を下げてから培養を開始する。同じ容器で好気培養を行う場合は、サンプリング用金属筒から逆に空気を送り込むことで、溶存酸素量を保つことが可能である (図3)2)。また、エノラーゼの分泌を調べる際には主にマルチコピープラスミドを用いていたため、タンパク質の過剰発現による影響を排除するために、ゲノムにGFPが組み込まれたGFP株33)を新たに用いて微嫌気培養を行った。その結果、ゲノム上のエノラーゼ3’-末端にGFP遺伝子を組み込んだ株で、低酸素条件下でenolase fociが形成されることを見出した (図4)2)。以上のように、エノラーゼによる局在変化は、低酸素条件下で引き起こされることが明らかとなった。以上の結果を2011年、第34回日本分子生物学会大会で初めて発表し、その際に大きな反響と多くの有用なアドバイスを得たことで、enolase fociの制御機構と生物学的な意義を解明するモチベーションを得ることができた。のちのJinらによる実験では、マルチガスインキュベーターを用いた低酸素条件下でのプレート培養でもG-body形成が見られており3)、G-body形成は液体培養・固体培養ともにみられる現象であることが示されている。
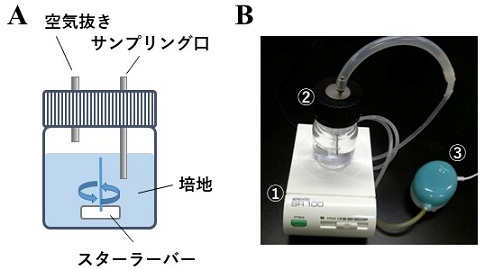

![]()
4-2 “Foci” を構成するタンパク質群
次に生じた疑問は、低酸素条件下でみられるenolase fociにどのようなタンパク質が相互作用しているのか、ということであった。Eno2p-EGFPには図1A同様、Flagタグを付与していたため、anti-Flag抗体を用いて細胞内タンパク質を共免疫沈降し、モノリスカラムを用いたLC-MS/MSにより定性解析したところ、fociを形成する条件下で15の代謝酵素を含む29タンパク質を特異的に検出した2)。さらに、検出できたタンパク質とのGFP融合タンパク質発現株を用いて、それぞれ低酸素条件下での局在を検証したところ、ほとんどの代謝酵素がエノラーゼと同様にfociを形成した2)。種々の代謝酵素が形成するfociとエノラーゼのN-末端 (1-28) アミノ酸をDsRed monomerに融合発現させたタンパク質は、ほぼ同じ局在を示したことから、低酸素条件下において15を超える代謝酵素の集合体が形成されていることが示唆された2)。後に、Jinらの報告ではさらに、G-bodyを細胞から抽出・濃縮しプロテオーム解析を行っているほか、ゲノムに組み込まれた全長の代謝酵素同士でも共局在がみられることを示しており3)、ここから低酸素条件下で複数の代謝酵素が構成する “G-body” の存在が提唱された。
4-3 G-body形成に必要なアミノ酸配列
エノラーゼのN-末端配列が、代謝酵素により形成されるfociと同様の局在を示したことから、エノラーゼによるG-bodyの形成にはN-末端配列が寄与していることが考えられた。そこでまず、foci形成に必要十分なN-末端配列を決定した。また、foci形成に必要十分なN-末端の(5-25)アミノ酸を一つずつアラニン置換し、22番目のバリン残基をアラニンに置換したときにfoci形成が阻害されることを見出した。この1アミノ酸置換をゲノム上に存在するエノラーゼに導入したところ、低酸素条件下でfoci形成が見られなかった (図4B) ことから、エノラーゼによるG-bodyの形成にはN-末端の(5-25)アミノ酸配列に加えて、22番目のバリン残基が重要であることが明らかになった2)。foci形成に必要十分なN-末端の(5-25)アミノ酸はβシート構造を取り、酵素の活性に関与するアミノ酸残基を含まない34)が、エノラーゼが二量体を形成する際の界面に位置することが結晶構造解析により明らかになっている34)。エノラーゼN-末端領域に類似したアミノ酸配列は他のG-body形成タンパク質群に共通して保存されていないため、G-body形成の配列依存性がどの程度見られるかは依然として不明である。また、エノラーゼN-末端領域の修飾や他のタンパク質との相互作用については不明な点が多く、さらなる解析が必要である。
4-4 G-body形成に関わる調節因子の探索
エノラーゼをはじめとするタンパク質の局在が、低酸素条件下でアミノ酸配列依存的に変化することは明らかになったものの、G-bodyの形成が細胞内で既知のシグナル伝達経路等により制御された機構であるのかは不明であった。そこで、種々の阻害剤や遺伝子ノックアウトにより、低酸素条件下でenolase foci形成が阻害される条件を探索した。その結果、複数のミトコンドリア阻害剤と抗酸化物質に加え、タンパク質合成阻害剤やTOR経路阻害剤を用いた場合にenolase fociの形成が阻害された。さらに、酵母におけるAMPキナーゼのホモログであるSNF1の欠損株でも、完全ではないもののenolase foci形成阻害がみられた2)。Jinらの報告でも、SNF1もしくはスーパーオキシドジスムターゼ (SOD1) の欠損株で他の解糖系酵素によるfoci形成が阻害された3)ことから、G-body形成は、低酸素条件下で機能的なミトコンドリアにより生産される活性酸素種 (ROS) と、AMPキナーゼを介したシグナル伝達経路により制御されていることが示唆された (図5)。
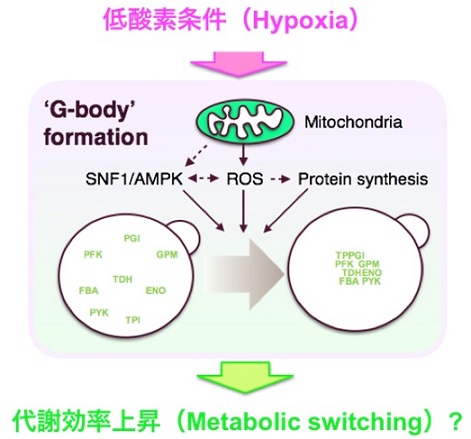
図5 低酵素条件下におけるG-bodyの形成調節機構と細胞における役割 (現時点での推定)。
4-5 G-body形成の生理的な意義
次に疑問となったのは、G-body形成が細胞で何らかの機能を果たしているのかということであった。G-body形成を制御するAMPキナーゼは主に糖代謝を制御することが知られていたことから、G-body形成はグルコース代謝に影響する可能性があると考えた。また、複数の代謝酵素が集合体を形成する場合、細胞内に一様に存在している場合に比べて酵素間距離が短くなり、基質の受け渡しが加速される可能性が考えられた。そこで、[U-13C]-Glucoseを基質とした経時的な代謝解析により、Glucose代謝をトレースする手法2, 35, 36)を採用した。幸いなことに、G-bodyは一度形成されると、細胞を好気条件においても20時間程度は保持されていたため、G-bodyを保持する株と保持しない株をほぼ同じ条件で比較することが可能だった。
13Cでラベルしたグルコースを酵母細胞に与えると、5分以内にピルビン酸まで変換される様子を測定することができる。低酸素条件下でG-bodyを形成する野生株では、好気条件下で培養した場合よりも低酸素条件下で培養した後 (G-bodyを保持している場合) の方が、有意に短時間でピルビン酸またはオキサロ酢酸への変換が行われていた。一方で、G-bodyを形成しない変異株では、好気条件・低酸素条件で培養した株にそのような違いは見られなかった。この結果から、G-bodyの形成は、低酸素条件下におけるグルコース代謝の調節に寄与していることが示唆された2) (図5)。
5.“Foci” もしくは “G-body” の再発見
以上のように、enolase fociの制御機構と機能の大枠が明らかとなり、2013年にアメリカ微生物学会発行のEukaryotic Cell 誌に論文が受理された。同年、内藤記念若手研究者海外派遣助成金の援助を得てアメリカ分子生物学会 (ASBMB2013) シンポジウム “Regulation and Evolution of Metabolic Pathways” にて一般選抜招聘講演を行ったものの、その後しばらく関連した報告は出てこなかった。2017年になり、Jinらによる酵母での追試と、ヒト培養細胞を用いた同様の現象の確認が報告され、他グループにも低酸素条件下で局在変化する解糖系酵素の存在が認められた。G-body以前にも、プリン合成系酵素が形成するPurinosome37)等、真核生物で代謝酵素が形成する集合体は報告されていたが、近年こうした細胞質で形成されるタンパク質集合体が脚光を浴びつつあり1)、今後タンパク質集合体群のさらなる実態解明が期待される。
6.おわりに
G-bodyは、その存在は認識されつつあるものの、未だ詳細については大部分が明らかでない。タンパク質の局在制御によって代謝調節を行うという機構が、転写調節による代謝調節機構に対してどのような進化を遂げてきたのかは興味深い。一方で、現在G-body集合体形成の分子機構はほぼ未解明であり、G-body構成因子についての解析も進んでいない。Jinらの報告では、G-bodyはミトコンドリアに匹敵する巨大な細胞内区画として観察されており、その構成を可視化することも今後大きな課題となる。また、G-body形成の制御機構については、部分的な要素は明らかになっているものの、要素間の相互作用については不明な点が多い。今後の解析ではこうしたG-body制御機構の完全解明に向けて注力する必要がある。将来的にはG-body形成にかかる分子機構の解明を通して、解糖系酵素をはじめ、生物種を超えて広く保存されるタンパク質が持つ新たな側面が明らかになると期待される。
謝辞
本稿で取り上げた研究は、生体高分子化学研究室・植田充美教授のもと、森坂裕信前助教と黒田浩一准教授、カールツァイス株式会社・佐藤康彦様、北陸先端科学技術大学院大学・高木昌宏教授と研究室の皆様、大阪大学・福崎英一郎教授と研究室の皆様、東京工業大学・門之園哲也助教、京都大学・原田浩教授はじめ、多くの方々のご協力・ご助言を得て行いました。本研究はまた、日本学術振興会特別研究員奨励費 (09J02920)、2012年度 (春季) 内藤記念若手研究者海外派遣助成金、JST-CREST (JPMJCR16G2) からそれぞれ部分的に補助を受けました。この場を借りて御礼申し上げます。
文献
1) Boeynaems, S., Alberti, S., Fawzi, N. L., Mittag, T., Polymenidou, M., Rousseau, F., Schymkowitz, J., Shorter, J., Wolozin, B., Van Den Bosch, L., Tompa, P., Fuxreiter, M.: Trends Cell Biol., 28, 420 (2018).
2) Miura, N., Shinohara, M., Tatsukami, Y., Sato, Y., Morisaka, H., Kuroda, K., Ueda, M.: Eukaryot. Cell, 12, 1106 (2013).
3) Jin, M., Fuller, G. G., Han, T., Yao, Y., Alessi, A. F., Freeberg, M. A., Roach, N. P., Moresco, J. J., Karnovsky, A., Baba, M., Yates, J. R., 3rd, Gitler, A. D., Inoki, K., Klionsky, D. J., Kim, J. K.: Cell Rep., 20, 895 (2017).
4) Jeffery, C. J.: Trends Biochem. Sci., 24, 8 (1999).
5) Jeffery, C. J.: Sci. Prog., 100, 363 (2017).
6) Sriram, G., Martinez, J. A., McCabe, E. R., Liao, J. C., Dipple, K. M.: Am. J. Hum. Genet., 76, 911 (2005).
7) Huberts, D. H., van der Klei, I. J.: Biochim. Biophys. Acta., 1803, 520 (2010).
8) Wistow, G. J., Lietman, T., Williams, L. A., Stapel, S. O., de Jong, W. W., Horwitz, J., Piatigorsky, J.: J. Cell Biol., 107, 2729 (1988).
9) Miles, L. A., Dahlberg, C. M., Plescia, J., Felez, J., Kato, K., Plow, E. F.: Biochemistry, 30, 1682 (1991).
10) Furuya, H., Ikeda, R.: Microbiology, 155, 2707 (2009).
11) Crowe, J. D., Sievwright, I. K., Auld, G. C., Moore, N. R., Gow, N. A., Booth, N. A.: Mol. Microbiol., 47, 1637 (2003).
12) Katakura, Y., Sano, R., Hashimoto, T., Ninomiya, K., Shioya, S.: Appl. Microbiol. Biotechnol., 86, 319 (2010).
13) Nickel, W.: Eur. J. Biochem., 270, 2109 (2003).
14) Futcher, B., Latter, G. I., Monardo, P., McLaughlin, C. S., Garrels, J. I.: Mol. Cell Biol., 19, 7357 (1999).
15) Kulak, N. A., Pichler, G., Paron, I., Nagaraj, N., Mann, M.: Nat. Methods, 11, 319 (2014).
16) Edwards, S. R., Braley, R., Chaffin, W. L.: FEMS Microbiol. Lett., 177, 211 (1999).
17) Lopez-Villar, E., Monteoliva, L., Larsen, M. R., Sachon, E., Shabaz, M., Pardo, M., Pla, J., Gil, C., Roepstorff, P., Nombela, C.: Proteomics, 6, Suppl 1, S107 (2006).
18) Nombela, C., Gil, C., Chaffin, W. L.: Trends Microbiol., 14, 15 (2006).
19) Oliveira, D. L., Nakayasu, E. S., Joffe, L. S., Guimaraes, A. J., Sobreira, T. J., Nosanchuk, J. D., Cordero, R. J., Frases, S., Casadevall, A., Almeida, I. C., Nimrichter, L., Rodrigues, M. L.: PLOS One, 5, e11113 (2010).
20) Chiellini, C., Cochet, O., Negroni, L., Samson, M., Poggi, M., Ailhaud, G., Alessi, M. C., Dani, C., Amri, E. Z.: BMC Mol. Biol., 9, 26 (2008).
21) Yang, C. K., Ewis, H. E., Zhang, X., Lu, C. D., Hu, H. J., Pan, Y., Abdelal, A. T., Tai, P. C.: J. Bacteriol, 193, 5607 (2011).
22) Lamonica, J. M., Wagner, M., Eschenbrenner, M., Williams, L. E., Miller, T. L., Patra, G., DelVecchio, V. G.: Infect. Immun., 73, 3646 (2005).
23) Morisaka, H., Matsui, K., Tatsukami, Y., Kuroda, K., Miyake, H., Tamaru, Y., Ueda, M.: AMB Express, 2, 37 (2012).
24) Shinya, R., Takeuchi, Y., Miura, N., Kuroda, K., Ueda, M., Futai, K.: Nematology, 11, 429 (2009).
25) Shinya, R., Morisaka, H., Takeuchi, Y., Ueda, M., Futai, K.: Phytopathology, 100, 1289 (2010).
26) Miura, N., Kirino, A., Endo, S., Morisaka, H., Kuroda, K., Takagi, M., Ueda, M.: Eukaryot. Cell, 11, 1075 (2012).
27) Baker, D., Hicke, L., Rexach, M., Schleyer, M., Schekman, R.: Cell, 54, 335 (1988).
28) Deshaies, R. J., Schekman, R.: J. Cell Biol., 105, 633 (1987).
29) Kaiser, C. A., Schekman, R.: Cell, 61, 723 (1990).
30) Novick, P., Schekman, R.: Proc. Natl. Acad. Sci. USA, 76, 1858 (1979).
31) Miura, N., Ueda, M.: Methods Mol Biol., 1270, 51 (2015).
32) Katahira, S., Mizuike, A., Fukuda, H., Kondo, A.: Appl. Microbiol. Biotechnol., 72, 1136 (2006).
33) Huh, W. K., Falvo, J. V., Gerke, L. C., Carroll, A. S., Howson, R. W., Weissman, J. S., O'Shea, E. K.: Nature, 425, 686 (2003).
34) Larsen, T. M., Wedekind, J. E., Rayment, I., Reed, G. H.: Biochemistry, 35, 4349 (1996).
35) Mashego, M. R., van Gulik, W. M., Vinke, J. L., Heijnen, J. J.: Biotechnol. Bioeng., 83, 395 (2003).
36) Tsugawa, H., Bamba, T., Shinohara, M., Nishiumi, S., Yoshida, M., Fukusaki, E.: J. Biosci. Bioeng., 112, 292 (2011).
37) Pedley, A. M., Benkovic, S. J.: Trends Biochem. Sci., 42, 141 (2017).