【トピック】
スマートアプタマー 〜グアニン四重鎖構造をもつ核酸の特性の利用〜
西尾真初、池袋一典
東農工大院・工
1.はじめに
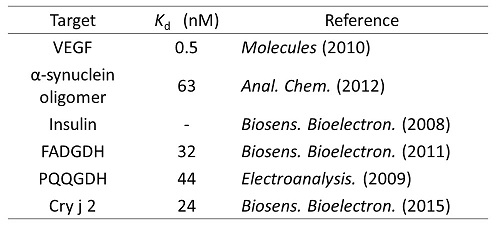
アプタマーとは、タンパク質や小分子など様々な標的分子に特異的に高い結合親和性で結合できる核酸分子である。アプタマーは核酸が形成する特定の二次構造により標的分子を認識する。その中でも我々はグアニン四重鎖構造 (G4; G-quadruplex) に着目している。G4は配列中で4分子のグアニンがHoogsteen型塩基対形成を介して安定な平面、G-quartetを形成し、そのG-quartetが積み重なることで形成される。また、G-quartet中央に1価のカチオン (主にNa+、K+) が配位することで安定化する事が知られている。G4のトポロジーは、parallel型、anti-parallel型、hybrid型と多様であり、G-quartet形成に関与する鎖の方向の組み合わせにより分類されている (図1) 。更に、テロメアに存在するG4は配位するカチオンの種類によって異なるトポロジーを取ることが報告されている1-3)。このように、G4を形成するオリゴヌクレオチドは、同じ塩基配列を有していても周囲の環境によって異なる構造を形成できる。我々は、様々な標的分子に対してアプタマーを獲得しているが、その中にG4を形成する配列が多く存在することから、G4はアプタマーの骨格として優れていると考えている (表1)。
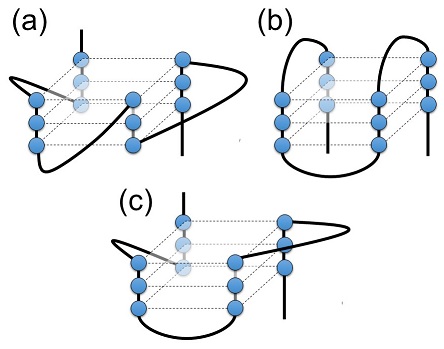
図1 G-quadruplex構造の模式図 (a) parallel型, (b) anti-parallel型, (c) hybrid型。●がグアニンを示す。
表1 我々が獲得したG4形成アプタマーの例
このG4の特性を利用すれば、周囲の環境により標的分子との結合が変化するスマートな分子認識素子としてアプタマーを利用することができる。しかしその反面、周囲の環境によって形成する構造が変化すると、標的分子との結合を評価する場合に再現性が取りづらいというという問題が生じる。よって、その応用にはG4形成アプタマーの構造を制御することが不可欠である。そこで本稿ではG4の特性を利用したアプタマー獲得の例とその構造を制御する手法について紹介し、アプタマーの骨格としてのG4の可能性について展望を述べる。
2.カチオン濃度の変化を利用した細胞内でタンパク質から脱離するアプタマーの獲得
本節では、ゲノム編集技術への応用を志向したFokIヌクレアーゼドメイン (FokI) 結合アプタマーについて紹介する4)。FokIとは、代表的な人工ヌクレアーゼであるジンクフィンガーヌクレアーゼ (ZFN) 及びTranscription activator-like effector nuclease (TALEN) に共通するヌクレアーゼドメインである。このZFNやTALENを遺伝子を改変したい細胞へ直接導入するために、細胞外ではFokIに結合し、細胞内では脱離するアプタマーの獲得を試みた。アプタマーを用いた人工ヌクレアーゼの直接導入には、ナノニードルという細胞へ直接物質輸送を可能にする針を用いる5)。ナノニードルにアプタマーを介して、FokIを有する人工ヌクレアーゼを担持して細胞内へ導入し、アプタマーが細胞内で構造変化することでFokIから脱離することを期待した (図2)。共有結合による固定化と比較して、本手法ではアプタマーの構造変化を利用するため、人工ヌクレアーゼを失活させずに導入することができると考えた。このアプタマーのスクリーニングで着目したのは、細胞内外で濃度差が大きく核酸の構造に影響を与えるNa+及びK+の2種のカチオンである。細胞内、特に人工ヌクレアーゼを導入したい核には、約150 mMと高濃度のK+が存在する。また、このK+はG4の安定化に大きく寄与する。そこで、アプタマーのスクリーニングでは細胞内と大きく異なる環境を選択した。高濃度のNa+中でDNAライブラリーとFokIを結合させる操作を繰り返し、FokIに結合する配列を濃縮した結果、G4を形成する配列が見出され、Kd 数十 nMでFokIに結合する配列が獲得できた。このアプタマーの形成するG4をCDスペクトル測定によって評価した結果、K+とNa+存在下では、parallel型G4に特徴的なピークの強度が異なった。よって、獲得したアプタマーは、周囲に存在するカチオンの種類によって形成するG4の構造が異なると思われる。実際にこのアプタマーを用いて、GFP融合ZFNをナノニードルに担持し、細胞へ導入した結果、細胞存在下でその脱離が観察された。このように、周囲の環境変化とG4の構造変化を利用することで、アプタマーと標的分子の結合や脱離が制御できる。
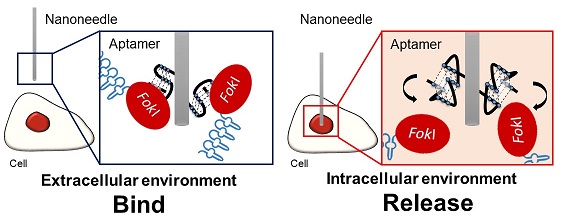
図2 FokI結合アプタマーとナノニードルを利用した実行ヌクレアーゼの細胞への直接導入模式図
3.G4リガンドを用いたG4形成アプタマーの構造制御
構造変化するG4形成アプタマーの欠点は周囲の環境により結合の強さが変化することである。センサー素子としてG4形成アプタマーを用いると、測定値の再現性が悪いことが多い。本節では、アプタマーの形成するG4を、G4に結合する低分子リガンドを使って制御し、結合能を向上させた例を紹介する6)。
αシヌクレインオリゴマー結合アプタマー、T-SO530はG4リガンド非存在下ではparallel型、anti-parallel型、hybrid型など複数のトポロジーのG4が混在している7)。しかし、G4に結合する低分子であるL1H1-7OTD (7OTD)8)存在下では、その構造はanti-parallel型のみと思われるCDスペクトルを示し、αシヌクレインに対する結合シグナルは7OTDを添加することで著しく向上した (図3) 。よってT-SO530のαシヌクレインへの結合にはanti-parallel型のG4構造の形成が重要であることが考えられる。
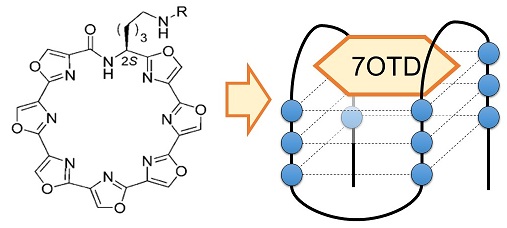
図3 7OTDの構造式 (左) とG4構造への結合模式図 (右)
また、アプタマーの結合能を向上させる手法として、「アプタマーの多価化」が有効である。しかしG4形成アプタマーの場合、ステムループ構造等を形成するアプタマーと比較して、多価化した際に形成しうるG4構造の種類が増えるため正しく構造を形成させるのは難しい。実際に、T-SO530においても5塩基もしくは10塩基のチミンリンカーを介して多価化した際に配列同士が多量体を形成してしまい、結合能の向上は観察されなかった。また、形成するG4も複数のトポロジーを持つ構造体の混合物と考えられた。しかし、この多価化したT-SO530に7OTDを添加して構造を形成させると多量体は形成されなくなり、anti-parallel型のG4が示すCDスペクトルを示した。更に、多価化T-SO530の結合能は7OTD非存在下では単量体とあまり変わらないのに対して、7OTDを作用させると結合能が大きく向上した。このように、7OTDを用いてT-SO530のG4構造をanti-parallel型に収束させることでαシヌクレインへの結合能を大きく向上させることができ、T-SO530の多価化においてもこれは有用であった。
次に、血管内皮細胞増殖因子結合アプタマー、3R02を10塩基のチミンリンカーを介して多価化したアプタマー (biv-3R02) について紹介する9)。biv-3R02は、T-SO530同様に様々な種類のG4構造が混在している。しかし、7OTDを添加すると非存在下と比較してparallel型のG4の形成が顕著になる。このbiv-3R02は、K+非存在下で結合能が大きく下がる。K+はG4形成に作用するため、結合能の減少はG4構造を形成しにくいことに起因すると思われる。しかし、biv-3R02はK+非存在下であっても7OTDを作用させると、K+存在下と同等の結合能を示す。これは、7OTDがbiv-3R02のparallel型のG4形成を促進するためだと考えた。また、他のG4リガンドについても同様の効果が観察された。
本節で紹介した2つのアプタマーは、G4リガンドである7OTD存在下で構造を形成させることによってG4形成が促進され、その結果、各標的分子に対する結合能が向上した。さらに、G4リガンドによる特定のG4形成の促進は、アプタマーの多価化においても有効だった。
4.おわりに
本稿では、周囲の環境変化による構造変化を利用したG4形成アプタマーの獲得とその応用の例を示し、更にG4リガンドを用いた構造制御の例を紹介した。G4形成アプタマーは、周囲の環境によって構造が変化することが利点であり、その反面結合の再現性の無さが課題だった。利点の例として、標的分子への結合と脱離の自発的制御が可能なことをFokI結合アプタマーの例を示した。また、結合の再現性については、アプタマーが形成するG4を標的分子への結合に適したトポロジーへG4リガンドを用いて制御することで克服できることを、複数の種類のアプタマーで示した。周囲の環境によるG4構造の変化を標的分子への結合と脱離に利用し、それがG4リガンドにより制御できれば、目的とする条件で、標的分子への結合を自発的に制御できる、「スマートアプタマー」としての利用が期待できる。
文献
1) Luu, K. N., Phan, A. T., Kuryavyi, V., Lacroix, L., Patel, D. J.: J. Am. Chem. Soc., 128, 9963 (2006).
2) Parkinson, G. N., Lee, M. P., Neidle, S.: Nature, 417, 876 (2002).
3) Wang, Y., Patel, D. J.: Structure, 1, 263 (1993).
4) Nishio, M., Matsumoto, D., Kato, Y., Abe, K., Lee, J., Tsukakoshi, K., Yamagishi, A., Nakamura, C., Ikebukuro, K.: Biosens. Bioelectron., 93, 26 (2017).
5) Obataya, I., Nakamura, C., Han, S., Nakamura, N., Miyake, J.: Nano. Lett., 5, 27 (2005).
6) Tsukakoshi, K., Ikuta, Y., Abe, K., Yoshida, W., Iida, K., Ma, Y., Nagasawa, K., Sode, K., Ikebukuro, K.: Chem. Commun., 52, 12646 (2016).
7) Tsukakoshi, K., Abe, K., Sode, K., Ikebukuro, K.: Anal. Chem., 84, 5542 (2012).
8) Tera, M., Iida, K., Ishizuka, H., Takagi, M., Suganuma, M., Doi, T., Shin-ya, K., Nagasawa, K.: Chembiochem., 10, 431 (2009).
9) Nonaka, Y., Yoshida, W., Abe, K., Ferri, S., Schulze, H., Bachmann, T. T., Ikebukuro, K.: Anal. Chem., 85, 1132 (2013).