【トピックス】
オルガネラ間コンタクトを担うVAP蛋白質の構造と機能
古板恭子、児嶋長次郎
阪大・蛋白研、横国大・理工, 阪大・蛋白研
1.はじめに
真核生物の細胞には、小胞体、ゴルジ体、ミトコンドリアなどの様々な機能を持ったオルガネラ (膜で囲まれた構造体) が存在している。従来、異なる種類のオルガネラは互いに隔絶しており、それぞれ独立して機能していると考えられていた。しかし近年、異なるオルガネラないし細胞膜が互いに10~30 nmに近接して膜接触部位 (MCS) と呼ばれる領域を形成し、これを介して物質の授受などを行っていることが明らかになってきた1)。通常、小胞体は細胞に一つしか含まれないが、管状、袋状など複雑な局所構造を成しながら細胞全体に広がっており、細胞膜のほかゴルジ体、エンドソーム、ミトコンドリアなどのほぼ全てのオルガネラとMCSを形成している1)。
小胞体膜貫通タンパク質、Vesicle-associated membrane protein-Associated Proteins (VAPs) は MCSで機能しているタンパク質である。VAPは酵母からヒトまであらゆる真核生物で保存されており、ほぼ全ての組織で発現している普遍的なタンパク質である2)。哺乳類では、VAP-A/Bの2種類のホモログが存在する2)。VAPは小胞体と細胞膜3)、ゴルジ体4)、エンドソーム5)、ペルオキシソーム6,7)、オートファゴソーム8)及びミトコンドリア9,10)とのMCSにおいて様々なタンパク質と相互作用し、それらを小胞体に局在させることで、MCSを安定化するとともに、脂質輸送タンパク質の足場として働いている。また、脂質輸送や恒常性の維持4,11,12)、神経伝達物質の放出13)、unfolded protein response14)、オートファゴソームの形成8)など、広範囲にわたる細胞機能へのVAPの関与が報告されている。このほか、VAPはいくつかのウイルスタンパク質と結合することが示されており、ウイルスの複製の足場としても利用されていると考えられている15,16)。本稿ではVAPの機能の中核である基質認識について、溶液NMR法を駆使し我々が行なった複合体構造解析の結果17)を中心に解説する。
2.VAPと結合するタンパク質
VAPは細胞質側のN末端領域にMajor Sperm Protein (MSP)ドメイン、中ほどにコイルドコイルドメイン、C末端側に膜貫通領域を持ち、コイルドコイル領域及び膜貫通領域で互いに相互作用し、ホモあるいはヘテロダイマーを形成している (図1A)。
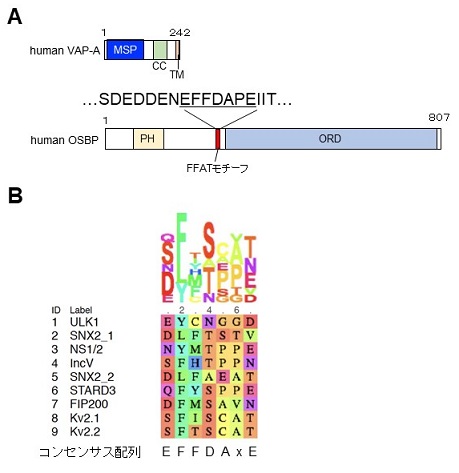

VAPが結合するタンパク質はsoluble N-ethylmaleimide-sensitive factor attachment protein receptor (SNARE) 関連タンパク質とFFATモチーフを含むタンパク質に大別される。VAPはシナプス小胞や細胞膜―小胞体間小胞輸送、ゴルジ体-小胞体間の小胞輸送に関わるSNARE分子と結合することが報告されている18)。また、SNAREの相互作用因子であるSNAP及びNSFとの結合も報告されている18)。SNAREとの相互作用には、膜貫通領域及びN末端領域が重要とされている18)。
FFATモチーフはVAPと相互作用するタンパク質の配列比較により同定されたモチーフで、VAPのMSPドメインに結合する。FFATはtwo phenylalanines (FF) in an Acidic Tractを意味し、EFFDAxEというコンセンサス配列のコア部分とその近傍の一連の酸性残基あるいはSer/ Thr残基からなる19)。FFATモチーフが発見された2003年には、VAPの相互作用相手はまだあまり知られておらず、FFATモチーフは16のVAPの相互作用相手に共通して含まれる配列として同定された19)。近年、VAPの相互作用相手が次々と同定されるにつれ、VAPはFFATモチーフのコンセンサス配列以外の多種多様な配列を結合することが分かってきた (図1B)。これらの配列はFFAT様モチーフと呼ばれている20)。VAP変異体を用いた研究などから、FFAT様モチーフも従来のFFATモチーフもVAPの同じ部位に対して相互作用すると考えられている8,16)。
3.VAP-A : OSBP複合体の立体構造解析
3-1 オキシステロール結合タンパク質 (OSBP) について
我々はVAPによるFFATモチーフの認識機構を明らかにするため、human VAP-A MSPドメイン (VAP-A(5-128)、以下VAP-AMSP) 及びFFATモチーフを含むhuman OxySterol Binding Protein (OSBP)フラグメント (OSBP(346-379)、以下OSBPF) を用い、溶液NMR法によりVAPとFFATモチーフとの相互作用を詳細に解析した。OSBPはオキシステロールの細胞内受容体として同定されたタンパク質で、ステロール類とホスファチジルイノシトール4-リン酸 (PI4P) を共に結合するOSBP関連ドメイン、FFATモチーフ、PI4Pに結合するpleckstrin homologyドメインを有している (図1A)。OSBPは小胞体とゴルジ体とのMCSにおける小胞体由来のコレステロールとゴルジ体由来のPI4Pの交換や、小胞体とエンドソームとのMCSにおけるPI4Pの輸送を担い、脂質膜の恒常性維持に重要な役割を果たしている12,21)。
3-2 複合体立体構造の特徴と分子間相互作用の詳細
最初にVAP-AMSPとOSBPFからなる複合体の立体構造を溶液NMR法により決定した。図2Aに決定した立体構造を示す。VAP-AMSPは7本のβストランドと1本のαヘリックスからなるβサンドイッチ構造を有し、OSBPF。
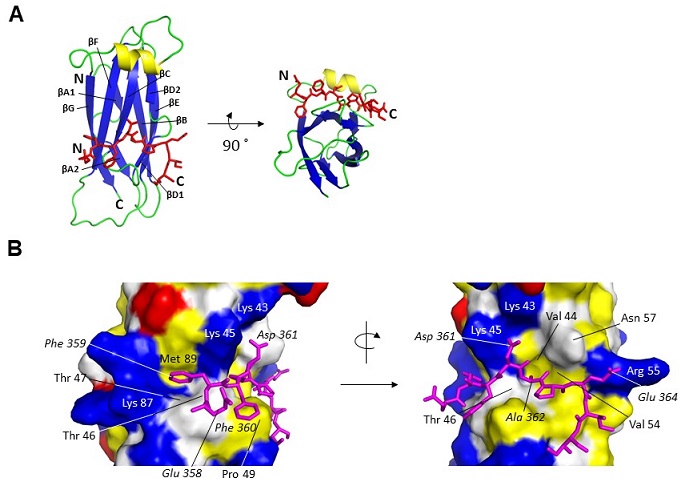

次に、立体構造中のVAP-AMSPとOSBPFの相互作用を詳細に検討した。タンパク質立体構造中の非共有結合性の相互作用を検出するソフトウェアMONSTER22)を用いて解析したところ、OSBPFはFFATモチーフおよびそのC末端側でVAP-AMSPと相互作用していた (図2B)。OSBPFのFFATモチーフ2残基目のPhe-359の側鎖は、VAP-AMSPのLys-45、Thr-47、Lys-87, 及びMet-89から形成される疎水性のポケットにはまり込んでいた。また、5番目のAla-362の側鎖はVal-44、Thr-46、Val-54及びAsn-57からなる疎水性のポケットに認識されていた。FFATモチーフの3番目のPhe-360はVAP-AMSPのPro-49と疎水性の相互作用をしていた。これらの他、OSBPFのPro-363、Glu-364、Ile-365及びIle-366とVAP-AMSPとの間にも疎水性相互作用が見られた。OSBPFのAsp-361及びGlu-362はVAP-AMSPのLys-43/ Lys-45及びArg-55とそれぞれ静電的に相互作用していた。さらに、OSBPFのPhe-360及びPro-362とVAP-AMSPのThe-46及びVal-54の主鎖間には水素結合が見られた。一方で、FFATモチーフ1つ目のGlu-358とVAP-AMSPの間には相互作用が見られなかった。
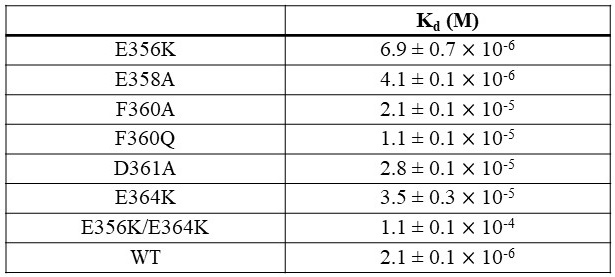
構造中で見られた相互作用を検証するため、一連のOSBPFの変異体を作成し、VAP-AMSPとの相互作用の強さを比較した。定量的に相互作用を評価するため、等温滴定カロリメトリー (ITC) を用いた。決定した一連のOSBPF野生型及び変異体とVAP-AMSPとの解離定数を表1に示す。VAP-AMSPと側鎖による直接的な相互作用が見られた残基の変異体 (F360A、D361A及びE364K) は野生型に比べて相互作用が1/10程度と大幅に減少しており、立体構造解析で示された相互作用の存在が支持された。一方、相互作用が見られなかった残基の変異体 (E356K及びE368A) では、親和性が1/2-1/3程度に低下した。
表1 ITC測定により得られたVAP-AMSPとOSBPF (変異体及びWT) との解離定数解離定数
3-3 VAP-A:OSBP複合体形成過程の解析
前述のように、FFATモチーフはコンセンサス配列のN末端側に一連の酸性残基を持つという特徴がある。OSBPの場合はDEDDENという配列を有する。我々が決定した立体構造では、これらの酸性残基とVAP-AMSPとの間に直接的な相互作用は見られなかった。また、FFATモチーフコンセンサス配列1残基目のGluとVAP-AMSPと間にも直接的な相互作用は見られなかった。しかしながらITC測定の結果、これらの酸性残基の変異体ではVAP-AMSPとの親和性が野生型と比べて低下していることが分かった。このことから、これらの酸性残基はVAP-AMSPと直接には相互作用しないものの何らかの役割があると考えられた。
そこでNMR滴定実験により、VAP-AMSPとOSBPFとの複合体形成過程を調べることにした。NMR滴定実験では、レセプターにリガンドを添加したときのレセプターのNMRスペクトルをモニターするが、レセプターのNMRスペクトルの変化の様子から、リガンドの結合の有無や、レセプター上のリガンド結合部位の情報を得ることができる。さらに、リガンドを段階的に添加した際のスペクトル変化を解析することで、解離定数や、反応中間体の形成などの結合過程の情報を得ることができる。図3Aに15N標識VAP-AMSPに対して非標識OSBPFを段階的に加えたときのVAP-AMSPの1H-15N HSQCスペクトルの変化を示す。多くの残基はフリーと複合体の2状態平衡を示す典型的なピーク変化を示したが、いくつかの残基においては特徴的なピークの変化が認められた。滴定実験におけるNMRスペクトル変化のパターンは、フリーと複合体の交換レート(kex)と信号の周波数差によって決まる。様々なkexを仮定して2状態平衡におけるピーク変化をシュミレーションし、実験結果を検討したが、4つの残基についてはピーク変化のパターンが2状態平衡では説明ができなかった (図3A)。従って、VAP-AMSPはOSBPFとの相互作用において、フリーとも最終の複合体とも異なる何らかの中間複合体を形成すると考えられた。次にOSBPFのE356K変異体を用いて同様の実験を行なった。その結果、2状態平衡で説明できなかった残基のうち、2残基については特殊なピーク変化が消失した (図3B)。このことから、Glu-356は結合中間体の形成に寄与すると考えられた。
以上の結果と、OSBPFの運動性の解析結果などから、OSBPのFFATモチーフ周辺は単独では明確な構造を持たない天然変性状態にあり、OSBPとVAP-Aとの複合体形成では、まずFFATモチーフN末端側の酸性残基とVAP-Aの塩基性領域との静電的相互作用による何らかの中間体が形成され、その後に安定な複合体へと移行するという、いわゆるfly-casting経路23)をたどると結論づけた。
4.FFATモチーフ各残基の役割
本研究により、FFATモチーフ各残基の役割が明らかになった。N末端側の酸性残基群はVAPとの中間複合体の形成に寄与すると考えられた。コンセンサス配列のうち、2番目のPhe及び5番目のAlaはVAP-Aの疎水性のポケットに堅固に認識されており、相互作用の要だと考えられた。また、4番目のAspと7番目のGluはVAP-Aと静電的相互作用を形成することから同じく重要だと考えられた。一方で3番目のPheはVAP-Aと疎水性の相互作用を示すものの側鎖が外側に配向しており、他の疎水性残基でもその役割を果たせる予想された。さらに1番目のGluはVAP-Aとの直接の相互作用は認められず、さほど重要ではないと考えられた。この見解とFFAT様モチーフに見出される配列の多様性は必ずしも一致しない (図1B)。不一致の原因しては、FFAT様モチーフとVAPの親和性が従来のFFATモチーフのそれに比べて低いためないし、両モチーフの相互作用様式が異なるためと思われる。FFAT様モチーフとVAPとの相互作用様式の解明は今後の課題である。
5.溶液NMRによるタンパク質解析技術の進展
最後に、溶液NMR法によるタンパク質解析の最近の進展について簡単に紹介したい。溶液NMR法は、溶液中という生体内に近い環境下において、原子分解能でタンパク質の運動性や立体構造情報が得られる優れた手法である。しかしながら、解析が難解で時間を要するといった問題があり、専門外の研究者にはあまり利用されてこなかった。
近年の技術の進歩により、タンパク質のNMRスペクトルの解析には大幅な自動化がもたらされている。現在では、性質の良い100残基程度の小型タンパク質であれば、専門家でなくともわずか数時間でNMR信号を帰属し、立体構造モデルを得ることが可能である。Güntertらにより開発された自動シグナル帰属プログラムFLYAは、タンパク質のシグナル帰属と立体構造計算を関連付けて実行することで、高精度の帰属と立体構造決定を可能にした24)。小林直宏博士 (大阪大学蛋白質研究所、現理化学研究所) によって開発されたMagROプラットフォームはFLYAと連携しており、FLYAへの入力ファイルの半自動的作成や、計算結果をスペクトル上に表示する機能を備え、FLYAを用いた解析に要する手間と時間を大幅に削減した。特に、機械学習の一種である畳み込みニューラルネットワークを利用したfilt_robotと呼ばれる最新のプログラムは、高ノイズスペクトルから高精度にNMR信号を選抜することができる25)。これまでは熟練者に依存せざるを得なかった信号選抜作業が自動化されたことにより、初心者でも短時間でNMRスペクトルを成しえる環境となっている。NMRを活用していなかった研究者にも溶液NMRが普及し、さらなる研究の進展がもたらされることを期待したい。
謝辞
本稿で取り上げた研究は、三島正規准教授 (首都大学東京)、池晙求教授 (Kyungpook National University)、深田はるみ准教授 (大阪府立大学)、木下紘子技術員、米山桃子技術員のご協力を得て実施しました。この場を借りてお礼申し上げます。
文献
1) Wu, H., Carvalho, P. & Voeltz, G. K.: Science, 361, eaan5835 (2018).
2) Lev, S., Halevy, D. Ben, Peretti, D., Dahan, N.: Trends Cell Biol., 18, 282 (2008).
3) Kim, Y. J., Guzman-Hernandez, M.-L., Wisniewski, E., Balla, T.: Dev. Cell, 33, 549 (2015).
4) Peretti, D., Dahan, N., Shimoni, E., Hirschberg, K., Lev, S.: Mol. Biol. Cell, 19, 3871 (2008).
5) Wilhelm, L. P., Wendling, C., Vedie, B., Kobayashi, T., Chenard, M., Tomasetto, C., Drin, G., Alpy, F.: EMBO J., 36, 1412 (2017).
6) Costello, J. L., Castro, I. G., Hacker, C., Schrader, T. A., Metz, J., Zeuschner, D., Azadi, A. S., Godinho, L. F., Costina, V., Findeisen, P., Manner, A., Islinger, M., Schrader, M.: J. Cell Biol., 216, 331 (2017).
7) Hua, R., Cheng, D., Coyaud, E., Freeman, S., Di Pietro, E., Wang, Y., Vissa, A., Yip, C. M., Fairn, G. D., Braverman, N., Brumell, J. H., Trimble, W. S., Raught, B., Kim, P. K.: J. Cell Biol., 216, 367 (2017).
8) Zhao, Y. G., Liu, N., Miao, G., Chen, Y., Zhao, H., Zhang, H.: Curr. Biol., 28, 1234 (2018).
9) De vos, K. J., Morotz, G. M., Stoica, R., Tudor, E. L., Lau, K. F., Ackerley, S., Warley, A., Shaw, C. E., Miller, C. C. J.: Hum. Mol. Genet., 21, 1299 (2012).
10) Gomez-Suaga, P., Paillusson, S., Stoica, R., Noble, W., Hanger, D. P., Miller, C. C. J.: Curr. Biol., 27, 371 (2017).
11) Kawano, M., Kumagai, K., Nishijima, M., Hanada, K.: J. Biol. Chem., 281, 30279 (2006).
12) Dong, R., Saheki, Y., Swarup, S., Lucast, L., Harper, J. W., De Camilli, P.: Cell, 166, 408 (2016).
13) Skehel, P., Martin, K. C., Kandel, E. R., Bartsch, D.: Science, 269, 1580 (1995).
14) Kanekura, K., Nishimoto, I., Aiso, S., Matsuoka, M.: J. Biol. Chem., 281, 30223 (2006).
15) Hamamoto, I., Nishimura, Y., Okamoto, T., Aizaki, H., Liu, M., Mori, Y., Abe, T., Suzuki, T., Lai, M. M. C., Miyamura, T., Moriishi, K., Matsuura, Y.: J. Virol., 79, 13473 (2005).
16) McCune, B. T., Tang, W., Lu, J., Eaglesham, J. B., Thorne, L., Mayer, A. E., Condiff, E., Nice, T. J., Goodfellow, I., Krezel, A. M., Virgin, H. W.: MBio, 8, e00668 (2017).
17) Furuita, K., Jee, J., Fukada, H., Mishima, M., Kojima, C.: J. Biol. Chem., 285, 12961 (2010).
18) Weir, M. L., Xie, H., Klip, A., Trimble, W. S.: Biochem. Biophys. Res. Commun., 286, 616 (2001).
19) Loewen, C. J. R., Roy, A., Levine, T. P., EMBO J., 22, 2025 (2003).
20) Murphy, S. E., Levine, T. P.: Biochim. Biophys. Acta, 1861, 952 (2016).
21) Mesmin, B., Bigay, J., Moser von Filseck, J., Lacas-Gervais, S., Drin, G., Antonny, B.: Cell, 155, 830 (2013).
22) Salerno, W. J., Seaver, S. M., Armstrong, B. R., Radhakrishnan, I.: Nucleic Acids Res., 32, W566 (2004).
23) Shoemaker, B. A., Portman, J. J., Wolynes, P. G.: Proc. Natl. Acad. Sci., 97, 8868 (2000).
24) Schmidt, E., Guntert, P.: J. Am. Chem. Soc., 134, 12817 (2012).
25) Kobayashi, N., Hattori, Y., Nagata, T., Shinya, S., Guntert, P., Kojima, C., Fujiwara, T.: Bioinformatics, 34, 4300 (2018).
26) Stanhope, R., Flora, E., Bayne, C., Derre, I.: Proc. Natl. Acad. Sci., 114, 201709060 (2017).
27) Johnson, B., Leek, A. N., Sole, L., Maverick, E. E., Levine, T. P., Tamkun, M. M.: Proc. Natl. Acad. Sci., 115, 201805757 (2018).
28) Yachdav, G., Wilzbach, S., Rauscher, B., Sheridan, R., Sillitoe, I., Procter, J., Lewis, S. E., Rost, B., Goldberg, T.: Bioinformatics, 32, btw474 (2016).