【トピックス】
芳香族化合物前駆体2-deoxy-scyllo-inosose (DOI) 合成酵素の高機能化
高久洋暁、岩澤裕喜、大内秀紀、山崎晴丈、和田光史
新潟薬大・応生科、新潟薬大・応生科、新潟薬大・応生科、新潟薬大・応生科、三井化学
1.はじめに
現代社会の豊かさの礎の1つは化石資源である。石炭や石油などの化石資源を活用した技術革新は、大量生産・大量消費スタイルの社会を作り出し、我々はそこから豊かさを手に入れてきた。化石資源を原料とする多数の製品が、社会の様々な場面で活用されており、原油 (ナフサ) を分解・加工したエチレンやフェノール類などから生産されている石油化学製品もその1つである。しかしながら、世界的な大量生産・大量消費スタイルは、化石資源の枯渇と二酸化炭素などの温室効果ガスの排出による地球温暖化問題を引き起こしている現状がある。このような課題に対して、2019年12月にスペインのマドリードで行われた国連気候変動枠組み条約第25回締約国会議 (COP25) のように国際的且つ政治的な取り組みが継続的に行われている。さらにその取り組みの目標に対して、技術的な面でも応答し、これまでの産業・社会構造を変革し、持続可能な社会を構築する必要性がある。
従来の高温、高圧下などの高エネルギー条件下で行われることが多く、環境負荷が高い化学プロセスの代わりに、カーボンニュートラルである再生可能なバイオマス資源を出発原料とし、常温常圧の温和な条件で反応する微生物や動植物等の生体触媒 (酵素) を活用したバイオプロセスは、持続可能な産業構造の構築に必要な技術の1つであると考えられる。
我々のグループでは、炭素六員環骨格を持つキラルな化合物である芳香族化合物前駆体2-deoxy-scyllo-inosose (DOI) に注目して研究を行っている。DOIは様々な医薬品、農薬、健康食品、化成品などの原材料として利用できる物質であり、特に、芳香族化合物の2価フェノールであるカテコールやヒドロキノン、3価フェノールであるヒドロキシヒドロキノン (1, 2, 4-トリヒドロキシベンゼン) などへ簡単に化学合成変換することができる有用化合物である1,2)。さらに、DOIは擬似糖のカルバグルコースにも変換ができ、用途の広い化合物である3)。しかしながら、工業利用するためのDOI大量生産技術が確立していないため実用化には至っていない。DOIはmyo-inositolを出発物質として9ステップで化学合成可能だが、収率が非常に低く4)、化学合成によるDOIの大量生産は困難である。しかしながら、1999年、東工大の柿沼らにより発見されたDOI合成酵素 (グルコース-6-リン酸を基質としてDOIへ変換する酵素) は、DOI大量生産の道を切り開いた5,6)。我々は、ブチロシン生産菌Bacilluscirculans SANK72073から単離されたDOI合成酵素遺伝子 (BtrC) をグルコース-6-リン酸が高蓄積するように代謝工学的改変を加えた大腸菌 (ΔpgiΔzwfΔpgm) に導入した7)。グルコースは大腸菌体内に取り込まれると同時にリン酸化されグルコース-6-リン酸となる。グルコース-6-リン酸は、解糖系、ペントースリン酸経路、多糖合成系の3経路で利用される。DOI高生産大腸菌はそれぞれの経路の酵素をコードする遺伝子pgi, zwf, pgmを欠失させ、菌体内にグルコース-6-リン酸を基質とする酵素がDOI合成酵素のみとした組換え大腸菌 (ΔpgiΔzwfΔpgm/BtrC) である。本組換え大腸菌はグルコースを生育炭素源として利用できないため、本組換え菌を生育させるためにキシロース、フルクトース、マンニトール、グリセロールなどのグルコース-6-リン酸より下流から解糖系へ流れる経路を使う生育炭素源を添加する必要がある。このDOI高生産大腸菌は、原料となるグルコースを99%の変換効率でDOIに変換させることのできる大腸菌である (図1)。
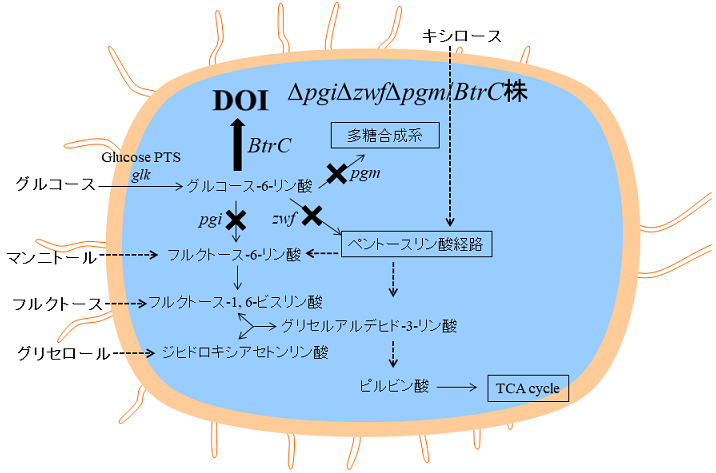
図1 DOI高生産組換え大腸菌の炭素源代謝マップ
2.DOI合成酵素の高機能化
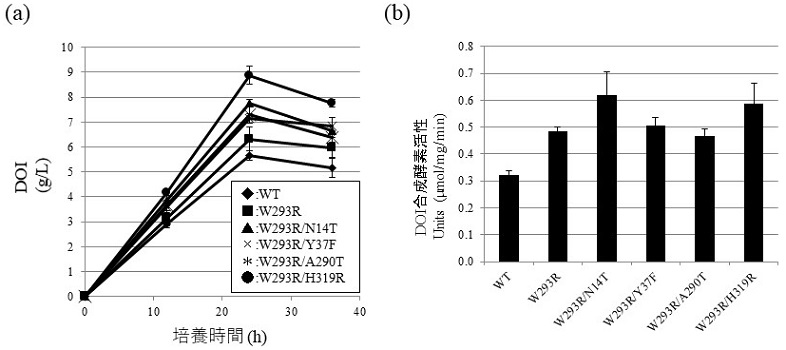
DOIの実用化へ向けた次の課題としてDOI生産速度の向上があげられた。そこで、進化工学的改変で高活性型DOI合成酵素を取得し、活用することを考えた。図2に示したスキームのように、変異型DOI合成酵素遺伝子ライブラリーを構築し、そのライブラリーを利用して高活性型DOI合成酵素遺伝子のスクリーニングを行った。具体的には、機能を向上させる目的遺伝子であるBtrCにエラープローンPCR法でランダムに変異を導入し、発現ベクターに連結して遺伝子ライブラリーを作製した。作製した遺伝子ライブラリーを大腸菌から抽出後、DOI合成酵素の基質であるグルコース-6-リン酸を高蓄積するΔpgiもしくはΔpgiΔzwfΔpgm株に形質転換し、コロニーを得た。得られたコロニーの前培養、本培養を行い、経時的に培地中のDOI濃度を測定することにより、DOI生産速度が向上した組換え大腸菌の選抜を行った。その結果、DOI生産速度が向上した6株の組換え大腸菌を得た。それらの組換え大腸菌から抽出されたプラスミド上のBtrC遺伝子のDNAシーケンス解析をした結果、全てのBtrC遺伝子で異なるミスセンス変異が見いだされた。それらのミスセンス変異は全て活性中心から大きく離れた場所に存在し、特にDOI生産速度向上の効果が大きかったのは、BtrC遺伝子の877番目のチミン (T) がアデニン (A) に置換されたことにより、293番目のトリプトファン (W) が塩基性アミノ酸のアルギニン (R) に置き換わったBtrC (W293R) であった。培養開始24時間後のDOI生産量は約1.12倍の増加であった (図3(a))。また、野生型 (WT) 及び変異型DOI合成酵素 (W293R) の酵素にHisタグが付加した組換え蛋白質を大腸菌で生産後、精製し、in vitro におけるDOI合成酵素活性の比較を実施した。その結果、野生型DOI合成酵素と比較して、W293Rは約1.5倍のDOI合成酵素活性を示したことからDOI合成酵素活性の向上がDOI生産速度の向上に繋がったと考えられた (図3(b))。
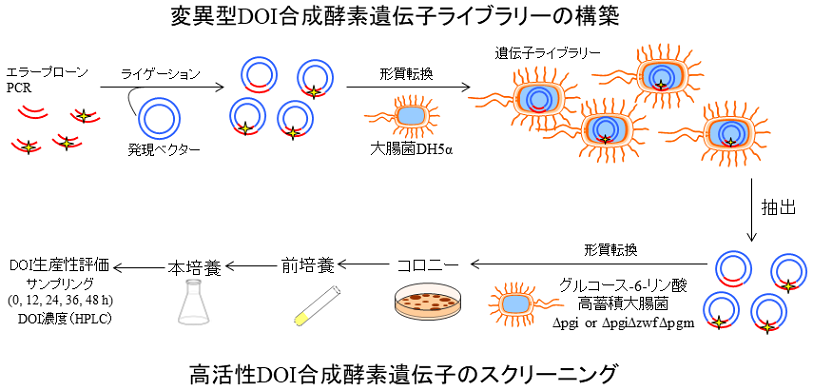
図2 高活性型DOI合成酵素遺伝子探索スキーム
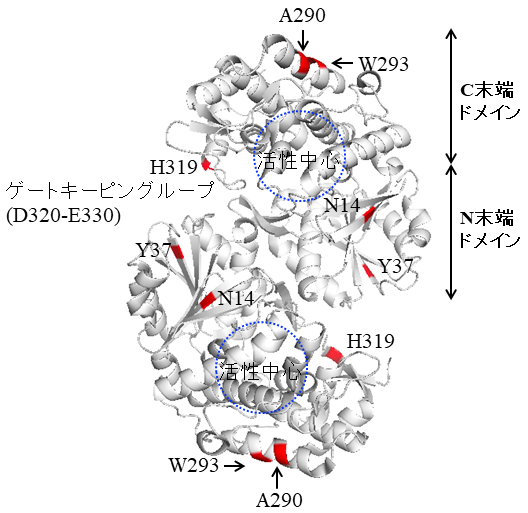
また、東工大の南後らによって、2005年に天然型DOI合成酵素の結晶構造が報告され8)、さらに2008年にDOI合成酵素、基質アナログ、補酵素の複合体の結晶構造が明らかにされた。結晶構造解析結果よりDOI合成酵素は、N末端ドメインとC末端ドメインの2つの大きなドメインで構成され、ホモダイマーを形成していることが明らかとなった9)。N末端ドメイン (2-172) には、NAD+結合ドメインモチーフ (Rosssmann-fold) が、C末端ドメイン (173-368) は、6つのα-ヘリックス、1つのショートα-ヘリックス、4つのβ-ストランドから構成されている。293番目のアミノ酸残基は、活性中心、さらには基質の取り込み等に関与すると考えられているゲートキーピングループから離れたところに位置していることから (図4)、そのDOI合成酵素活性の向上に繋がった効果の推察が困難であった。DOI合成酵素の高活性化に寄与する変異部位は、活性中心付近やゲートキーピングループ付近である可能性を考えながらスクリーニングしたが、さらなる高活性な変異型DOI合成酵素は取得できなかった。
そこで、さらなる高活性な変異型DOI合成酵素の取得に向け、BtrC (W293R) 遺伝子を鋳型として、エラープローンPCR法でランダム変異の導入を試みた。先と同様に、遺伝子ライブラリーの作製、大腸菌ΔpgiΔzwfΔpgm株に形質転換しスクリーニングを実施した結果、培養開始24時間後のDOI生産量がBtrC (W293R) よりも増加した組換え大腸菌を4株見いだした。組換え大腸菌から抽出されたプラスミド上のBtrC遺伝子のDNAシーケンス解析をした結果、W293Rに加えてさらにミスセンス変異を持つ4種類の変異型BtrC遺伝子 (W293R/N14T, W293R/Y37F, W293R/A290T, W293R/H319R) が見いだされた10)。それらの組換え大腸菌の培養開始24時間後のDOI生産量は、野生株と比較して、BtrC (W293R/N14T) は約1.37倍、BtrC (W293R/Y37F) は約1.29倍、BtrC (W293R/A290T) は約1.29倍、BtrC (W293R/H319R) は約1.57倍であった (図3(a))。さらに、新たに取得した変異型DOI合成酵素にHisタグを付加した組換え蛋白質を生産後、精製し、in vitro におけるDOI合成酵素活性の比較を実施した。野生型DOI合成酵素と比較して、BtrC (W293R/N14T) は約2.0倍、BtrC (W293R/Y37F) は約1.6倍、BtrC (W293R/A290T) は約1.5倍、BtrC (W293R/H319R) は約1.9倍のDOI合成酵素活性を示した (図3(b))。以上のことよりDOI合成酵素活性の向上がDOI生産速度の向上に繋がったと考えられた。新たに見いだされた変異部位N14、Y37はN末端ドメイン上、A290はW293に隣接した部位、H319はゲートキーピングループに隣接した部位であり (図4)、特にH319は基質であるグルコース-6-リン酸との親和性に大きく関与している可能性が考えられた。そこでDOI生産速度及びDOI合成酵素活性を大きく向上させたW293RとH319RのDOI合成酵素特性への影響を明らかにするため速度論的解析を実施した。変異型DOI合成酵素W293R、H319R、W293R/H319Rを生産する組換え大腸菌を作製し、各精製酵素の基質 (グルコース-6-リン酸) 親和性Km値を評価したところ、変異型DOI合成酵素W293R、H319R、W293R/H319RのKm値は、野生型DOI合成酵素のKm値の0.9倍、0.9倍、0.8倍程度小さい値を示したことから、変異型DOI合成酵素W293R、H319R、W293R/H319Rの基質親和性が高くなったことが明らかとなった。また、酵素の代謝回転能を示すKcat値は、変異型DOI合成酵素H319Rでは、野生型DOI合成酵素よりも約1.1倍大きい値を示したが、変異型DOI合成酵素W293R、W293R/H319Rは野生型DOI合成酵素よりも 0.94倍程度小さい値を示した。総合的な酵素の能力を示すKcat/Km値は、野生型DOI合成酵素よりも1.02倍 (W293R)、1.13倍 (H319R)、1.21倍 (W293R/H319R) 程度大きな値を示した。すなわち、W293R及びW293R/H319Rは基質親和性に、H319Rは基質親和性と代謝回転能に影響を与えていると考えられた。W293R/H319Rは基質親和性に大きな影響を与えたことによりDOI合成酵素活性が向上し、その結果DOI生産速度が向上したと推測された。
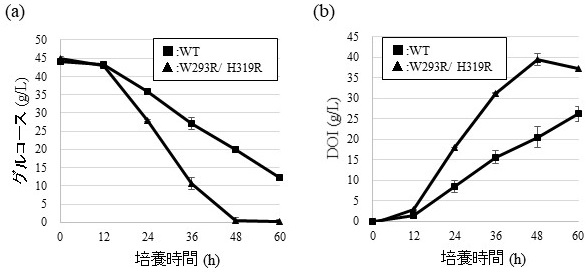
今回作製した形質転換体 (ΔpgiΔzwfΔpgm/BtrC (W293R/H319R) ) を、生育炭素源としてマンニトールを添加し、フラスコスケールによるDOI発酵生産実験を行ったところ、培養開始48時間後に初発グルコース濃度45 g/Lは枯渇し、DOI濃度は39.4 g/Lとなった。野生型DOI合成酵素遺伝子を導入した組換え大腸菌ΔpgiΔzwfΔpgm/BtrCの培養開始48時間後のDOI濃度 (20.6 g/L) と比較して、約2倍DOI生産速度が向上していた (図5)10)。グルコースからDOIのモル変換率は、98.5%であることから、BtrC (W293R/H319R) は、変換効率へ影響は与えずにDOI生産速度を約2倍向上させる高活性型DOI合成酵素であることが明らかとなった。

3.おわりに
カーボンニュートラルのバイオマス資源から石油化学製品の代替となるクリーンな化成品等を製造するホワイトバイオテクノロジーは、我々が持続可能な社会を形成していくための技術として、二酸化炭素削減の観点からも今後必要な技術である。特に地球上で最も豊富に存在する天然高分子セルロースの原料としての活用は、ホワイトバイオテクノロジーの鍵となる。芳香族化合物などの目的化合物が微生物に毒性を示し、微生物による大量発酵生産が難しい場合は、DOIのように目的化合物の前駆体 (芳香族化合物前駆体) を微生物に発酵生産させ、その後、化学変換等により目的化合物を生産する方法が有効であると考えられる。今後、DOIが化学工業原料として利用されていくためには、2価フェノールのヒドロキノンやカテコールの原料としてだけでなく、有用なファインケミカル製品などの原料のように、多岐にわたって利用される原料でなければならず、DOIの用途開発の必要性は高いと考えている。
謝辞
実験にご協力頂いた新潟薬科大学鈴木涼子氏、脇坂直樹氏、荒学志氏に感謝する。本稿で紹介した研究の一部は独立行政法人新エネルギー・産業技術総合開発機構の「非可食性植物由来化学品製造プロセス技術開発」の助成を受けて行われたものである。
文献
1) Kakinuma, K., Nango, E., Kudo, F., Matsushima, Y., Eguchi, T.: Tetrahedron Lett., 41, 1935 (2000).
2) Hansen, C. A., Frost, J. W.: J. Am. Chem. Soc., 124, 5926 (2002).
3) Ogawa, S., Asada, M., Ooki, Y., Mori, M., Itoh, M., Korenaga, T.: J. Carbohydr. Chem., 24, 677 (2005).
4) Yamauchi, N., Kakinuma, K.: J. Antibiot., 45, 756 (1992).
5) Kudo, F., Hosomi, Y., Tamegai, H., Kakinuma, K.: J. Antibiot., 52, 81 (1999).
6) Kudo, F., Tamegai, H., Fujiwara, T., Tagami, U., Hirayama, K., Kakinuma, K.: J. Antibiot., 52, 559 (1999).
7) Kogure, T., Wakisaka, N., Takaku, H., Takagi, M.: J. Biotechnol., 129, 502 (2007).
8) Nango, E., Kumasaka, T., Sato, T., Tanaka, N., Kakinuma, K., Eguchi, T.: Acta Cryst., F61, 709 (2005).
9) Nango, E., Kumasaka, T., Hirayama, T., Tanaka, N., Eguchi, T.: Proteins., 70, 517 (2008).
10) Takaku, H., Yamazaki, H., Wada, M., Miyazawa, D.: PCT/JP2018/010349.