【トピックス】
ポスト抗体医薬:免疫チェックポイントを制御するヘリックス-ループ-ヘリックスペプチド—マイクロ抗体—
藤原大佑、道上雅孝、藤井郁雄
阪府大院・理学
1.はじめに
ヒトゲノムには、タンパク質-タンパク質相互作用 (Protein-protein Interaction: PPI) が65万種類存在とすると予測されている1)。疾患に関わるPPIを特異的に制御する阻害剤は、医薬品として期待される2)。しかし、このような明確な目標があるにもかかわらず、PPIを阻害する低分子阻害剤の創出は未だ困難な課題である3)。これは、PPI相互作用面が広く、低分子化合物では十分な結合活性と特異性が得られないためである。抗体はPPIを阻害できる点で際立った利点をもち、また生体内安定性が高い。PPIを阻害する医薬品として、抗体医薬品の開発が成功を収めてきた4)。しかしながら、創薬という観点でいえば、抗体医薬は必ずしも万能ではない。抗体は分子量が15万と大きく、細胞内へ入らないため、細胞外のPPIを標的とした医薬品開発に限られる。また、抗体は培養細胞を用いた一連の製造設備を必要とするため製造コストが下がらないという課題を有している5)。そこで、抗体にかわるPPI阻害剤として、非免疫グロブリン構造をもつタンパク質を基盤とした新規PPI阻害剤の創出が試みられている。しかしながら、これらのタンパク質の化学合成は一般に難しい6)。より小さいペプチドであれば化学合成が可能であり、抗体医薬品よりも安価である。このことは、例えば36アミノ酸からなる抗HIVペプチド医薬品FUZEONなどで示されている7)。
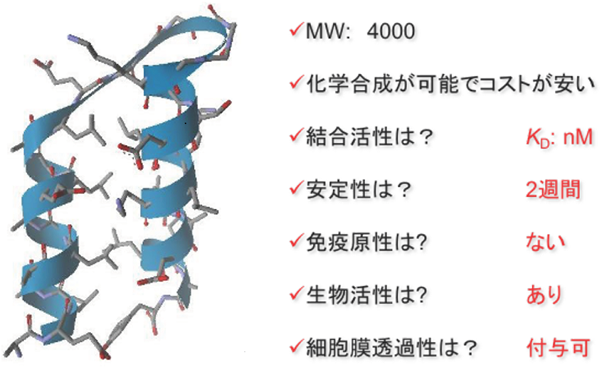
我々はこれまでに、PPI阻害活性を示して抗体様機能をもつ立体構造規制ペプチド「マイクロ抗体」 (分子量:約4000) を創出してきた (図1)8)。進化分子工学の代表的手法である、ファージ表層提示法を用いてライブラリーを作製し、顆粒球コロニー形成刺激因子受容体 (G-CSF-R)、血管内皮細胞増殖因子 (VEGF)、IgGFcドメイン、プロテインキナーゼなどに対する分子標的HLHペプチドを獲得してきた9-11)。さらに、p53由来の結合エピトープをHLHペプチドにグラフティングし、細胞膜透過性を付与することで、細胞内PPIであるp53-HDM2相互作用を阻害するマイクロ抗体cHLHp53-Rの創出にも成功している12)。中でも、本稿では、酵母表層提示法を用いたライブラリー・スクリーニングによって得た、免疫チェックポイントを制御して免疫細胞を活性化できるマイクロ抗体ERY2-4について紹介する13)。
2. 免疫チェックポイントを制御するマイクロ抗体ERY2-4
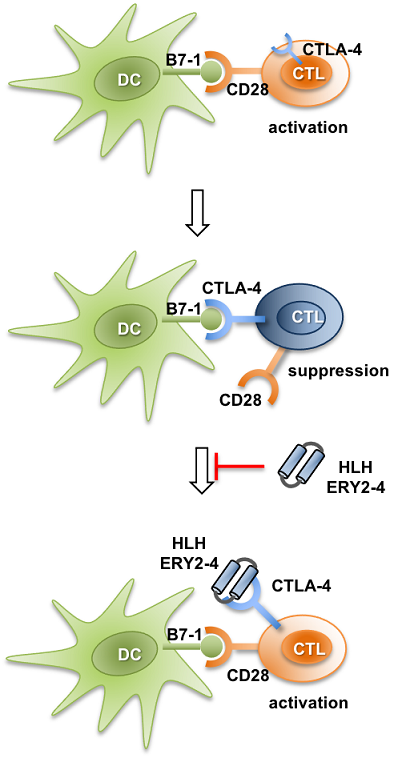
我々は、細胞傷害性Tリンパ球抗原4 (CTLA-4) と抗原提示細胞上のB7との相互作用を阻害し、免疫細胞を活性化させるマイクロ抗体を創出した (図2)。CTLA-4は、T細胞の表面に発現する免疫チェックポイント受容体の1つで、がん細胞はCTLA-4経路を利用し、免疫応答の進行を抑制する14,15)。樹状細胞から腫瘍抗原の提示を受けて活性化したT細胞表面には、CTLA-4が発現し、樹状細胞B7に結合することで、T細胞の活性化が抑制される。抗CTLA-4分子標的ペプチドは、CTLA-4に結合することでB7との結合を阻害し、T細胞活性化を増強・持続し、活性化した抗腫瘍T細胞を誘導することが期待される。
本研究では、酵母表層提示マイクロ抗体ライブラリーを作製して、組み換えタンパク質CLTA-4に対するスクリーニングを行った。さらに、得られたペプチドの親和性成熟を行い、CTLA-4に対してKD値 196 nMで結合するHLHペプチドERY2-4を得た。ERY2-4は、CTLA-4/B7相互作用を選択的に阻害し、さらに免疫細胞を活性化した。用いた酵母表層提示マイクロ抗体ライブラリーについて述べ (3)、CTLA-4に対するスクリーニングついて説明する (4)。さらに、得られたマイクロ抗体ERY2-4が示す生物活性を紹介する (5)。
3.酵母表層提示マイクロ抗体ライブラリー
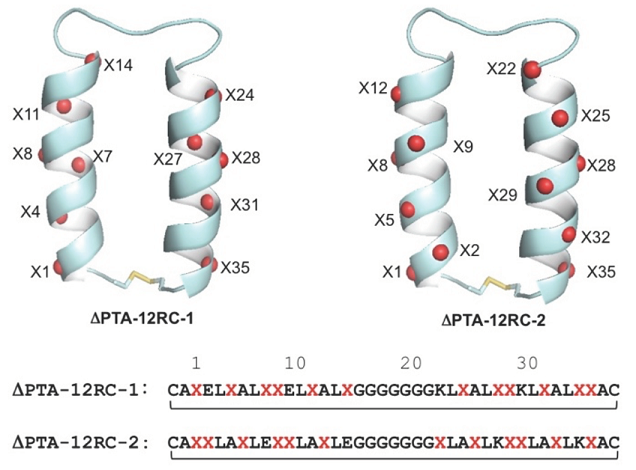
本研究では、2種のマイクロ抗体ライブラリー (ΔPTA-12RC-1とΔPTA-12R-2) を作製してライブラリー・スクリーニングに用いた (図3)。
マイクロ抗体は、ヘリックス-ループ-ヘリックス (HLH) 構造を基盤とする、立体構造規制ペプチドである。HLHペプチドは3つの領域で構成される。N末端側から順に、14アミノ酸からなる構造支持領域、7残基のグリシンからなるループ、14アミノ酸からなるC末端側の構造支持領域である。α -ヘリックス構造は、それぞれの構造支持領域に配したLeu基の疎水相互作用および、Glu基とLys基の間の静電相互作用により会合し、安定なヘリックス-ループ-ヘリックス構造を形成している。本稿で用いた酵母表層提示マイクロ抗体ライブラリーは、HLHペプチドをさらにジスルフィド結合で分子内環化させた、環状化HLH (cHLH) ペプチドYT1-Sを土台分子としている10)。図3に示す通り、一つのcHLHペプチド中の、α-ヘリックス構造をもつ2箇所の領域中のアミノ酸をそれぞれランダム化している。
4.CTLA-4結合性cHLHペプチドERY2-4の獲得
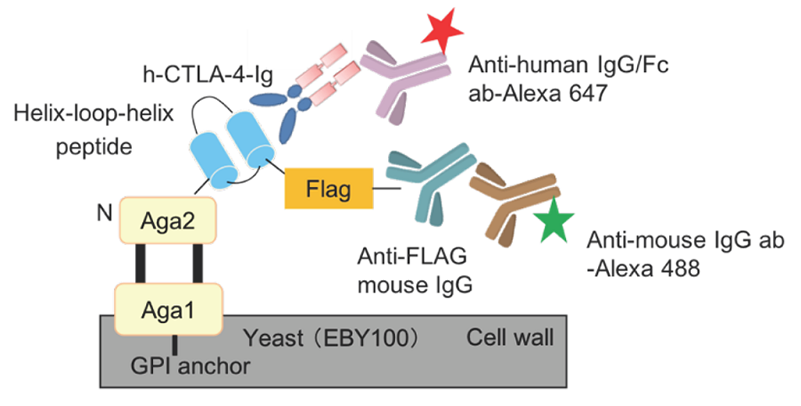
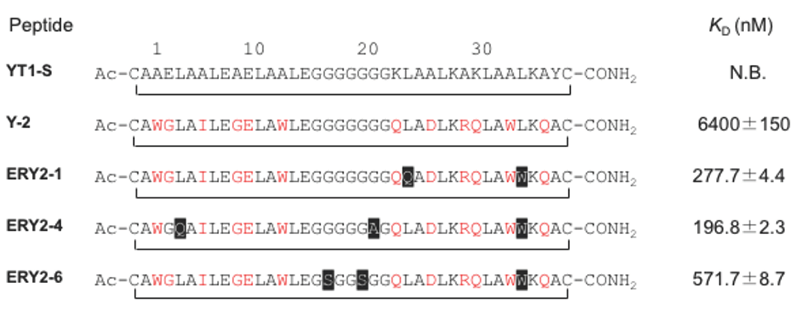
マイクロ抗体ライブラリーΔPTA-12RC-1とΔPTA-12RC-2の混合溶液を用いてライブラリー・スクリーニングを行った。IgG Fcドメインとの融合タンパク質であるヒトCTLA-4 (h-CTLA-4-Ig) を標的分子としてスクリーニングを行った。まず、Magnetic-activated cell sorting (MACS) によって、ビーズに固定したビオチン化h-CTLA-4-Igに結合する酵母クローンを回収して増幅した。さらに、FACSセルソーターを用いて、h-CTLA-4-Ig結合性の酵母クローンをソーティングした (図4)。得られた酵母クローンがコードするHLHペプチドの塩基配列を確認し、獲得したペプチドのアミノ酸配列を推定した。15種のクローンについてDNA配列解析をしたところ、4種の異なったアミノ酸配列に収束したことが判明した。得られたペプチドを、固相法で化学的に合成した。SPR法による結合活性測定を行ったところ、ペプチドY-2 (KD値 6.4µM) を獲得した。Y-2は、CTLA-4に選択的に結合し、また、CTLA-4/B7相互作用を阻害することが推定されたため、こちらを土台分子として親和性成熟 (Affinity Maturation) を行うこととした。
クローンY-2の配列をコードするプラスミドを鋳型としてエラープローンPCRを行い,Y-2を土台分子とした酵母表層提示cHLHペプチド・ライブラリーを作製した (ペプチドあたり1-3個のアミノ酸変異確率)。先と同様条件でh-CTLA-4-Igに対するライブラリー・スクリーニングを実施したところ、3種のペプチドERY2-1、-4、-6を得た (図5)。これらのペプチドを固相法により化学的に合成した。CDスペクトル測定の結果、これらの合成ペプチドが、α-ヘリックス構造をもつことを確認した。さらにSPR法を用いてh-CTLA-4-Igに対する結合活性を測定したところ,図5に示す結果となった。ペプチドERY2-4の結合活性は (KD値 196.8 nM)、土台分子Y-2と比較して約75倍結合活性が上昇しており、親和性成熟に成功した。また標的分子h-CTLA-4-Igに対する特異性も確認した。
5. マイクロ抗体ERY2-4: CTLA-4/B7阻害相互作用阻害活性と、免疫細胞の活性化
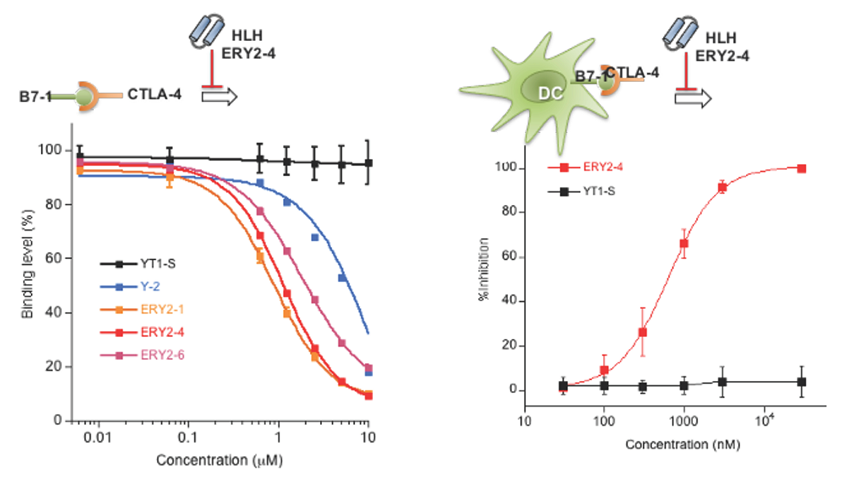
h-CTLA-4-Ig結合性cHLHペプチドERY2-1、ERY2-4、ERY2-6について、CTLA-4/B7-1相互作用阻害活性を評価した (図6)。まず、SPR法を用いて、固化B7-1とh-CTLA-4-Igとの相互作用阻害活性を測定したところ、Y-2、ERY2-1、ERY2-4、ERY2-6のIC50値はそれぞれ、11.2µM、0.92µM、1.1µM、1.7µMであった。さらに、樹状細胞上に提示されたB7と、組換えタンパク質h-CTLA-4-Igとの相互作用阻害活性を測定した。樹状細胞上の細胞表層に結合したh-CTLA-4-Igを、フローサイトメーターを用いて、抗ヒトIgG Fc-FITCを使って検出した。測定の結果、樹状細胞上のB7とh-CTLA-4-Igとの相互作用阻害活性 (IC50) は、632 nMであった。また、ERY2-4のCTLA-4に対する特異性が高いことを確認できたことから、CTLA-4/B7相互作用選択的な阻害によって、ERY2-4が免疫細胞の活性化することが期待された。そこで最後に、ヒト血液由来の混合リンパ球培養における同種間 (アロジェネイック) リンパ球増殖活性測定を行った。大変興味深いとことに、ERY2-4を加えることで、免疫細胞活性化特有の現象であるクラスターが形成されたことを形態学的に確認した。さらに、チミジン取り込み法によって免疫細胞が増殖したことを確認した。すなわち、マイクロ抗体ERY2-4は、CTLA-4/B7-1相互作用を阻害して免疫細胞を活性化させることを明らかにした。

6.おわりに
ペプチドERY2-4を今後さらに親和性成熟させることで、CTLA-4結合活性ならびに、CTLA-4–B7相互作用阻害活性の高いマイクロ抗体が得られると期待できる。これによって、今後がん細胞増殖を止める抗がん活性をもつマイクロ抗体の創出が期待できる。マイクロ抗体のコンセプトに合致する、強固な二次構造をもち生体内でも安定な立体構造規制ペプチドであれば、HLHペプチドに限らずいずれの構造であっても、ポスト抗体医薬品としての開発が期待できる。本研究事例をきっかけとして、多種多様な立体構造を土台とする分子標的ペプチドが創出され、立体構造規制ペプチドを分子基盤とする、中分子医薬品の開発がより一層発展すれば幸いである。
謝辞
本研究は、大阪府立大学大学院生命環境科学研究科杉浦喜久弥教授と和歌山県立医科大学の井上德光教授との共同研究の成果です。両教授に深く感謝申し上げます。また、本研究にあたり、国立研究開発法人日本医療研究開発機構 (AMED) 創薬等ライフサイエンス研究支援基盤事業 創薬等先端技術支援基盤プラットフォーム (BINDS) の課題番号19am0101097j0003の支援を受けました。
文献
1) Stumpf, M. P. H., Thorne, T., De Silva, E., Stewart, R., An, H. J., Lappe, M., Wiuf, C.: Proc. Natl. Acad. Sci. USA, 105, 6959 (2008).
2) Higueruelo, A. P., Jubb, H., Blundell, T. L.: Curr. Opin. Pharm., 13, 791 (2013).
3) Sheng, C., Dong, G., Miao, Z., Zhang, W., Wang, W.: Chem. Soc. Rev., 44, 8238 (2015).
4) Moore, P., Clayton J.: Nature, 426, 725 (2003).
5) 長森英二, 加藤竜司, 清水一憲, 柳原佳奈: 生物工学, 91, 494 (2013).
6) Binz, H. K., Amstutz, P., Plückthun, A.: Nat. Biotechnol., 23, 1257 (2005).
7) Vlieghe, P., Lisowski, V., Martinez, J., Khrestchatisky, M.: Drug Discov. Today, 15, 40 (2010).
8) Fujiwara, D., Fujii, I.: Curr. Protoc. Chem. Biol., 5, 171 (2013).
9) 道上雅孝,藤井郁雄: 特開2019-172647 (P2019-172647A).
10) Kawabata, K., Nagai, H., Konishi, N., Fujiwara, D., Sasaki, R., Ichikawa, T., Fujii, I.: Bioorg. Med. Chem., 22, 1845 (2014).
11) Fujiwara, D., Ye, Z., Gouda, M., Yokota, K., Tsumuraya, T., Fujii, I.: Bioorg. Med. Chem. Lett., 20, 1776 (2010).
12) Fujiwara, D., Kitada, H., Oguri, M., Nishihara, T., Michigami, M., Shiraishi, K., Yuba, E., Nakase, I., Im, H., Cho, S., Joung, J. Y., Kodama, S., Kono, K., Ham, S., Fujii, I.: Angew. Chem. Int. Ed., 55, 10612 (2016).
13) Ramanayake Mudiyanselage T. M. R., Michigami, M., Ye, Z., Uyeda, A., Inoue, N., Sugiura, K., Fujii, I., Fujiwara, D.: ACS Chem. Biol., 15, 360 (2020).
14) Hendriks, L., Besse, B.: Nature, 558, 376 (2018).
15) Ramagopal, U. A., Liu, W., Garrett-Thomson, S. C., Bonanno, J. B., Yan, Q., Srinivasan, M., Wong, S. C., Bell, A., Mankikar, S., Rangan, V. S., Deshpande, S., Korman, A. J., Almo, S. C.: Proc. Natl. Acad. Sci. USA, 114, E4223 (2017).