【トピックス】
発酵生産における機械学習の活用:培地成分に着目して
小西正朗
北見工大・工
1.はじめに
酵素や微生物、アミノ酸、二次代謝物を工業生産する際、単一成分で構成された組成が既知の合成培地もしくは糖蜜・酵母エキス・ペプトン等様々な天然成分を用いる天然培地が用いられる。合成培地はその名の通り、既知の単一成分の試薬を配合して作成される。そのため、再現性の高い培養が可能であるが、一般的に高価である。天然培地成分は、豊富な栄養成分が含まれ、安価であることから培地の添加成分として多用される。研究用の試薬として売られているエキス成分は高度に品質管理されており高価であるが、比較的安定した培養結果が得られるため、再現性が重視される試験研究に用いられる。しかし、高価であることから、工業微生物培養ではより安価な工業用のエキスが使用される。例えば、酵母エキスの場合、工業用のものは研究用試薬の標準とされるBD BactoTM yeast extractの1/3以下の価格である。しかしながら、試薬グレードのものほど高度には品質管理されておらず、生物原料が由来であり、原料生物の種類、株、季節によって成分が変動する他、製造プロセスの違いによっても成分が変わることが知られている。また、一部の試薬グレードの製品は成分組成が公開されているが、ほとんどの製品の組成は公にはなっていない。したがって、工業的な微生物培養の場合、コストや目的生産物の生産性のバランスを考量して、最も費用対効果の高いエキスを培養試験等により選抜し利用する。また、サプライチェーンの問題等、種々の理由で供給が滞る場合もあり、代替エキス成分を選定する必要に迫られる場合もある。代替エキス成分は比較培養試験にて評価されることが多い。これまではエキス類の成分組成分析を行わずトライ&エラーで適切な培地を選別するのが一般的である。より多くのバイオプロセスを工業化し、安定的な生産を実現するためには、工業用天然培地の設計方法をスマート化が必要である。それによって新規バイオプロセスの実用化障壁の低減につながると思われる。筆者らは、天然培地の成分組成をガスクロマトグラフ質量分析計 (GC-MS) 等による網羅的分析で把握し、機械学習モデルによって培養データとの相関関係をモデル化することにより、培養結果の予測および予測に重要な成分 (=培養に重要な成分) を推定する方法を提案している。
本稿では、天然培地成分に関する解説および、筆者らの研究成果を中心に、天然培地を用いた微生物培養の課題と研究例について、紹介する。
2.天然培地成分
2-1 炭素源 (糖蜜・糖液)
製糖工程で生産される糖成分を多く含んだ液体であり、製造中間体のものと晶析後の廃液 (廃糖蜜) がある。サトウキビ由来の糖蜜は糖含量が多いため、バイオプロセス原料としても用いられる1)。Raw juiceと呼ばれるビートの搾汁液を直接利用する場合もあるが、保存性が低く、食品原料と競合する問題がある2,3)。Thick juiceと濃縮中間体4)もバイオエタノール生産などに用いられる。デンプン加水分解物やリグノセルロース系バイオマスの糖化液を用いた微生物生産に関する研究も多く報告されている1)。その他、トウモロコシでんぷん工場の副産物であるコーンスティープリカーやクローバーやアルファルファ等の家畜飼料の乾燥工程で副生成するgreen juiceやbrown juiceなど様々な農業副産物を用いた微生物生産が試みられている5)。
2-2 酵母エキス
パン酵母 (Saccharomyces cerevisiae,Debaryomyceshansenii)・ビール酵母 (Saccharomyces pastorianus)・トルラ酵母 (Cyberlindnera jadinii) などから抽出されるエキス成分であり、肉エキスの代替品や調味料としての食品用途で利用されることも多い。酵母を自己消化もしくは酸や熱分解して得られる。アミノ酸、ビタミン、タンパク質 (ペプチド) を多く含み微生物培養や動物細胞培養の添加剤として利用される。由来となる酵母種の種類、製造工程の違いからその成分組成に違いがあることが知られている6)。筆者らが研究試薬として用いられる酵母エキスと工業用酵母エキスの成分 (計5種,a, b, c, d, e) をトリメチルシリル化 (TMS化) 処理後、ガスクロマトグラフ質量分析法で網羅解析した際のトータルイオンクロマトグラフを図1に示す。ピークパターンの違いから各酵母エキスの低分子成分の組成が異なることがわかる。また、ペプチドは乳酸菌の増殖に影響を及ぼすことが良く知られており、培養液中Streptococcus thermophilusが消費するペプチドと膜受容体が認識するペプチドのパターンが一致することが報告されている7)。次項に示すペプトンとともに、酵母エキスはペプチドの供給源としても機能していると考えられる。
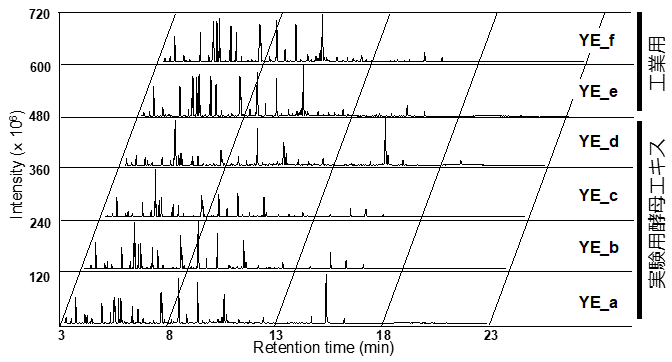
図1 酵母エキスのGC-MSチャート
2-3 ペプトン
ペプトンはミルクカゼイン・獣肉・大豆などタンパク質が多く含まれる原料をタンパク質加水分解酵素もしくは酸などにより分解したものの総称であり、微生物培地には窒素源として利用されることが多い。タンパク質が部分分解したペプチドやアミノ酸を多く含むが、炭化水素や無機塩、ビタミン類も含まれる。原料や使用される加水分解酵素により様々な製剤が利用されている8)。ペプトン類と判断できる製品は試薬用、工業用のものを合わせると国内だけでも数十種類の製品がラインナップされている。
2-4 その他
肉エキス・麦芽エキス・カゼイン等を培地に添加する場合もある。一部の嫌気菌の培養では、動物の血液もしくはその加水分解物を添加する場合がある。
3.天然成分の影響評価手法
工業的なプロセスに関わる培養技術者は微生物を用いた製造を行う際、上述の様々な原料から、コストおよび性能面で優れたものを選別し組み合わせる必要がある。筆者らは天然培地成分の組成をメタボロミクスで用いられている網羅的機器分析により、その成分をプロファイリングし、そのデータセットと培養結果を機械学習で紐づけることができれば、培養結果を予測できる他、培養結果に影響を及ぼす重要成分を推定できると考えた。
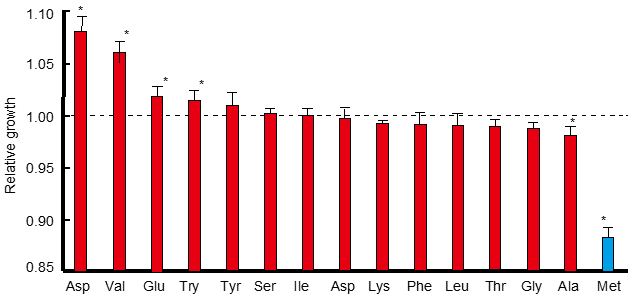
まず、複数の酵母エキスが大腸菌のNBRC 3301株 (K12株) の増殖に与える影響を調べた6)。大腸菌最少培地M9培地に試薬グレードの酵母エキス4種類と工業グレードの酵母エキス2種類の添加量を4水準 (0.5, 1.0,2.0, 5.0 g/l)、計24パターンで添加して、大腸菌を培養した。同時に、酵母エキスをTMS化前処理し、GC-MSにより成分を網羅的に分析した。その結果、208個のピークが検出された。検出されたピークを標準化した後、208×24のデータマトリックスを作成した。メタボローム解析と同様に部分最小二乗法 (partial leastsquares regression, PLS) により、増殖量 (18時間後の濁度) との相関関係をモデル化した。学習データおよび評価データの組合せを変えて、モデルを評価したところ、いずれの組合せでも、学習データについて、高い寄与率 (決定係数) R2>0.98が得られた。一方で、評価データについては、寄与率 (決定係数) Q2は0.328から0.850であった。一般的にQ2>0.65で十分な予測精度とされる。評価データの寄与率が低い場合は評価データに類似した学習データがない傾向が認められた。Q2が最も高かった組合せで構築したモデルを用いて、重要成分を予測した。相関係数から算出されるvariableimportance in projection (VIP値) が1以上となる成分を抽出したところ、3-aminoisobutyric acid (3-ABA) ,butanedioic acid ( succinate) , urea, Met, ethanol amine,3-hydroxypropionic acid (3HP) が増殖に負の相関を示し、Phe, Gly, Ile, Leu, Val, Asn, Ser, Glu, Asp, Lys, Tyr,5-oxoproline, ornithineが増殖と正の相関を示した。負の相関を示す物質を増殖阻害物質、正の相関を示す物質を増殖促進物質と推定した。実際にM9培地に14種類のアミノ酸を添加して培養したところ、Asp, Val, Glu,Trpは増殖促進効果を示し、Ala, Metは阻害効果を示した。一部の予想と検証実験結果は一致し、重要な栄養成分を探索する手法としても有効であることが示された (図2)。

筆者らは、さらに、メタボローム解析手法を木質バイオマスの糖化液を原料としたバイオエタノール生産に適用した9)。木質バイオマスからバイオエタノールを生産する場合、酸やアルカリによる加水分解およびセルラーゼによる糖化工程により、バイオマスを糖化し、酵母により糖化液を発酵させエタノールを生産する。この際、糖の過分解やリグニン分解物など、フラン化合物やフェノール性化合物等、酵母に毒性を示す様々な揮発性成分 (発酵阻害物質) が副生成することが知られている。GC-MSを用いることでこれらの揮発性成分を一斉分析できることに着目し、揮発性成分プロファイルとバイオエタノール生産の関係をモデル化し、成分の影響を評価した。産地の異なるコーンコブ (トウモロコシ芯) やコーンストーバー (茎や葉の部分) を種々濃度の硫酸で処理することで、阻害物質の組成が異なる糖化液を16種類調製し、糖化液を用いたバイオエタノール生産試験を行った。糖化液により、酵母の増殖やエタノール生産量に違いが認められた。そこで揮発性成分プロファイルと酵母の増殖およびエタノール生産の関係をPLSでモデル化した。酵母エキスの場合と同様に培地成分から培養結果を予測できることが示された。糖化液中の阻害物質としてよく知られていたfurfuralや5-hydroxymethyl furfuralの他、methoxyacetophenone,apocynin, syringaldehydeなどが重要な阻害成分であることがわかった (図3)。
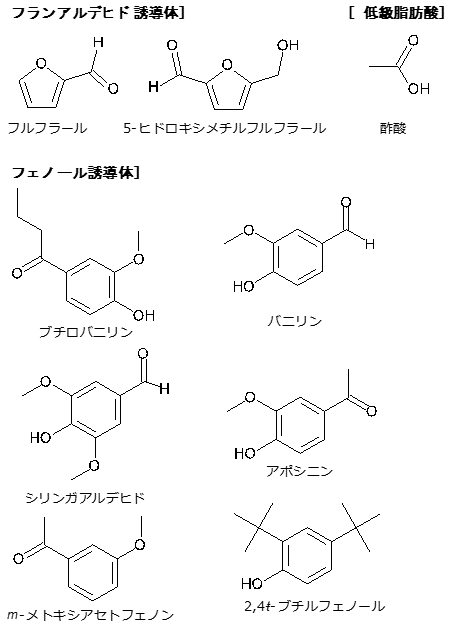
図3 バイオマス糖化液の組成から予測された発酵阻害成分9)
さらに、PLSの代わりに深層学習 (deep neural network,DNN) モデル を適用すると、さらに高い精度で予測可能であること、培養経時変化を直接予測できることも報告している10)。また、オートエンコーダーと呼ばれるアルゴリズムを用いることで、特徴的な成分を抽出することができた。
以上の結果から、天然培地原料の組成を機器分析でプロファイリングし、培養結果との相関関係を教師つき機械学習で数理モデル化できることを示した。成分プロファイルから培養結果を予測できる他、モデルの判定に重要な成分を抽出することができるため、培養に影響が大きい成分を見出すことができる。
培地の評価にはGC-MSのみならず様々な分光分析法が利用できることが培養細胞の研究でも報告されている。フーリエ変換近赤外分光法 (FT-NIR) はフーリエ変換中赤外分光法 (FT-MIR)、二次元蛍光分光法 (2DFL) やラマン分光法と比べて、チャイニーズハムスター卵巣 (CHO) 細胞を用いたヒト抗体生産をよく予測できることが報告されている11)。Brunnerらは、CHO細胞培養に用いる培養液の調整過程でMIRや2D FLを適用でき、培地の変動解析には2D FLが優れていると報告している12)。培養プロセスの管理や監視を目的とする場合、より簡便にデータが得らる分光法を活用も考えられる。著者らも微生物培地の変動評価にFT-MIRや2D FLが有効であることを確認している。
最少培地中での栄養成分による細胞挙動はアミノ酸や核酸の合成速度に依存するが、リッチな栄養培地においては、翻訳遺伝子の発現上昇が支配的でタンパク質合成速度に依存して細胞増殖すると考えられている13)。著者らは増殖や有用物質の生産に支配的な栄養成分に着目して解析を進めているが、栄養培地中での微生物の挙動は単一の成分によって支配されていないようで、複数の成分の影響またはそれらの相乗効果があるようだ。
4.おわりに
メタボロミクスアプローチにより、複雑な組成を持つ天然成分による培養挙動の解析が可能なことを示した。しかし、天然培地原料は多様な成分を含んでおり、今回紹介したGC-MSによる低分子成分の分析手法で成分を網羅して検出できていないと思われる。ペプトンなどの成分であれば低分子成分のみならず、ペプチドを多く含んでおり、乳酸菌などの栄養要求性の高い微生物を培養する場合、ペプチドの網羅的解析データを必要になる可能性がある。この場合、液体クロマトグラフ質量分析計 (LC-MS) やマトリックスレーザー支援飛行時間質量分析計 (MALDI-TOF/MS) など複数の分析手法を組み合わせて評価することも有効であろう。機械学習手法も様々なものが提案されており、PLSやDNNの他、ランダムフォレスト14)、サポートベクトルマシン15)など様々な機械学習手法を組み合わせることができる。影響を評価したい培地成分、微生物種などにより、適切な分析手法と機械学習手法を組み合わせることにより、より真実に近い予測が得られであろう。また、教師付き機械学習手法を利用する際は過学習になっていないか慎重に確認する必要がある。学習データの収集方法は予測結果に大きな影響を及ぼすので注意する必要がある。学習データの収集には実験計画法16)における無作為化や局所管理化、反復などの考え方を利用することが望ましいが、天然成分を用いているため、成分濃度の完全な無作為化が困難なことも解析法の課題である。
文献
1) Kuenz, A., Krull, S.: Appl. Microbiol. Biotechnol., 102, 3901 (2018).
2) Pavlecic, M., Vranal, I., Vibovec, K., Santek, M. I., Horvat, P., Santek, B.: Food Technol. Biotech., 48, 362 (2010).
3) Hinková, A., Bubník, Z.: Czech J. Food Sci., 19, 224 (2001).
4) Dodic, S., Popov, S., Dodic, J., Rankovic, J., Zavargo, Z., Mucibabic, R. J.: Biomass Bioenergy, 33, 822 (2009).
5) Thomsen, M. H.: Appl. Microbiol. Biotechnol., 68, 598 (2005).
6) Tachiana, S., Watanabe, K., Konishi, M.: J. Biosci. Bioeng., 128, 468 (2019).
7) Proust, L., Souabié, A., Pedersen, M., Besançon, I., Haudebourg, E., Monnet, V., Juillard, V.: Front Microbiol., 10 : 906 (2019) doi: 10.3389/fmicb.2019.00906.
8) 駒 大輔,中山勇人,森芳邦彦,大本貴士: 生物工学会誌,4, 195 (2011).
9) Watanabe, K., Tachibana, S., Konishi, M.: Bioresour. Technol., 281, 260 (2019).
10) Konishi, M.: J. Biosci. Bioeng., 129, 723 (2020).
11) Trunfio, N., Lee, H., Starkey, J., Agrabi, C., Liu, J., Yoon, S.: Biotechnol. Prog., 33, 1127 (2017).
12) Brunner, M., Brosig, P., Losing, M., Kunzelmann, M., Calvet, A., Stiefel, F., Bechmann, J., Unsoeld, A., Schaub, J.: Eng. Life Sci., 16, 666 (2019).
13) Baez, A., Kumar, A., Sharma, A. K., Anderson, E. D., Shiloach, J.: N. Biotechnol., 25, 120 (2019).
14) Breiman, L.: Machine Learning, 45, 5 (2001).
15) Cortes, C., Vapnik, V.: Machine Learning, 20, 273 (1995).
16) 田口玄一, <復刻版>第3版 実験計画法 上, 丸善株式会社 (2010).