【トピックス】
高度な安定同位体標識による高分子量蛋白質の溶液NMR 解析法
宮ノ入洋平、武田光広、寺内 勉、甲斐荘正恒
大阪大・蛋白研/名古屋大・理、熊本大・生命/名古屋大・理、SAIL テクノロジーズ株式会社/都立大・理、都立大・理/名古屋大・理
1.はじめに
蛋白質が有する高次な生命機能を理解するうえで、その立体構造が非常に重要な情報を与える。原子分解能に至る立体構造情報は、蛋白質の機能解明のみならず、様々な疾患に対する治療薬の創製においても大きな役割を担ってきた。現在、猛威を奮う新型コロナウイルス感染症に対しても、関連蛋白質の立体構造解析が世界的に進められており、ワクチンや革新的な治療薬の開発に期待が寄せられている。構造生物学研究が様々な分野で貢献していくなかで、その研究手法も大きく発展している。クライオ電子顕微鏡による単粒子解析の高速・高精度化、X線結晶構造解析やNMR構造解析法の自動化といった構造決定法の改良は、目覚ましい進化をみせている。それに付随して、近年では蛋白質の動的な構造情報を解析する技術開発が注目を浴びている。
多くの蛋白質は、生体内において様々な時間域で揺らいでおり、その運動性の変化が、高次な生命機能を調節していることが示唆されてきた。溶液NMR法は、ピコ秒から数日にわたる広範な時間軸に渡って、時間変数を含む構造動態情報を得ることができるため、蛋白質動態と機能発現との相関関係を明らかにするうえで、非常に重要な役割を担っている。これまでに、高圧NMR測定法や常磁性緩和促進法を利用した蛋白質の準安定状態の解析や生細胞内における蛋白質の状態を観測するIn cellNMR法が新たに開発されてきた。
一方、溶液NMR法の最大の欠点である分子量限界の問題は、今尚、高い壁として立ちふさがっている。一般的に、溶液NMR法では分子量30kDaを超える高分子量蛋白質や蛋白質―蛋白質複合体を対象とした動態解析は困難を極めている。生体内においては、抗体や膜蛋白質など分子量100 kDaを超えるものも多く存在しており、さらには蛋白質同士が会合した超分子複合体を形成することで高次な機能を発現する例が多い。蛋白質の構造動態と機能との相関関係を示し、高次な生命機能を理解するためには、この溶液NMR法の “分子量の壁” を解決することが重要な課題であり、世界中で技術開発が繰り広げられている。そのなかで、我々は立体整列安定同位体標識 (Stereo array isotope labeling : SAIL) 法の開発を長年進めてきた。SAIL法では、蛋白質を構成する20種のアミノ酸について、立体・位置特異的に安定同位体標識を施したSAILアミノ酸を合成し、それを用いて目的蛋白質を調製する (SAIL蛋白質)。SAIL蛋白質を利用することで、蛋白質の分子量増大に伴うNMR信号の縮重や低感度化を抑えることが可能となり、40 kDaを超える蛋白質についても精密な溶液立体構造を決定できることを示してきた1)。この独自のSAIL技術を改良することで、より高分子量の試料についても、高感度かつ高精度なNMR信号を捉えることが可能となってきた。本項では、80 kDaを超える蛋白質について、メチル基および芳香環由来のNMR信号を対象とした解析法について紹介する。
2.選択的メチル基安定同位体標識法
溶液NMR法において、観測対象の分子量が増加すると、核緩和の影響が増大するため、NMR信号の感度が大幅に低減する。また、NMR信号同士の縮重も多くなるため、一義的な信号帰属は困難となる。これら問題を解決するため、対象蛋白質の安定同位体標識手法やTROSY、CRINEPTなどの測定技術が開発されてきた2,3)。とくに、観測対象を蛋白質中のアミド基やメチル基に限定した、部位特異的な安定同位体標識手法が広く利用されている4)。この手法を用いることで、分子量100 kDaを超えるような高分子量蛋白質においても、アミド基やメチル基のシグナルを高感度に観測することができる。一般的に、アミド基やイソロイシン (Ile), バリン (Val), ロイシン (Leu) およびアラニン (Ala) 残基等のメチル基は蛋白質全体に広く分布しており、蛋白質中の全原子数の30%ほどを占める。したがって、リガンドとの相互作用領域や、それに伴う構造変化などを解析することが可能となり、膜蛋白質等の活性化機構の解明に、広く利用されている。
解析対象の蛋白質に、メチル基特異的に安定同位体を標識する手法として、アミノ酸前駆体を利用した調製法がよく知られている5,6)。例えば、Leu、Val残基のメチル基のみを13CH3に標識する場合、安定同位体標識されたα-ケト酸を利用する (図1a)。α-ケト酸は、大腸菌のLeuおよびVal生合成経路の中間体であるため (図3a)、図1aの標識パターンを有するα-ケト酸を大腸菌の培養培地に添加するだけで、Leu残基のδ1, δ2位およびVal残基のγ1,γ2位のメチル基に13CH3を導入することができる。図1bには、図1aに示したα-ケト酸を利用して調製した分子量82 kDaのリンゴ酸合成酵素 (Malatesynthase G: MSG) の1H-13C HMQCスペクトルを示している。LeuおよびVal残基のメチル基の信号を高感度に捉えることができている。比較的簡便に高精度なスペクトルが得られる反面、欠点も散見される。MSGの場合、Leu残基が70個、Val残基が46個あるため、1H-13CHMQCスペクトル上には、232個のメチル基信号が観測される。このため、シグナル同士の縮重が顕著な領域が多い。メチル基の信号帰属は、アミノ酸残基内のアミド基―メチル基間の化学結合を介した “out-and-back” 実験やアミノ酸置換体を利用して帰属するケースが多い7)。対象となる蛋白質および蛋白質複合体の分子量が増大すると、out-and-back実験の感度も低減することや、信号同士の縮重がさらに深刻となり、アミノ酸配列特異的かつ立体特異的 (Leu δ1とδ2およびVal γ1とγ2の区別) な信号帰属は困難を極める。
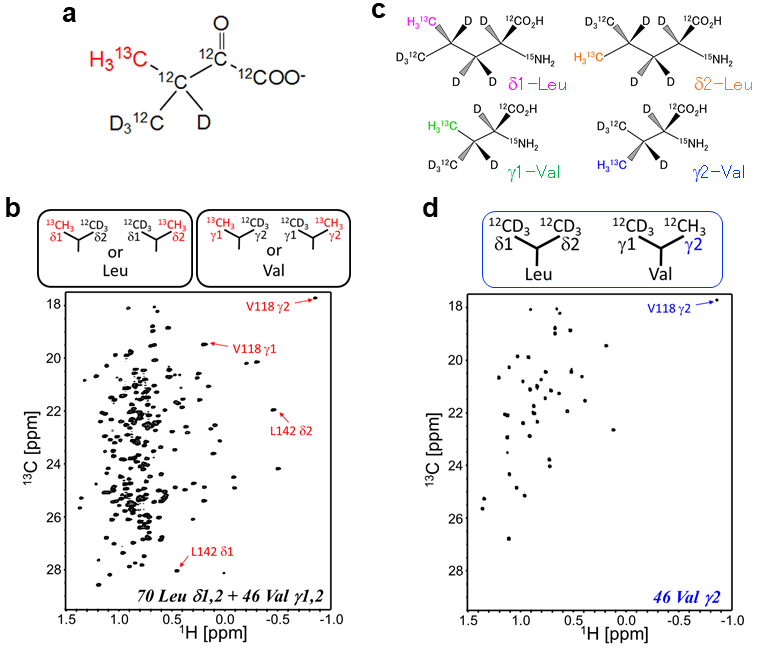

また、図1aのα-ケト酸はメチル基の立体特異性を保持していない。そのため、得られる蛋白質中のLeu残基及びVal残基の各メチル基への13CH3導入率は最大でも50%が上限となる (図1b上段)。この場合、微量な蛋白質に対しては1H-13C HMQCスペクトルでは十分な感度を得られないばかりか、メチル基間の距離情報を得るためのNOESYスペクトルではさらなる感度低下を引き起こし、構造動態解析に困難を生じる。標識率の低減を克服するため、異なるアミノ酸前駆体を利用する手法も開発されているが8)、立体特異的およびアミノ酸残基特異的な安定同位体標識は不可能である。
我々は、このようなメチル基標識技術の問題点を克服するため、新たな安定同位体標識手法を開発した。従来の、安定同位体標識されたLeu, Valの前駆体を利用する代わりに、独自のSAIL技術を駆使して、立体特異的に13CH3を導入したLeu, Valを合成した (図1c)9)。α-ケト酸の代わりに、これら立体特異的メチル標識アミノ酸を利用することで、アミノ酸残基特異的かつ立体特異的にメチル基を標識することができる。つまり、Leu残基のδ1メチル、δ2メチル、Val残基のγ1メチル、γ2メチルを自由自在に組み合わせて、観測することが可能となる。例として、MSGについてVal残基のγ2メチルのみを13CH3標識したときの1H-13C HMQCスペクトルを示す(図1d)。α-ケト酸を利用した例 (図1b) と比較して、大幅にNMR信号同士の縮重が抑えられていることがわかる。分子量100 kDaを超えるような超高分子量蛋白質や蛋白質複合体においても、一つ一つのNMR信号を明確に帰属することができ、リガンドとの相互作用解析や構造変化に伴うNMR信号の変化を正確に捉えることが可能となる。また、立体特異的メチル標識アミノ酸を利用することで、安定同位体の導入率も大幅に改善できた。蛋白質生合成に広く利用されている大腸菌BL21 (DE3) 株を利用する場合、培養培地1Lあたり20 mgのLeuおよび100 mgのValを添加することで、目的蛋白質のLeu,Val残基にアミノ酸残基特異的かつ立体特異的に、13CH3を90%以上の導入率で標識することができる9)。α-ケト酸を利用する場合と同等のコストで、メチル基のNMR信号を高感度に観測できるため、微量な高分子量蛋白質を対象としてNMR実験が可能であり、メチル基同士の距離情報も効率よく観測することができる。
3. 芳香族アミノ酸残基のNMR信号高感度観測法の開発
前述のように、アミド基やメチル基を観測対象とした手法は、分子量100 kDaを超えるような高分子量蛋白質についても広く活用され、相互作用様式の理解に大きく貢献している。一方で、得られる情報はアミド基とメチル基に限定されるため、局所的な構造動態を捉えることに留まる。チャネル蛋白質複合体や、マルチドメイン蛋白質などは、リガンドとの相互作用を引き金に、相互作用領域のみならず分子全体に構造変化が引き起こされ、メッセンジャー物質の輸送・排出を促したり、酵素活性を調節したりすることが知られている。このようなアロステリックな分子認識、機能発現機構を詳細に理解するうえでは、蛋白質とリガンドとの相互作用領域のみならず、分子全体の構造動態を捉えることが必要となる。したがって、高分子量蛋白質においても、アミド基やメチル基に加えて、新たな観測対象を増やすことが重要と考えられる。そこで我々は、芳香族アミノ酸残基に注目した。
蛋白質中のフェニルアラニン (Phe) やチロシン (Tyr)、トリプトファン (Trp) といった芳香族アミノ酸残基は、蛋白質内部で疎水性相互作用を介したコア構造を形成し立体構造の形成・維持において重要な役割を担っている。また、蛋白質表面に疎水パッチを形成し、他の蛋白質や様々なリガンドとの相互作用にも、直接関与することが多い。これら残基は、側鎖に嵩高いリング構造を有しており、周囲のアミノ酸残基とも多くの相互作用を形成する。実際に、芳香族アミノ酸残基側鎖のNMR信号からは、数多くの原子間距離情報を抽出することができ、立体構造決定に重要な情報を与えている10)。さらに、この嵩高いリング構造は、回転運動やフリップ―フロップ運動を有している。これらの動態は、自身のみならず、相互作用する周囲のアミノ酸残基とも連動するので、蛋白質中の特定のアミノ酸残基といった局所情報のみならず、機能ドメイン単位の広範囲な構造動態を特徴づけると考えられる11)。したがって、高分子量蛋白質について、アミド基やメチル基に加えて芳香環由来のNMR信号を高感度に捉えることができれば、より精密な構造動態を、分子全体にわたって捉えることが期待できる。
一方で、芳香環の13C-1H NMR信号の高感度観測や立体特異的かつアミノ酸配列特異的な信号帰属は、分子量10 kDa程度の蛋白質でも容易ではない。この原因として、芳香環構造内における磁気的相互作用の影響が大きいことが挙げられる。実際に、13C,15N均一安定同位体標識を施したTrp (図2a) を用いて調製したMSGについて、1H-13C TROSYスペクトルを測定した。この試料では、MSG中のTrp残基のみが1H,13C,15N標識され、そのほかのアミノ酸残基は均一に2H,12C,14Nで標識しており、Trp残基周囲からの核緩和の影響を抑えている。それにも関わらず、Trp残基インドール間の13C-1H 相関NMR信号は、ほぼ観測されていない (図2b)。つまりインドール環内の1Hおよび13C核間で働く核緩和機構がNMR信号の低感度化に大きく影響を与えていることがわかる。そこで我々は、Trpインドール環内の安定同位体標識パターンを改良することで、核緩和機構の最適化を行うことにした。
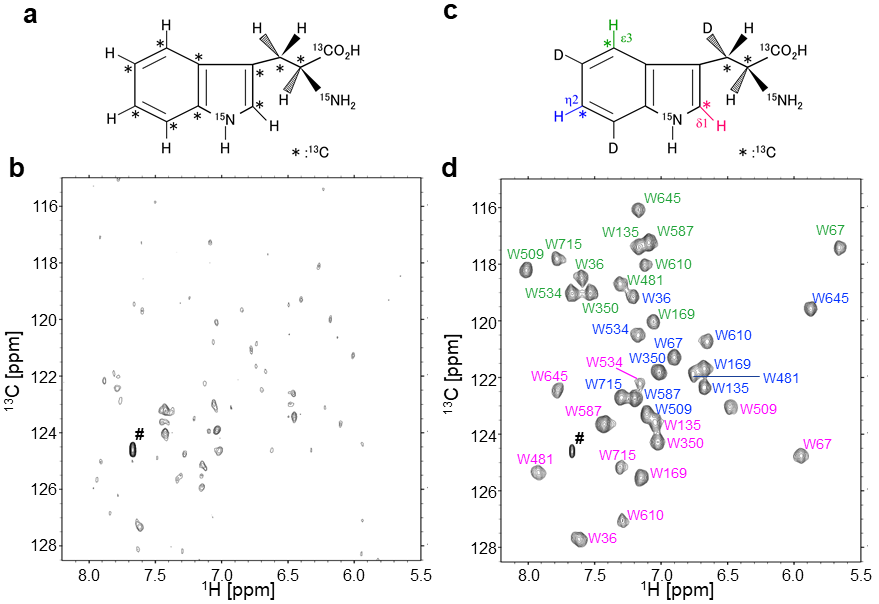

我々は、長年培ってきたSAIL技術を活用することで、Trpインドール環内の核緩和機構を最適化した新しい安定同位体標識Trp (核緩和最適化SAIL-Trp) を開発した (図2c)。この核緩和最適化SAIL-Trpでは、インドール環内のδ1,εおよびη2位の13C-1H相関信号を高感度に検出するために、該当箇所以外の炭素を12C,2Hに特異的に置換して、核緩和の影響を抑える設計を行った。図2dには、核緩和最適化SAIL-Trpで標識したMSGについて、1H-13C TROSYスペクトルを示している。前者の13C,15N均一安定同位体標識Trpの例 (図2b) と比較して、信号感度が劇的に改善していることがわかる。MSG中には12個のTrp残基があるが、そのすべてについてδ1,ε3およびη2の13C-1H相関信号が観測されている。各NMR信号の帰属に関しては詳細を省くが、既存のパルスプログラムを使った実験ならびにアミノ酸置換体を利用することで、立体特異的およびアミノ酸配列特異的な信号帰属に成功している12,13)。このような核緩和最適化技術は、Trpのインドール環のみならず、Phe残基やTyr残基の芳香環にも適用可能であり、本手法を利用することで、高分子量蛋白質について、新たに芳香環のNMR信号を観測対象に加えることに成功した12,13)。
4.安価な安定同位体標識蛋白質の調製法
前述のように、高分子量蛋白質を対象とした溶液NMR解析法においては、試料の安定同位体標識法の工夫が重要となる。ここまで、高度な安定同位体標識を施したアミノ酸前駆体やアミノ酸を紹介してきたが、これらを利用したアミノ酸残基特異的な標識試料を効率よく、大量に調製することが必要となる。蛋白質への安定同位体標識法としては、無細胞蛋白質合成系など、様々な手法が確立されているが、大腸菌蛋白質発現系が広く利用されている。本稿で示したアミノ酸選択標識試料も、すべて汎用な大腸菌BL21 (DE3) 株を利用した大量発現系を用いて調製されている。培養方法も比較的簡便であり、基本的には、培養培地に安定同位体標識されたアミノ酸やアミノ酸前駆体を添加するだけで、目的蛋白質の特定のアミノ酸残基に対し、安定同位体標識することが可能である。
一方で、大腸菌のアミノ酸生合成過程の問題上、アミノ酸の種類によっては、代謝拡散によって、目的外のアミノ酸にも安定同位体が標識されてしまうことがある。また、添加したアミノ酸が、目的蛋白質へ効率よく取り込まれるために、添加量を十分に検討する必要がある。例えば、前述したLeuおよびValの場合、大腸菌の生合成経路においていずれもピルビン酸を出発点として合成されるが、Val生合成の下流でLeuが合成されている (図3a)。したがって、大腸菌培養培地にValを添加するとLeuも同時に安定同位体標識されてしまう。実際に、培地1LあたりにValを100 mg、Leuを20 mg同時に添加しないと、Valの目的蛋白質への取り込み率は90%を超えない9)。Val残基以外にも、Alaなどは生合成経路が多岐にわたるため、数百mgのAlaを加えても高い取り込み率を実現することは難しい14)。大量の安定同位体標識アミノ酸を利用することは、試料調製にコストがかかるため、発現量の少ない高分子量蛋白質への溶液NMR研究の応用に大きな障壁となる。我々は、目的蛋白質へのアミノ酸取り込み率の改善を図るため、ラトガース大の井上正順博士および石田洋二郎博士に協力を得て、アミノ酸要求性大腸菌株を調製した15)。
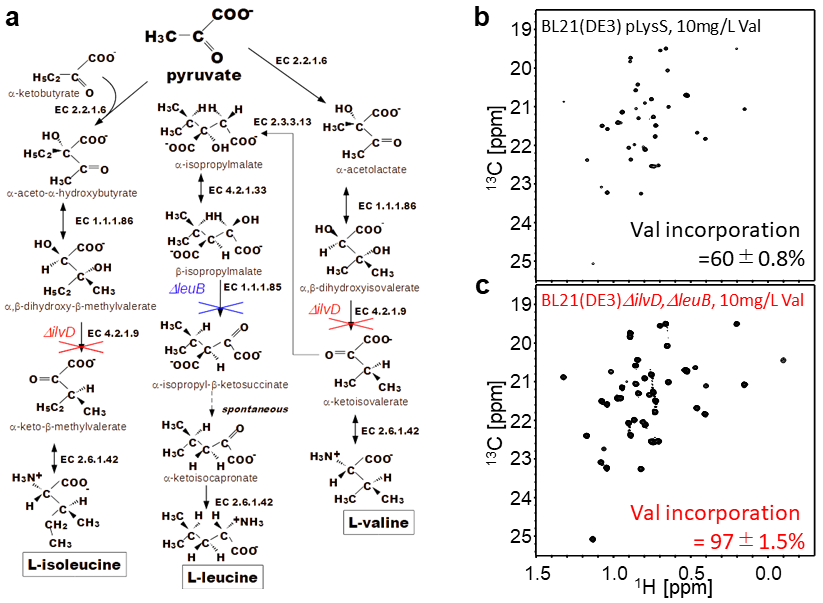

我々は、汎用の大腸菌BL21 (DE3) 株に対して図3a中のEC 4.2.1.9 (dihydroxy acid dehydratase; ilvD) およびEC1.1.1.85 (3-isopropylmalate dehydrogenase; leuB) の遺伝子を破壊した大腸菌BL21 (DE3) ΔilvD,ΔleuBを作成した。これによりピルビン酸からのVal, Leu, Ileの合成経路が遮断され、培養培地に該当のアミノ酸を添加しない限り、大腸菌が生育できない状態となる。この大腸菌では、添加されたアミノ酸を直接利用して蛋白質生合成を行うため、目的蛋白質へのアミノ酸取り込み率が高くなることが期待できる。そこで、MSGへのVal選択標識を例に、通常の大腸菌と比較を行った (図3b,c)。前述のように、通常の大腸菌BL21 (DE3) 株の場合は、安定同位体標識されたValを100 mg/L、非標識のLeuを20 mg/L添加することで、ようやくValの取り込み率が90%に到達する9)。Valの添加量を10 mg/Lに減らすと、取り込み率は60%程度をとどまり、高感度なNMR信号の観測は望めない (図3b)。一方、新たに作製した大腸菌BL21 (DE3) ΔilvD,ΔleuB株で培養条件を検討したところ、Ile, Leu, Valを培地中に各々10 mg/L加えると十分に生育し、通常の大腸菌BL21 (DE3) 株と同等のMSG発現量が確認できた。この条件で、安定同位体標識されたValの取り込み率を確認したところ、97%以上となっていることが明らかとなった (図3c)15)。添加量を大幅に減らしつつも、高感度にNMR信号を観測することが可能となった。
同様な、アミノ酸要求性大腸菌を利用した選択標識法は芳香族アミノ酸に対しても確立されており、やはり10 mg/L 前後のアミノ酸添加で高効率かつ特異的に標識することができる16)。試料調製のコストを大幅に減らすことで、高分子量蛋白質について様々な標識パターンを有する試料を準備することができ、正確な信号帰属、原子間距離情報の決定に大きく貢献することができる12,13)。
5.おわりに
本稿では、高度な安定同位体標識技術を利用することで、分子量80 kDaを超えるような高分子量蛋白質についても、高感度かつ先鋭的にNMR信号を捉える手法を紹介した。核緩和最適化SAILアミノ酸により、メチル基やアミド基のみならず芳香環のNMR信号を捉えることで、より高精度な構造解析や相互作用に伴う構造変化を解明することが期待できる。我々は既に本手法を利用することで、蛋白質中の芳香環回転運動を、定量的に解析する新しい動態解析法も確立している11)。
核緩和最適化安定同位体標識技術は、メチル基や芳香環に限らず、さまざまなアミノ酸の原子団についても適用可能であり、既にメチレン基への導入を実証済みである12)。試料調製法に関しても、大腸菌生合成系に限らず、無細胞蛋白質合成系を利用した手法も確立しているため、今後はより多くのアミノ酸残基を対象とした高感度観測ならびに側鎖原子団が関与する水素結合などを対象とした動態解析法17)への展開、更には1000 kDaに及ぶ超高分子複合体への応用を進めていきたい。
謝辞
本項で紹介した研究に関し、ラトガース大学の井上正順博士と石田洋二郎博士、アメリカ国立衛生研究所のVitali Tugarinov博士にご協力いただきました。この場をお借りして御礼申し上げます。
文献
1) Kainosho, M., Torizawa, T., Iwashita, Y., Terauchi, T., Ono, A. M., Güntert, P.: Nature, 440, 52 (2006).
2) Pervushin, K., Riek, R., Wider, G., Wüthrich, K.: Proc. Natl. Acad. Sci. USA, 94, 12366 (1997).
3) Riek, R., Wider, G., Pervushin, K., Wüthrich, K.: Proc. Natl. Acad. Sci. USA, 96, 4918 (1999).
4) Tugarinov, V., Hwang, P. M., Ollerenshaw, J. E., Kay, L. E.: J. Am. Chem. Soc., 125, 10420 (2003).
5) Gardner, K. H., Rosen, M. K., Kay, L. E.: Biochemistry, 36 (6), 1389 (1997).
6) Goto, N. K., Gardner, K. H., Mueller, G. A., Willis, R. C., Kay, L. E.: J. Biomol. NMR, 13, 369 (1999).
7) Tugarinov, V., Kay, L. E.: J. Am. Chem. Soc., 125, 45, 13868 (2003).
8) Gans, P., Hamelin, O., Sounier, R., Ayala, I., Durá, M. A., Amero, C. D., Noirclerc-Savoye, M., Franzetti, B., Plevin, M. J., Boisbouvier, J.: Angew Chem Int Ed Engl, 49 (11), 1958 (2010).
9) Miyanoiri, Y., Takeda, M., Okuma, K., Ono, A. M., Terauchi, T., Kainosho, M.: J. Biomol. NMR, 57, 237 (2013).
10) Takeda, M., Terauchi, T., Ono, A. M., Kainosho, M.: J. Biomol. NMR, 46, 45 (2010).
11) Yang, C. J., Takeda, M., Terauchi, T., Jee, J. G., Kainosho, M.: Biochemistry, 54, 6983 (2015).
12) Kainosho, M., Miyanoiri, Y., Terauchi, T., Takeda, M.: J. Biomol. NMR, 71, 119 (2018).
13) Miyanoiri, Y., Takeda, M., Terauchi, T., Kainosho, M.: BBA. Gen. Subj., 1864 (2), 129439 (2020).
14) Ayala, I., Sounier, R., Usé, N., Gans, P., Boisbouvier, J.: J. Biomol. NMR, 43, 111 (2009).
15) Miyanoiri, Y., Ishida, Y., Takeda, M., Terauchi, T., Inouye, M., Kainosho, M.: J. Biomol. NMR, 65, 109 (2016).
16) Rajesh, S., Nietlispach, D., Nakayama, H., Takio, K., Laue, E. D., Shibata, T., Ito, Y.: J. Biomol. NMR, 27, 81 (2003).
17) Takeda, M., Miyanoiri, Y., Terauchi, T., Yang C. J., Kainosho M.: J. Magn. Reson., 241, 148 (2013).