【トピックス】
酵母スクリーニングからワイン醸造
上垣浩一、松尾啓史
近大農・応用生命化学科、近大院農/滋賀県工技総セ
1.はじめに
Saccharomyces cerevisiaeは、世界中で酒類や発酵食品に利用されており、もちろん日本でも清酒、ワイン、ビールといった酒類の生産において主要な役割を担っている。アルコール発酵の原因として酵母が発見されて以来、酒類醸造に利用される、多くのS. cerevisiae株が発見されている。酒類醸造に使用されているS. cerevisiae株は繰り返し植え継がれていく中で醸造環境に適応しながら家畜化されてきたものだと考えられている1,2)。そのため、市販の清酒、ワインやビールに使用されている株は遺伝的および醸造特性の多様性を欠いていることが報告されている3-5)。このような多様性の低下は酒質の均一化を招き、近年では海外輸出、消費者のニーズの変化にも対応するために新しい香味を醸す酵母株が求められている。
長年、S. cerevisiaeは主に果物や発酵環境中に存在すると考えられていたが、昨今では果物や発酵環境だけでなくハチ、ハエ、樹皮、土壌、花といった分離源から採取されるようになった。野生株を含むゲノム研究から、野生株のS. cerevisiaeの遺伝構造は家畜化された酵母の遺伝構造よりも複雑な構造を示し6,7)、新たな発酵特性を持つことが期待できる8,9)。このような背景の下、各地方の公設試験研究機関、大学等では、従来とは異なる発酵特性を持つ新規酵母株を求め自然界から酵母株の探索を行い、地域発・大学発ブランドとして酒類開発に利用しようとしている10,11)。しかし、野生酵母の遺伝的多様性と醸造特性に関する報告は少ない。我々の研究室でも大学キャンパスの花々から酵母のスクリーニングを行い、遺伝的多様性や発酵特性を調べ、得られた野生酵母の醸造分野への応用可能性を調べている。本稿では、我々のスクリーニングから得られた酵母株の遺伝的多様性や醸造特性の違い、分離酵母株を利用した果実酒開発への利用を紹介する。
2.野生酵母のスクリーニングと遺伝的多様性解析
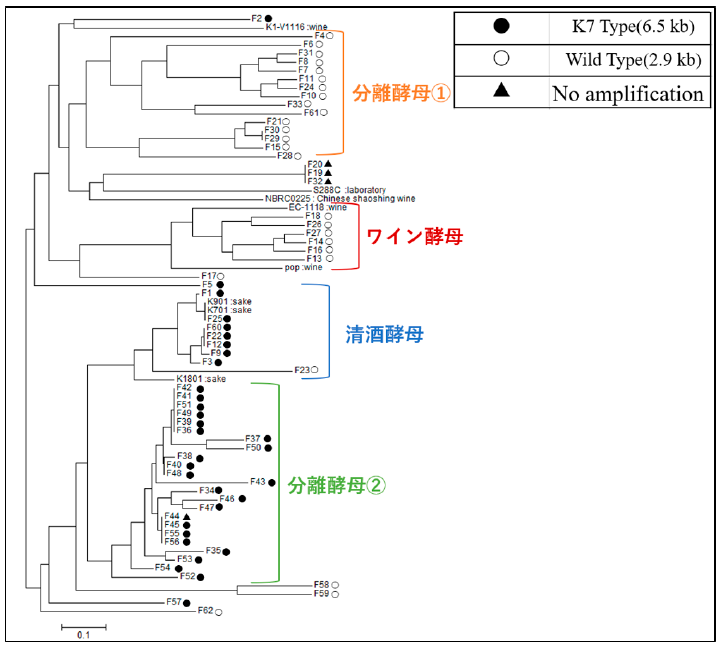
分離源である花は2018年春から2019年秋にかけて近畿大学の農学部キャンパスで採取した。スクリーニングは米麹汁培地を用いた集積培養法でおこない、種同定には26S rDNAのD1/D2領域の塩基配列を用いた。スクリーニングの結果、62株の野生酵母を分離した (分離株をF1からF62とする)。分子系統樹の作成には12のマイクロサテライト (数塩基の単純反復配列) 遺伝子座 (C5,C3, C8, C11, C9, SCYOR267c, YKL172w, ScAAT1, C4,SCAAT5, C6, YPL009c) のフラグメントサイズを利用し、近隣結合法で分子系統樹を作成した (図1)6,12)。

分離酵母は、分離酵母のみからなる独立した2つのクラスター (分離酵母①クラスター、分離酵母②クラスター)、清酒醸造に用いられるきょうかい酵母と分離酵母によって構成されるクラスター (清酒酵母クラスター)、醸造用ワイン酵母と分離酵母によって構成されるクラスター (ワイン酵母クラスター) の4つの主要なクラスターを形成した。
さらに分子系統樹の分離酵母の分布と分離時期との関係を調べたところ、ワイン酵母クラスターの株は2019年春に、分離酵母②クラスターの株は2019年秋に新規に取得された。これにより、採取時期が進むにつれて新たなクラスターが出現する傾向にあることがわかった。
3.きょうかい酵母との相違点・類似点
現在、日本全国で最も清酒醸造に使用されている酵母は日本醸造協会が頒布しているK7グループ株 (K6、K7、K9、K10およびその派生株) と呼ばれる酵母で、その特徴は低温での高いアルコール発酵能力、低い酢酸生成や高い香気成分生成が特徴であり淡麗な清酒の酒質向上に大きく貢献している。一方でK7グループ株は遺伝的に非常に近縁5)である。酵母は花の蜜をエサに繁殖する事から、地域オリジナル酵母の探索源として花から野生酵母のスクリーニングも行われている11,13)。
K7グループ株はいくつかの特徴的な配列を持ち、その中でも代表的なものが①PPT1遺伝子のトランスポゾンTy2への置き換わり、②プロテインキナーゼをコードするRIM15遺伝子のフレームシフト変異 (5055番目の塩基の直後にAが挿入し、キナーゼ活性の消失/低下)、③転写因子をコードするMSN4遺伝子のナンセンス変異 (1540番目の塩基がCからTに置換、転写活性化能の消失/低下) であり、これらの変異がK7グループ株の高アルコール発酵を担っていると考えられている14)。これらの3つの遺伝子座はいずれもホモ接合体としてK7グループの菌株に共通しているため、スクリーニングした酵母がK7グループ株と異なるかどうかの指標とすることができる5)。そこで、スクリーニングした野生酵母がK7グループ由来であるかどうかを確認するため、K7グループ株に特徴的なPPT1/Ty2領域のPCRフラグメント (6.5 kb) のサイズを調べた (図1)。
その結果、K7グループ株に特徴的なTy2に相当する約6.5 kbのPCR産物サイズを示すもの、PPT1遺伝子の大きさ約2.9 kbを示すもの、増幅バンドのないものの3パターンの分離酵母を確認することができた。野生酵母62株のうち32株がK7型を示し、これらの多くは分子系統樹において清酒酵母クラスターおよび野生酵母②クラスターに属した。また、2.9 kbの増幅を示す株は、ワイン酵母クラスターや分離酵母①クラスターに分布する傾向があった。例外も複数見つかり、1) F23は野生型の2.9 kbであるにもかかわらず、清酒酵母クラスターに属した。2) これまでの研究で報告されていないPPT1/Ty2領域の増幅が確認できない菌株を4株 (F19,F20,F32,F44) 確認することができた。
さらにK7グループ株を特徴づけるRIM15遺伝子、MSN4遺伝子内の変異の有無を調べた。その結果、PPT1/Ty2領域においてK7型を示した株のほとんどは、RIM15遺伝子・MSN4遺伝子に関してもK7型と同じ変異を持つことがわかり、PPT1/Ty2領域において野生型を示した株はRIM15遺伝子・MSN4遺伝子に関しても野生型を示した。
一方、例外となる株も見られた。1) F4株はPPT1/Ty2領域が野生型でありながら、RIM15遺伝子・MSN4遺伝子内の変異がK7型を示した。また、2) F43株、F50株はPPT1/Ty2領域及びRIM15遺伝子内変異がK7型でありながら、MSN4遺伝子のみが野生型を示すなど、今までに例が見られないK7とのハイブリッドと思われる株も確認された。
RIM15やMSN4遺伝子の変異は胞子形成能の喪失、胞子の発芽率の低下、ストレス耐性の低下、高アルコール発酵に関与しており、K7グループ株の表現型にも深く関与している14,15)。この観点から、野生酵母の胞子形成能を調べた所、K7型変異を持つ酵母株あるいは、K7ハイブリッド型株では、全ての株で胞子形成が見られず、K7型の特徴と一致した。一方、ワイン酵母、分離酵母①では、例外もあるが大部分の株が胞子形成を行うことができた。このことから、スクリーニングした酵母K7グループ由来かどうかの簡易判断材料として胞子形成を調べる事が有力な判別方法であることがわかった。また、本結果から、環境中にK7グループ株を祖先とする酵母が存在/繁殖し、野生型の酵母と交雑した結果、ハイブリッド型酵母が出現したと考えられる。新規酵母を求めてスクリーニングを行う際には、遺伝子型にも注意して、単離した酵母の選択を行えばよいことがわかった。
4.醸造特性解析
獲得した野生酵母の遺伝的な多様性はわかったので次に、実際の醸造において分離酵母間で差異があるのか確認するため、1段仕込みの清酒小仕込み試験 (α化米45 g、乾燥麹11 g、汲水歩合140%、15℃で14日間発酵) を行い、アルコール発酵能、香気成分量や有機酸量、アルコール濃度といった観点から醸造特性の評価を行った。分離酵母で醸造した清酒のアルコール濃度は5.7%から18.9%まで幅広いアルコール発酵力を示した。K7グループ株に特徴的な3つの遺伝子変異;PPT1遺伝子の欠失、RIM15フレームシフト変異、MSN4ナンセンス遺伝子変異は、K7グループ株特有の高アルコール発酵の原因遺伝子であると考えられている。K7型酵母から構成される清酒酵母クラスターに属する株のほとんどはアルコール濃度16%以上を有していた。一方、分離酵母②クラスターに属する株はK7型の遺伝子型を示すもののアルコール濃度はおおよそ13%~15%の株が多く、K7遺伝子型を持つ酵母が必ずしも高いアルコール発酵を示さないことがわかった。また、ワイン酵母クラスターに属する株及びクラスター外に位置するいくつかの株は、野生型でありながらアルコール濃度16%以上と高い値を示した。前述したように、K7型株はPPT1遺伝子の欠損及びRIM15遺伝子、MSN4遺伝子の変異は高アルコール発酵能に関与しているとされているが、ワイン酵母クラスターに属する株を筆頭とする野生型かつ高アルコール発酵を示す株は、清酒酵母とは異なる高アルコール発酵機構に関わる遺伝的形質を有している可能性が示唆された。例えば、RIM15pによるリン酸化のターゲットとして同定されたENSA (α-endosulfine) ファミリーに属するIgo1/2pの機能欠損も、RIM15pの機能欠損と同様にアルコール発酵力を上昇させることから16)、Igo1/2pを含めた他のリン酸化シグナル経路上のタンパク質に変異が起こっているのかもしれない。
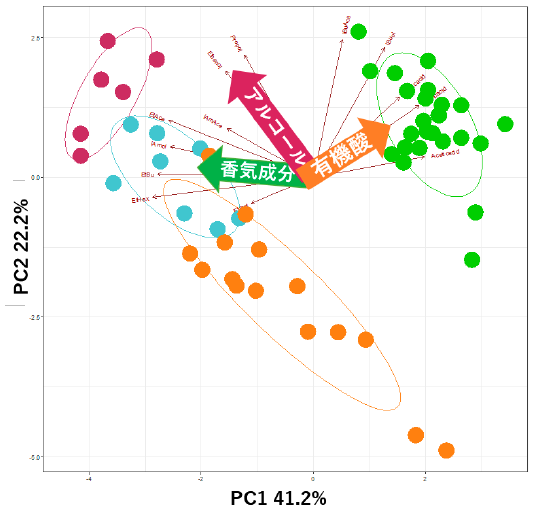
次に分離酵母間で醸造特性に差異があるのか確認するため、アルコール発酵能に加え、清酒小仕込み試験の成分分析結果 (有機酸3成分、香気成分9成分) を用いてPCA分析を行った (図2)。清酒酵母及びワイン酵母クラスターに属する酵母はPCA図の左上に位置し、高アルコール発酵、高香気成分生成 (酢酸イソアミル、イソアミルアルコール) 及び低有機酸という特徴を示した。一方で分離酵母②クラスターに属する酵母は右上に位置し、香気成分の量が少なく有機酸生産量が高い傾向が見られた。K7型の遺伝子変異を持つ分離酵母②クラスターは、胞子形成能も消失している。この形質は自然界での生存競争に勝つには不利な特徴と思われる。この弱点を補うため、自然界に進出/漏洩したK7型酵母株は自然界で生き延びるため、酢酸などの有機酸をより多く生成し、周りの環境のpHを下げる生存戦略をとっているのかもしれない。分離酵母①クラスターの酵母は中央下側に位置し、アルコール及び香気成分の生成がやや控えめである傾向を示した。また、各酵母クラスターはPCA図上でも同じようにクラスターを示した。このことから、マイクロサテライトを用いた分類と醸造特性は良い相関がある事がわかり、醸造を行う際の酵母選択の目安にすることがわかった。

5.柿ワインへの応用
野生酵母のスクリーニングから約60株の野生酵母を単離することができた。これらの酵母を利用し、柿を材料とした果実酒開発に取り組んだ。柿は奈良県の特産物であるが、生柿は季節性の果物で収穫と消費期間が限られており、加工品もドライフルーツがほとんどであり、新たな利用法が求められていた。近大農学部もまた奈良市にあり、地域連携・地域貢献の一環として奈良県と協力し柿を用いた県産オリジナルワインの開発に取り組んだ。分子系統樹の各グループから柿果実酒試験醸造で官能評価の良かった酵母株を複数選び、ワイナリー、柿生産業者、酒類卸売業者等、関係者と再度評価を行うことで酵母の最終選別を行った。ブランディングの事を考え、K7型の酵母は除外した。選別された酵母はPCA分析の結果から、香気成分や有機酸の生成がやや控えめである酵母であった。これは柿風味を殺さない事が良かったと考えられる。最終的には甘味果実酒として、アルコール度数が12%、甘酸っぱいテイストの果実酒が完成した。販売に関しては、ネーミング、ラベルデザインにもこだわり、近大・文芸学部の協力を仰ぎ、大学発の柿ワインとしてブランディングを行い「柿の音」として販売している。
6.まとめ
大学構内という狭い範囲から複数年にわたり分離した酵母の遺伝的多様性と醸造特性の幅はスクリーニングのたびに増加している。ただスクリーニングの継続によりさらにクラスターが増加し続けるのか、収束していくのかは不明である。本研究から意外と身近な環境からでもアルコール飲料産業へ利用できる酵母株をスクリーニングできることがわかった。またK7型グループは胞子形成をしないため、交雑法などで育種することが困難なことが知られている。しかし、本研究結果からK7/野生型ハイブリットと思われる株も取得されており、何らかの状況 (まだ知られていない) が揃えば交雑することが示唆された。これはK7型酵母との交雑条件のヒントが自然界にはあるかもしれない事を示している。また、分離酵母の中にはアルコール発酵能に関与しているとされているPPT1遺伝子、RIM15遺伝子、MSN4遺伝子の変異を持たず高アルコール発酵を示す株が確認でき、これらの株について調べていくことでK7グループ株とは異なる高アルコール発酵のメカニズムの解明の助けになると考えられる。本結果は2018年、2019年のスクリーニングによる単離酵母約60株の結果であるが、現在、100株を超えるS. cerevisiaeが新たに単離されており、解析を進めていく予定である。また、非サッカロ酵母も多数、単離しており、食品開発への利用可能性の検討を進めている。
謝辞
本研究の遂行にあたり、スクリーニング法、試験醸造、分析に関して多大なサポートをいただいた奈良県産業振興センターの皆様にお礼申し上げます。また、試験醸造、分析に関してご助言、サポートいただきました京都市産業技術研究所の皆様にこの場を借りてお礼申し上げます。また、本研究の一部は奈良県からの受託研究費「県産オリジナル柿ワイン開発事業」により行ったものである。
文献
1) Gallone, B., Steensels, J., Prahl, T., Soriaga, L., Saels, V., Herrera-Malaver, B., Merlevede, A., Roncoroni, M., Voordeckers, K., Miraglia, L., Teiling, C., Steffy, B., Taylor, M., Schwartz, A., Richardson, T., White, C., Baele, G., Maere, S., Verstrepen, K. J.: Cell, 166, 1397 (2016).
2) Gonçalves, M., Pontes, A., Almeida, P., Barbosa, R., Serra, M., Libkind, D., Hutzler, M., Gonçalves, P., Sampaio, J. P.: Curr. Biol., 26, 2750 (2016).
3) Borneman, A. R., Forgan, A. H., Kolouchova, R., Fraser, J. A., Schmidt, S. A.: G3 (Bethesda)., 6, 957 (2016).
4) Gallone, B., Mertens, S., Gordon, J. L., Maere, S., Verstrepen, K. J., Steensels, J.: Curr. Opin. Biotechnol., 49, 148 (2018).
5) 赤尾 健、周 延、渡辺大輔、岡崎直人、下飯 仁: J. Brew. Soc. Japan, 11, 631 (2018).
6) Legras, J. L., Merdinoglu, D., Cornuet, J. M., Karst, F.: Mol. Ecol., 16, 2091 (2007).
7) Liti, G., Barton, D. B., Louis, E. J.: Genetics, 174, 839 (2006).
8) Legras, J. L., Galeote, V., Bigey, F., Camarasa, C., Marsit, S., Nidelet, T., Sanchez, I., Couloux, A., Guy, J., Franco-Duarte, R., Marcet-Houben, M., Gabaldon, T., Schuller, D., Sampaio, J. P., Dequin, S.: Mol Biol Evol., 35, 1712 (2018).
9) Liti, G., Carter, D. M., Moses, A. M., Warringer, J., Parts, L., James, S. A., Davey, R. P., Roberts, I. N., Burt, A., Koufopanou, V., Tsai, I. J., Bergman, C. M., Bensasson, D., O’Kelly, M. J., van Oudenaarden, A., Barton, D. B., Bailes, E., Nguyen, A. N., Jones, M., Quail, M. A., Goodhead, I., Sims, S., Smith, F., Blomberg, A., Durbin, R., Louis, E. J.: Nature, 458, 337 (2009).
10) Abe, T., Toyokawa, Y., Sugimoto, Y., Azuma, H., Tsukahara, K., Nasuno, R., Watanabe, D., Tsukahara, M., Takagi, H.: Front. Genet., 10, 490 (2019).
11) Tsutsumi, S., Mochizuki, M., Sakai, K., Ieda, A., Ohara, R., Mitsui, S., Ito, A., Hirano, T., Shimizu, M., Kato, M.: Sci. Rep., 9, 13908 (2019).
12) Takezaki, N., Nei, M., Tamura, K.: Mol. Biol. Evol., 27, 747 (2010).
13) 都築正男、大橋正孝、清水浩美: 奈良県産業振興総合センター研究報告, 41, 5 (2015).
14) 渡辺大輔: 生物工学, 91, 2 (2013).
15) Fukuda, N., Takeuchi, M.: Yeast, 39, 272 (2022).
16) 渡辺大輔、高木博史: 生物工学, 98, 170 (2019).