【トピックス】
ゲノム時代に変わりゆくロドプシン観
井上圭一
東大・物性研
1.はじめに
光は地球上の大部分の生命の活動を支える最も根源的なエネルギーであり、生物はそのために様々な光受容タンパク質を利用している。その中でも、我々ヒトの視覚などに関わるのが、ロドプシン (Rhodopsin) と呼ばれ膜タンパク質型の光受容タンパク質である。これらは7本の膜貫通ヘリックスからなる、オプシンと呼ばれるタンパク質部分の中央に、発色団としてビタミンAの誘導体であるレチナール (Retinal) を結合する1,2)。そして、レチナールが光を吸収するとタンパク質内部で異性化を起こし、それに応じてオプシン部分が構造を変化させることで、多様な生理機能が発現する。
歴史的に、最も早くに報告がなされ、私達にとって馴染み深いのが、動物の網膜中の視細胞に存在し、光受容体として働く視覚ロドプシンであり、19世紀にはその存在が知られていた3,4)。そして20世紀の中頃、George Waldらの主にウシのロドプシン (Bovine rhodopsin、bvRh) を用いた研究によって、視覚の初期過程がロドプシン中のレチナールの光異性化であることが明らかとなった5)。Waldはこれら一連の研究によって、1967年にノーベル生理学・医学賞を授賞し、今ではロドプシンは視細胞内のヘテロ三量体Gタンパク質 (Transducin、Gt) を光依存的に活性化する、Gタンパク質共役型受容体 (GPCR) の一種であることも分かっている。一方で、Waldがノーベル賞を受賞した直後、当時の研究者が思いもしなかったところから、ロドプシン研究は新たな局面を迎える。
それが1971年に、Walther StoeckeniusとDieter Oesterheltによる、高度好塩性アーキア (古細菌) Halobacterium salinarumが持つ、動物のロドプシンに似て7本のヘリックスとレチナールからなる膜タンパク質の発見であった6)。当時はバクテリア (細菌) とアーキアの区別が為されていなかったことから、この分子はバクテリオロドプシン (Bacteriorhodopsin、BR) と名付けられた。目を持たないアーキアが、なぜ動物ロドプシンに似た分子を持つのか、当初その理由は不明であった、その後のStoeckeniusとOesterheltの研究によって、BRは光をエネルギー源として、H+を細胞内から細胞外側へ能動的に輸送する、光駆動型のH+ポンプであることが明らかとなった7)。これにより、H. salinarumはBRを用いることで、ATP合成などの生命活動に必要なエネルギーを光から得ることができる8)。
1-1 20世紀までのロドプシン観
BRの発見に続いて、H. salinarumは光駆動型塩化物イオン (Cl−) ポンプのハロロドプシン (Halorhodopsin、HR) 9,10)と走光性のセンサーとしてはたらく2つのセンサリーロドプシン (SRI、SRII) を持つことが明らかとされた11-13)。またH. salinarumに近縁な多くの高度好塩性アーキアが同様のロドプシンを持つことが明らかとなり14)、現在ではこれらは総称して微生物ロドプシンと呼ばれる。
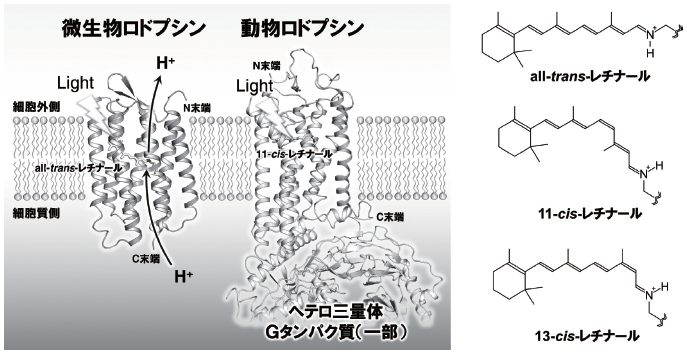
動物と微生物、2種類のロドプシンは、光で反応を開始できるという特長を活かして、時代ごとの最先端の分光法を用いることで、光受容タンパク質のモデル分子として数多くの研究でそのメカニズムが調べられた。これにより2つのロドプシンのレチナールが、光照射後300fs以下の極めて短い時間で異性化することや、BRが複数のアスパラギン酸やグルタミン酸残基を用いて多段階的にH+を輸送することなどが、極めて精緻に明らかになっている (図1) 15-17) (ちなみにBRのH+輸送メカニズムとその赤外分光研究については、名古屋工業大学・神取秀樹特別教授が、以前本誌の第47号のトピックスで詳しい解説をされている18)ので、興味のある方はそちらをぜひご一読いただきたい)。
図1 微生物ロドプシン (BR)と動物ロドプシン (bvRh) (左)、および主なレチナールの異性体の構造 (右)
1997年の最初のBRのX線結晶構造解析19)に続き、1999年には高分解能の構造が決定され20)、さらに20世紀最後の年である2000年にはウシロドプシンの結晶構造が明らかになったことで、ロドプシン研究は一つのピークを迎えた21)。そして、当時は中間体の構造など、興味ある未解決問題は残されているものの、それらが解決され、bvRhとBRの二大分子の主要な部分の理解が為されるのは時間の問題と思われた。
2. ゲノム科学がもたらした二つの発見:プロテオロドオプシンとチャネルロドプシン
2-1 海洋の微生物ロドプシン
H. salinarum由来の4つの微生物ロドプシンは、多くの研究の対象とされたことで、7回膜貫通型構造とレチナール発色団から、H+ポンプ、Cl−ポンプ、正および負の走光性センサーといった全く異なる機能が発現する分子メカニズムの理解が進められ、タンパク質の機能-構造相関研究のモデル系としての地位を確立した。一方で、これらを有する生物種は塩湖などの極限環境に棲息する高度好塩性アーキアに限定されていたことから、微生物ロドプシンの生物界全体における分布は狭く、自然界への寄与はあまり高くないと考えられていた。
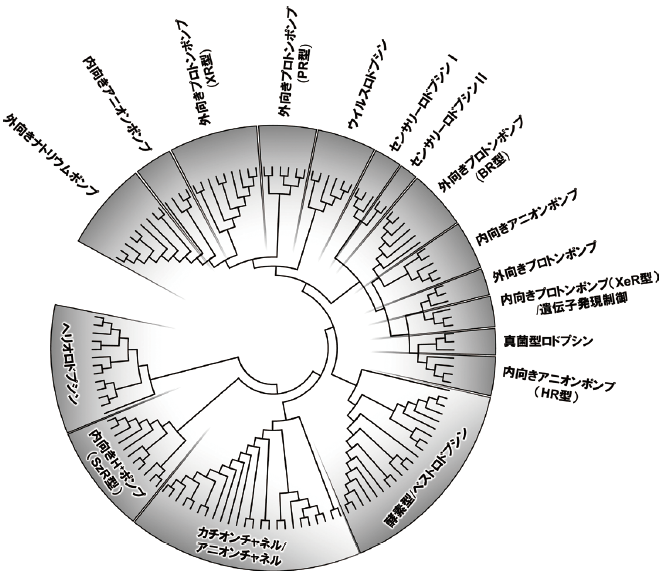
その中で、1999年に真核生物であるアカパンカビ (Neurospora crassa) のcDNAライブラリから、微生物ロドプシン様の遺伝子が発見された22)。このタンパク質 (Neurospora rhodopsin、NR) はイオンポンプではなく光センサーであると考えられており23)、その詳しい分子機能はいまだ不明だが、高度好塩性アーキア以外にも微生物ロドプシンはかなり広汎に存在することが示唆され、NRの発見は驚きを持って迎えられた (図2)。
図2 微生物ロドプシンの分子系統樹
そして、さらに大きな発見が2000年にEdward F. DeLong、Oded Béjàらによってもたらされた。彼らは当時技術が確立したばかりのメタゲノム解析によって、世界中の海洋に広汎に棲息するγ-プロテオバクテリアが微生物ロドプシン様遺伝子を持ち、さらにそれがBRと同様の光駆動型の外向きH+ポンプ機能を有することを明らかにした24,25)。このロドプシンは新たにプロテオロドオプシン (Proteorhodopsin、PR) と名付けられたが、PRの発見は、海洋の炭素循環に投入される太陽光のエネルギーは、クロロフィルをベースとした光合成によるものであるという、これまでの認識を大きく変化させるものであった。現在では、光が届く水深に棲むバクテリアやアーキアのおよそ半分が、PRなどのH+ポンプ型ロドプシンを有しているとされており26)、生物界における微生物ロドプシンの分布はそれまでの認識より格段に広いことが分かった。
2-2 チャネルロドプシンとオプトジェネティクス
NRやPRの発見に端を発し、幅広い微生物から微生物ロドプシンの探索が進む中、日本のかずさDNA研究所から公開された、クラミドモナス (Chlamydomonas reinhardtii) のcDNAデータベース中に、微生物ロドプシン様の遺伝子が多数含まれていることが見出された27,28)。そして、その中で青色光受容体として働く2つの微生物ロドプシンが、光開閉式のカチオンチャネルであることが、Peter Hegemann、Georg Nagelらの電気生理学実験によって明らかとなった29,30)。これら2つのロドプシンはチャネルロドプシン (C. reinhardtii channelrhodopsin 1 および2、Cr ChR1および2) と名付けられた。C. reinhardtiiなどの緑藻は、古くから青色光に対する走光性を示すことが知られており31)、様々な研究からその光受容体の実体は眼点に存在するロドプシンではないかと考えられていたが32)、Cr ChRのイオン輸送によって光依存的に細胞の膜電位変化やCa2+流入を引き起こすことがその実体であることが初めて明らかとなった33)。
これらChRの発見は、それを動物の神経細胞などに発現させることで、BRなどでは困難な神経細胞の脱分極、すなわち神経の興奮を、光を使って自在に誘起できることを示唆しており、その可能性については最初の論文でも提案されていた29)。そして、Karl Deisserothらと八尾寛らのそれぞれ独立の研究によって、実際にCrChR2を用いることで、マウスの神経細胞を光で発火させられることが示された34,35)。動物の神経活動やそれに付随する行動を光で操作する、オプトジェネティクス (光遺伝学) の幕開けである。一方で、外向きH+型や内向きCl−型のロドプシンを細胞に発現させ、そこに光を照射すると、膜電位が過分極し、CrChR2とは逆に神経興奮が抑制される36,37)。これにより、微生物ロドプシンを用いることで、光で神経を興奮させる、もしくはその活動を抑制することが可能となり、神経操作の時空間分解能が飛躍的に向上された。そして、オプトジェネティクスは神経生理学研究において、今日では不可欠な技術となっている。
しかし、最初に用いられたCrChR2は光感度や応答速度、応答波長、イオン輸送量、イオン選択性、細胞内局在などの物性が、理想的な光操作を達成するには十分なものではなかった。そのため、より短波長および長波長光に応答するものや38-43)、イオン輸送量や光感度に優れたものなど44-46)、様々なChRが、ゲノム情報を活用したゲノムマイニングやタンパク質工学的な分子改変によって、探索・開発されている。とりわけ、陰イオンを輸送するアニオンチャネルロドプシン (ACR) や47-49)、カリウムイオン (K+) 選択的なカリウムチャネルロドプシン (KCR) は50,51)、それまでイオンポンプが用いられていた抑制性オプトジェネティクスツールのイオン輸送量を飛躍的に向上させ、極めて弱い強度の光を用いた、光毒性の低い神経抑制を可能としている。また最近、筆者らは、名古屋大学・竹内一郎教授、名古屋工業大学・烏山昌幸准教授らとともに、アミノ酸配列のみからその吸収波長が予測可能な機械学習モデルの開発を行っており、これによりオプトジェネティクスツール開発の効率を格段に向上できると期待される52,53)。
これら、ゲノムデータをもとにしたPRとChRの発見は、bvRhとBRを基軸としたロドプシ研究の様相を一変させ、生態系におけるロドプシンの重要性と、新規分子ツールとしての有用性にパラダイムシフトをもたらした。
3.常識を覆す微生物ロドプシンの発見
3-1 微生物ロドプシンの多様なイオン輸送とメカニズム
PRやChRの他にも、次世代シーケンサーなどの進歩に伴って、数多くの微生物ロドプシン様遺伝子が発見されており、今日ではその数は数十万に及ぶ。その中で、高度好塩性バクテリアのSalinibacter ruberが持つキサントロドプシン (Xanthorhodopsin、XR) はレチナールに加え、カロテノイドの一種であるSalinixanthin (SXN) を結合する54)。このSXNが光を吸収すると、165 fsの超高速のエネルギー移動によってレチナールが励起され、そのエネルギーがH+輸送に用いられる54,55)。これにより、XRはレチナール単独の時よりも格段に幅広い波長の光に応答することができ、SXNの高いモル吸光係数によってその効率も極めて高いとされている。その後長い間、XRの様にカロテノイドを利用するロドプシンは数種類に限られると考えられていたが56,57)、2023年の大規模なスクリーニング実験によって、PRやXRのグループに属するロドプシンのかなりの割合 (環境などによって変動するが18~82%程度) が、カロテノイドを利用していることが明らかとなった58)。
一方、筆者らのグループは東京湾に棲む海洋性の細菌から、Na+イオンを外向きに輸送するNa+ポンプ型ロドプシン (NaR) を新たに発見した59)。また深海性の細菌であるParvularcula oceaniが持つゼノロドプシン (XeR)60)と、真核生物の共通祖先に最も近い現生アーキアであるとされる、アスガルドアーキアから発見され、XeRとは独立した、新しい系統のロドプシンが内向きH+ポンプ機能を持つことを報告した61)。後者のものは、系統学的に後述するヘリオロドプシンと、従来の微生物ロドプシンとの中間的な位置に存在することから、英語の“split”を意味するギリシャ語の“schizo”を冠した、シゾロドプシン (Schizorhodopsin、SzR) と名付けられた。またHRとは異なる系統の内向きCl−ポンプ62,63)や硫酸イオン (SO42−) ポンプ64,65)なども相次いで発見されており、イオンポンプ型の微生物ロドプシンは、微生物ロドプシン全体の中で最も数が多く、またその機能も多様性に富んでいることが分かっている。
3-2 他のドメインと共同して働く微生物ロドプシン
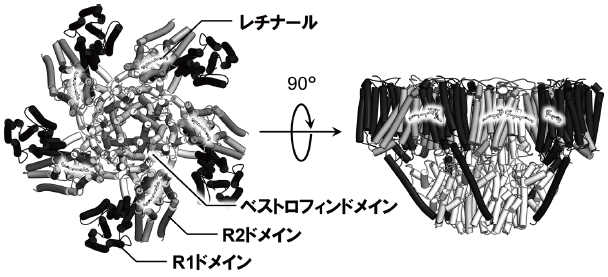
これまでロドプシンは単独でイオン輸送や、シグナル伝達を行うと考えられていた。しかし、クラミドモナスのcDNA中に発見されたロドプシンのいくつかは、C末端側にシグナル伝達に関わるとされるヒスチジンキナーゼや応答制御因子、アデニル酸/グアニル酸シクラーゼ (AC/GC) が融合した、これまでにない配列を持っていた28)。このロドプシンはヒスチジンキナーゼロドプシン (Histidine kinase rhodopsin、HKR) と呼ばれ、ロドプシンが他の機能性ドメインと結合してその活性を光依存的に制御する、新しいタイプの分子であることが示唆された。実際にその様な機能性が初めて示されたのは、真菌の一種Blastocladiella emersonii由来で、ロドプシンのC末端側にGCドメインが融合した、ロドプシングアニル酸シクラーゼ (Rhodopsin–guanylyl cyclase、Rh-GC) であり、ロドプシンドメインが光を吸収するとGC部分の活性が向上する66)。さらにその後、縦襟鞭毛虫からロドプシンホスホジエステラーゼ (Rhodopsinphosphodiesterase、Rh-PDE) が見付かり、光依存的なcAMP/cGMP加水分解反応を示すことが報告された67)。これらのロドプシンの発見により、ロドプシンは単独で存在するだけでなく、他のドメインと融合することで、よりその機能性を拡張させていることが明らかとなった。そして、酵素ドメインではなく、1~2個のロドプシンにイオンチャネルであるベストロフィンが融合したベストロドプシン (Bestrhodopsin) が海洋性の藻類から見付かり、ChRとは全く異なる機構のイオン輸送を行う68)。この分子では、ベストロフィン部分が5量体を作ることで、中央に巨大なイオンチャネルが形成され、さらにその周囲を最大10個のロドプシンが囲む巨大な複合体となる (図3)。
図3 ベストロドプシンのクライオ電子顕微鏡構造
4.第3のロドプシン:ヘリオロドプシン
2018年、既知の動物および微生物いずれのロドプシンとも進化的に異なる第3のロドプシンファミリーがOded Béjàらの機能性メタゲノミクスによって発見され、ギリシャ語で太陽を意味するHeliosをもとに、ヘリオロドプシン (Heliorhodopsin、HeR) と名付けられた69)。このHeRが他のロドプシンと比べて、最も大きく異なる点として、細胞膜中でのタンパク質の配向が挙げられる。すなわち、既知のロドプシンは動物型も微生物型もN末端とC末端をそれぞれ細胞外側と内側に向ける形で存在するが (細胞内を起点として、タンパク質合成の開始点であるN末端が奇数回細胞膜をくぐると最終的に細胞外に出るため、7回膜貫通型のロドプシンがこの様な配向を持つのは自然である)、HeRではこれとは逆に、N末端が細胞内に、C末端が細胞外に向いた配向となっている69,70)。
またその後、日・欧・米中の三つのグループからほぼ同時にHeRの構造が報告され、巨大な細胞質ループが互いを橋渡しする、特徴的な構造を持つことが明らかとなった70-72)。一方で、HeRのほとんどはその機能がいまだ不明である。だがHeRの持つ特殊な配向はその機能と密接に関わると考えられており、タンパク質の細胞質側を用いた細胞内シグナルネットワークへのシグナル伝達や70)、細胞内の低分子に対する酸化還元反応71,73)、両親媒性分子の輸送74)などに関わる可能性が示唆されている。その中で、一部の巨大ウイルス由来のHeRが光依存的にH+を輸送したり75)、バクテリアのHeRがグルタミン合成酵素や光回復酵素の活性を光依存的に向上させることが報告されており76,77)、少しずつその機能が解明されつつある。
5.非視覚動物ロドプシン
微生物ロドプシンだけでなく、動物ロドプシンにおいても多様な生物種のゲノム解析が急速に進んだ結果、数千種類の遺伝子が同定されている78)。例えばヒトの場合、視覚ロドプシンに加え、光強度の高いところで色の認識に関わる、3種類の色覚視物質と呼ばれる動物ロドプシンがあることが古くから知られていた79)。さらに、ヒトゲノム上には全9種類の動物ロドプシン様遺伝子があることがわかっており、動物界全体では、分類の方法にもよるが10あまりのグループに大別される80)。中でも、メラノプシン (Opn4とも呼ばれる) が瞳孔制御や概日リズム調整など様々な非視覚応答に81)、Opn3がメラニン色素の合成の調節82)や、光依存的な褐色脂肪細胞の代謝活性制御に関わる83)ことが報告されており、非視覚動物ロドプシンの役割が次第に明らかとなってきている。
一方、イカなどの無脊椎動物においては、レチノクロムが、視覚ロドプシンに必要な11-cis型のレチナールを、all-trans-レチナールから生成する光異性化酵素としてはたらくことが古くから知られていた84)。一方、脊椎動物では、11-cis-レチナールは別種の酵素によって光非依存的に作られると考えられてきたが、動物ロドプシンの一種Retinal G-protein-coupled receptor (RGR) もレチノクロム同様の光異性化酵素機能を持つことが最近明らかとなっている85,86)。
また、脊椎動物の視覚ロドプシンは光を受けると、11-cis-レチナールがall-trans-レチナールへと異性化後、タンパク質から脱離するが、無脊椎動物の視覚ロドプシンや多くの非視覚ロドプシンではall-transレチナールが解離せず、再び光子を吸収するともとの11-cis型へ戻る双安定性を示す1)。それ以外に、光で生成したall-transレチナールは解離しないが光でもとの11-cis型へ戻らないbleach-resist型87)や、光吸収後レチナールが近傍のシステイン残基と共有結合を形成する動物ロドプシン88)が報告されており、レチナールの光反応の多様性も大きく広がっている。
そして動物ロドプシンには光活性化すると一分子で多数のGタンパク質を活性化するシグナル増幅機構があり、これによって微生物ロドプシンよりも高い光感度を持つことから、オプトジェネティクスツールとしての利用も数多く報告されている89-91)。この様に動物ロドプシンにおいても、従来の視覚ロドプシンとは大きく異なる機能や物性を持つ分子が数多く報告されており、今後新たな発見のスピードはより速くなると思われる。
6.おわりに
筆者がまだ大学院生であった2005年ごろ、共同研究で米国の研究室に滞在した際、「米国ではもうBRの研究では研究費が獲得できなくなっている」と言われ驚いたことを鮮明に記憶している。そして、その後20年弱の間に起こった急激なロドプシン研究の拡大と発展は、当時は想像することもできないものであった。ロドプシン分野では2年に一度、150名程度が参加するレチナールタンパク質国際会議 (ICRP) が開催されるが、コロナ禍での延期を経て、前回2022年11月に札幌で開催された会議では、極めて刺激的な新事実が数多く報告され、ロドプシン研究の勢いは全く衰えを見せていない (次回のICRPは2024年スイスでの開催が予定されている)。筆者の研究室でも毎日のように新しい配列を持つロドプシンや、新規な発見が研究室のメンバーや世界中の共同研究者からもたらされている。今後のロドプシン研究のさらなる発展と可能性に興味は尽きない。
謝辞
本稿で一部紹介したロドプシンに関する筆者らの研究は、筆者が2018年まで在籍していた名古屋工業大学・神取秀樹特別教授の研究室のメンバーの方々、および2018年から現在まで、東京大学物性研究所において筆者が主宰する研究室のメンバー、ならびに研究室外の多数の共同研究者の方々との研究によって達成されたものです。これら全ての方々に、この場を借りて厚く御礼申し上げます。また紙面の都合上、全てのロドプシン研究を紹介することが適わず、関連の方々には深くお詫び申し上げます。
文献
1) Nagata, T., Inoue, K.: J. Cell Sci., 134, jcs258989 (2021).
2) Spudich, J. L., Jung, K.-H.: Handbook of Photosensory Receptors, 1 (2005).
3) Boll, F.: Monatsber. Akad. Wissensch. Berlin, 783 (1876).
4) Kühne, W.: Hermann’s Handbuch der Physiologie, 3, 235 (1879).
5) Wald, G.: Nature, 219, 800 (1968).
6) Oesterhelt, D., Stoeckenius, W.: Nat. New Biol., 233, 149 (1971).
7) Oesterhelt, D., Stoeckenius, W.: Proc. Natl. Acad. Sci. USA, 70, 2853 (1973).
8) Racker, E., Stoeckenius, W.: J. Biol. Chem., 249, 662 (1974).
9) Matsuno-Yagi, A., Mukohata, Y.: Biochem. Biophys. Res. Commun., 78, 237 (1977).
10) Lanyi, J. K., Oesterhelt, D.: J. Biol. Chem., 257, 2674 (1982).
11) Bogomolni, R. A., Spudich, J. L.: Proc. Natl. Acad. Sci. USA, 79, 6250 (1982).
12) Takahashi, T., Tomioka, H., Kamo, N., Kobatake, Y.: FEMS Microbiol. Lett., 28, 161 (1985).
13) Spudich, E. N., Sundberg, S. A., Manor, D., Spudich, J. L.: Proteins, 1, 239 (1986).
14) Mukohata, Y., Ihara, K., Uegaki, K., Miyashita, Y., Sugiyama, Y.: Photochem. Photobiol., 54, 1039 (1991).
15) Schoenlein, R. W., Peteanu, L. A., Mathies, R. A., Shank, C. V.: Science, 254, 412 (1991).
16) Mathies, R. A., Brito Cruz, C. H., Pollard, W. T., Shank, C. V.: Science, 240, 777 (1988).
17) Ernst, O. P., Lodowski, D. T., Elstner, M., Hegemann, P., Brown, L. S., Kandori, H.: Chem. Rev., 114, 126 (2014).
18) 神取秀樹: 酵素工学ニュース, 47, 17 (2002).
19) Pebay-Peyroula, E., Rummel, G., Rosenbusch, J. P., Landau, E. M.: Science, 277, 1676 (1997).
20) Luecke, H., Schobert, B., Richter, H. T., Cartailler, J. P., Lanyi, J. K.: J. Mol. Biol., 291, 899 (1999).
21) Palczewski, K., Kumasaka, T., Hori, T., Behnke, C. A., Motoshima, H., Fox, B. A., Le Trong, I., Teller, D. C., Okada, T., Stenkamp, R. E., Yamamoto, M., Miyano, M.: Science, 289, 739 (2000).
22) Bieszke, J. A., Braun, E. L., Bean, L. E., Kang, S., Natvig, D. O., Borkovich, K. A.: Proc. Natl. Acad. Sci. USA, 96, 8034 (1999).
23) Bieszke, J. A., Spudich, E. N., Scott, K. L., Borkovich, K. A., Spudich, J. L.: Biochemistry, 38, 14138 (1999).
24) Béjà, O., Aravind, L., Koonin, E. V., Suzuki, M. T., Hadd, A., Nguyen, L. P., Jovanovich, S. B., Gates, C. M., Feldman, R. A., Spudich, J. L., Spudich, E. N., DeLong, E. F.: Science, 289, 1902 (2000).
25) Rozenberg, A., Inoue, K., Kandori, H., Béjà, O.: Annu. Rev. Microbiol., 75, 427 (2021).
26) Finkel, O. M., Béjà, O., Belkin, S.: ISME J., 7, 448 (2013).
27) Asamizu, E., Nakamura, Y., Sato, S., Fukuzawa, H., Tabata, S.: DNA Res., 6, 369 (1999).
28) Kateriya, S., Nagel, G., Bamberg, E., Hegemann, P.: News Physiol. Sci., 19, 133 (2004).
29) Nagel, G., Ollig, D., Fuhrmann, M., Kateriya, S., Musti, A. M., Bamberg, E., Hegemann, P.: Science, 296, 2395 (2002).
30) Nagel, G., Szellas, T., Huhn, W., Kateriya, S., Adeishvili, N., Berthold, P., Ollig, D., Hegemann, P., Bamberg, E.: Proc. Natl. Acad. Sci. USA, 100, 13940 (2003).
31) Faminzin, A.: Melanges Biologiques tires du Bulletin de l’Ácademie Imperial des Sciences De St.-Petersbourg, 6, 73 (1866).
32) Foster, K. W., Saranak, J., Patel, N., Zarilli, G., Okabe, M., Kline, T., Nakanishi, K.: Nature, 311, 756 (1984).
33) Mittelmeier, T. M., Boyd, J. S., Lamb, M. R., Dieckmann, C. L.: J. Cell Biol., 193, 741 (2011).
34) Boyden, E. S., Zhang, F., Bamberg, E., Nagel, G., Deisseroth, K.: Nat. Neurosci., 8, 1263 (2005).
35) Ishizuka, T., Kakuda, M., Araki, R., Yawo, H.: Neurosci. Res., 54, 85 (2006).
36) Chow, B. Y., Han, X., Dobry, A. S., Qian, X., Chuong, A. S., Li, M., Henninger, M. A., Belfort, G. M., Lin, Y., Monahan, P. E., Boyden, E. S.: Nature, 463, 98 (2010).
37) Gradinaru, V., Thompson, K. R., Deisseroth, K.: Brain Cell Biol., 36, 129 (2008).
38) Lin, J. Y., Knutsen, P. M., Muller, A., Kleinfeld, D., Tsien, R. Y.: Nat. Neurosci., 16, 1499 (2013).
39) Schneider, F., Grimm, C., Hegemann, P.: Annu. Rev. Biophys., 44, 167 (2015).
40) Marshel, J. H., Kim, Y. S., Machado, T. A., Quirin, S., Benson, B., Kadmon, J., Raja, C., Chibukhchyan, A., Ramakrishnan, C., Inoue, M., Shane, J. C., McKnight, D. J., Yoshizawa, S., Kato, H. E., Ganguli, S., Deisseroth, K.: Science, 365, eaaw5202 (2019).
41) Tashiro, R., Sushmita, K., Hososhima, S., Sharma, S., Kateriya, S., Kandori, H., Tsunoda, S. P.: Commun. Biol., 4, 235 (2021).
42) Kato, H. E., Kamiya, M., Sugo, S., Ito, J., Taniguchi, R., Orito, A., Hirata, K., Inutsuka, A., Yamanaka, A., Maturana, A. D., Ishitani, R., Sudo, Y., Hayashi, S., Nureki, O.: Nat. Commun., 6, 7177 (2015).
43) Oda, K., Vierock, J., Oishi, S., Rodriguez-Rozada, S., Taniguchi, R., Yamashita, K., Wiegert, J. S., Nishizawa, T., Hegemann, P., Nureki, O.: Nat. Commun., 9, 3949 (2018).
44) Yizhar, O., Fenno, L. E., Prigge, M., Schneider, F., Davidson, T. J., O’Shea, D. J., Sohal, V. S., Goshen, I., Finkelstein, J., Paz, J. T., Stehfest, K., Fudim, R., Ramakrishnan, C., Huguenard, J. R., Hegemann, P., Deisseroth, K.: Nature, 477, 171 (2011).
45) Yizhar, O., Fenno, L. E., Davidson, T. J., Mogri, M., Deisseroth, K.: Neuron, 71, 9 (2011).
46) Hososhima, S., Ueno, S., Okado, S., Inoue, K. I., Konno, M., Yamauchi, Y., Inoue, K., Terasaki, H., Kandori, H., Tsunoda, S. P.: Sci. Rep., 13, 7625 (2023).
47) Govorunova, E. G., Sineshchekov, O. A., Janz, R., Liu, X., Spudich, J. L.: Science, 349, 647 (2015).
48) Oppermann, J., Fischer, P., Silapetere, A., Liepe, B., Rodriguez-Rozada, S., Flores-Uribe, J., Peter, E., Keidel, A., Vierock, J., Kaufmann, J., Broser, M., Luck, M., Bartl, F., Hildebrandt, P., Wiegert, J. S., Béjà, O., Hegemann, P., Wietek, J.: Nat. Commun., 10, 3315 (2019).
49) Rozenberg, A., Oppermann, J., Wietek, J., Fernandez, Lahore, R. G., Sandaa, R. A., Bratbak, G., Hegemann, P., Béjà, O.: Curr. Biol., 30, 4910 (2020).
50) Govorunova, E. G., Gou, Y., Sineshchekov, O. A., Li, H., Lu, X., Wang, Y., Brown, L. S., St-Pierre, F., Xue, M., Spudich, J. L.: Nat. Neurosci., 25, 967 (2022).
51) Vierock, J., Peter, E., Grimm, C., Rozenberg, A., Chen, I. W., Tillert, L., Castro, Scalise, A. G., Casini, M., Augustin, S., Tanese, D., Forget, B. C., Peyronnet, R., Schneider-Warme, F., Emiliani, V., Béjà, O., Hegemann, P.: Sci. Adv., 8, eadd7729 (2022).
52) Karasuyama, M., Inoue, K., Nakamura, R., Kandori, H., Takeuchi, I.: Sci. Rep., 8, 15580 (2018).
53) Inoue, K., Karasuyama, M., Nakamura, R., Konno, M., Yamada, D., Mannen, K., Nagata, T., Inatsu, Y., Yawo, H., Yura, K., Béjà, O., Kandori, H., Takeuchi, I.: Commun. Biol., 4, 362 (2021).
54) Balashov, S. P., Imasheva, E. S., Boichenko, V. A., Anton, J., Wang, J. M., Lanyi, J. K.: Science, 309, 2061 (2005).
55) Polívka, T., Balashov, S. P., Chábera, P., Imasheva, E. S., Yartsev, A., Sundström, V., Lanyi, J. K.: Biophys. J., 96, 2268 (2009).
56) Balashov, S. P., Imasheva, E. S., Choi, A. R., Jung, K. H., Liaaen-Jensen, S., Lanyi, J. K.: Biochemistry, 49, 9792 (2010).
57) Kopejtka, K., Tomasch, J., Kaftan, D., Gardiner, A. T., Bína, D., Gardian, Z., Bellas, C., Dröge, A., Geffers, R., Sommaruga, R., Koblízvek, M.: Proc. Natl. Acad. Sci. USA, 119, e2211018119 (2022).
58) Chazan, A., Das, I., Fujiwara, T., Murakoshi, S., Rozenberg, A., Molina-Márquez, A., Sano, F. K., Tanaka, T., Gómez-Villegas, P., Larom, S., Pushkarev, A., Malakar, P., Hasegawa, M., Tsukamoto, Y., Ishizuka, T., Konno, M., Nagata, T., Mizuno, Y., Katayama, K., Abe-Yoshizumi, R., Ruhman, S., Inoue, K., Kandori, H., León, R., Shihoya, W., Yoshizawa, S., Sheves, M., Nureki, O., Béjà, O.: Nature, 615, 535 (2023).
59) Inoue, K., Ono, H., Abe-Yoshizumi, R., Yoshizawa, S., Ito, H., Kogure, K., Kandori, H.: Nat. Commun., 4, 1678 (2013).
60) Inoue, K., Ito, S., Kato, Y., Nomura, Y., Shibata, M., Uchihashi, T., Tsunoda, S. P., Kandori, H.: Nat. Commun., 7, 13415 (2016).
61) Inoue, K., Tsunoda, S. P., Singh, M., Tomida, S., Hososhima, S., Konno, M., Nakamura, R., Watanabe, H., Bulzu, P. A., Banciu, H. L., Andrei, A., Uchihashi, T., Ghai, R., Béjà, O., Kandori, H.: Sci. Adv., 6, eaaz2441 (2020).
62) Yoshizawa, S., Kumagai, Y., Kim, H., Ogura, Y., Hayashi, T., Iwasaki, W., DeLong, E. F., Kogure, K.: Proc. Natl. Acad. Sci. USA, 111, 6732 (2014).
63) Hasemi, T., Kikukawa, T., Kamo, N., Demura, M.: J. Biol. Chem., 291, 355 (2016).
64) Niho, A., Yoshizawa, S., Tsukamoto, T., Kurihara, M., Tahara, S., Nakajima, Y., Mizuno, M., Kuramochi, H., Tahara, T., Mizutani, Y., Sudo, Y.: J. Am. Chem. Soc., 139, 4376 (2017).
65) Singh, M., Ito, S., Hososhima, S., Abe-Yoshizumi, R., Tsunoda, S. P., Inoue, K., Kandori, H.: J. Phys. Chem. B, 127, 7123 (2023).
66) Scheib, U., Stehfest, K., Gee, C. E., Körschen, H. G., Fudim, R., Oertner, T. G., Hegemann, P.: Sci. Signal., 8, rs8 (2015).
67) Yoshida, K., Tsunoda, S. P., Brown, L. S., Kandori, H.: J. Biol. Chem., 292, 7531 (2017).
68) Rozenberg, A., Kaczmarczyk, I., Matzov, D., Vierock, J., Nagata, T., Sugiura, M., Katayama, K., Kawasaki, Y., Konno, M., Nagasaka, Y., Aoyama, M., Das, I., Pahima, E., Church, J., Adam, S., Borin, V. A., Chazan, A., Augustin, S., Wietek, J., Dine, J., Peleg, Y., Kawanabe, A., Fujiwara, Y., Yizhar, O., Sheves, M., Schapiro, I., Furutani, Y., Kandori, H., Inoue, K., Hegemann, P., Béjà, O., Shalev-Benami, M.: Nat. Struct. Mol. Biol., 29, 592 (2022).
69) Pushkarev, A., Inoue, K., Larom, S., Flores-Uribe, J., Singh, M., Konno, M., Tomida, S., Ito, S., Nakamura, R., Tsunoda, S. P., Philosof, A., Sharon, I., Yutin, N., Koonin, E. V., Kandori, H., Béjà, O.: Nature, 558, 595 (2018).
70) Shihoya, W., Inoue, K., Singh, M., Konno, M., Hososhima, S., Yamashita, K., Ikeda, K., Higuchi, A., Izume, T., Okazaki, S., Hashimoto, M., Mizutori, R., Tomida, S., Yamauchi, Y., Abe-Yoshizumi, R., Katayama, K., Tsunoda, S. P., Shibata, M., Furutani, Y., Pushkarev, A., Béjà, O., Uchihashi, T., Kandori, H., Nureki, O.: Nature, 574, 132 (2019).
71) Kovalev, K., Volkov, D., Astashkin, R., Alekseev, A., Gushchin, I., Haro-Moreno, J. M., Chizhov, I., Siletsky, S., Mamedov, M., Rogachev, A., Balandin, T., Borshchevskiy, V., Popov, A., Bourenkov, G., Bamberg, E., Rodriguez-Valera, F., Büldt, G., Gordeliy, V.: Proc. Natl. Acad. Sci. USA, 117, 4131 (2020).
72) Lu, Y., Zhou, X. E., Gao, X., Wang, N., Xia, R., Xu, Z., Leng, Y., Shi, Y., Wang, G., Melcher, K., Xu, H. E., He, Y.: Cell Res., 30, 88 (2020).
73) Bulzu, P.-A., Kavagutti, V. S., Chiriac, M.-C., Vavourakis, C. D., Inoue, K., Kandori, H., Andrei, A.-S., Ghai, R., Hallam, S. J.: mSphere, 6, e00661 (2021).
74) Flores-Uribe, J., Hevroni, G., Ghai, R., Pushkarev, A., Inoue, K., Kandori, H., Béjà, O.: Environ. Microbiol. Rep., 11, 419 (2019).
75) Hososhima, S., Mizutori, R., Abe-Yoshizumi, R., Rozenberg, A., Shigemura, S., Pushkarev, A., Konno, M., Katayama, K., Inoue, K., Tsunoda, S. P., Béjà, O., Kandori, H.: eLife, 11, e78416 (2022).
76) Cho, S. G., Song, M., Chuon, K., Shim, J. G., Meas, S., Jung, K. H.: PLoS Biol., 20, e3001817 (2022).
77) Shim, J. G., Cho, S. G., Kim, S. H., Chuon, K., Meas, S., Choi, A., Jung, K. H.: Microbiol. Spectr., 10, e0221522 (2022).
78) Fleming, J. F., Feuda, R., Roberts, N. W., Pisani, D.: Genome Biol. Evol., 12, 3906 (2020).
79) Brown, P. K., Wald, G.: Nature, 200, 37 (1963).
80) Shichida, Y., Matsuyama, T.: Philos. Trans. R. Soc. Lond., Ser. B: Biol. Sci., 364, 2881 (2009).
81) Spitschan, M.: Curr. Opin. Behav. Sci., 30, 67 (2019).
82) Ozdeslik, R. N., Olinski, L. E., Trieu, M. M., Oprian, D. D., Oancea, E.: Proc. Natl. Acad. Sci. USA, 116, 11508 (2019).
83) Sato, M., Tsuji, T., Yang, K., Ren, X., Dreyfuss, J. M., Huang, T. L., Wang, C.-H., Shamsi, F., Leiria, L. O., Lynes, M. D., Yau, K.-W., Tseng, Y.-H.: PLoS Biol., 18, e3000630 (2020).
84) Hara, T., Hara, R.: Nature, 219, 450 (1968).
85) Hao, W., Fong, H. K. W.: J. Biol. Chem., 274, 6085 (1999).
86) Morimoto, N., Nagata, T., Inoue, K.: Biochemistry, 62, 1429 (2023).
87) Koyanagi, M., Takano, K., Tsukamoto, H., Ohtsu, K., Tokunaga, F., Terakita, A.: Proc. Natl. Acad. Sci. USA, 105, 15576 (2008).
88) Sato, K., Yamashita, T., Ohuchi, H., Takeuchi, A., Gotoh, H., Ono, K., Mizuno, M., Mizutani, Y., Tomonari, S., Sakai, K., mamoto, Y., Wada, A., Shichida, Y.: Nat. Commun., 9, 1255 (2018).
89) Airan, R. D., Thompson, K. R., Fenno, L. E., Bernstein, H., Deisseroth, K.: Nature, 458, 1025 (2009).
90) Koyanagi, M., Shen, B., Nagata, T., Sun, L., Wada, S., Kamimura, S., Kage-Nakadai, E., Terakita, A.: Proc. Natl. Acad. Sci. USA, 119, e2204341119 (2022).
91) Tsukamoto, H., Kubo, Y.: Proc. Natl. Acad. Sci. USA, 120, e2301269120 (2023).