【トピックス】
未培養微生物遺伝子の大規模データを活用した酵素探索
細川正人、津田宗一郎
早稲田大学 大学院先進理工学研究科/bitBiome 株式会社、bitBiome 株式会社
1.はじめに
環境破壊や資源枯渇などの社会課題の解決と経済成長を両輪で実現するものとして、「バイオものづくり」が注目されている。日本は古くから発酵・醸造の技術を有しており、バイオものづくり分野での競争力を持つと言われる。一方、バイオものづくりでは、酵素の活用・スマートセルの作製など、微生物機能を人工的に活用するためのデータの蓄積・技術の高度化も欠かすことができない。我々は、未培養微生物遺伝子を大規模に収集することのできるシングルセル解析技術を開発し、その成果を活用して創業したスタートアップbitBiome社にて、大規模微生物遺伝子データから酵素探索を行うための基盤技術を開発してきた。本稿では、これらの技術がどのように有用酵素の探索からバイオものづくりに貢献できるのか、技術の詳細と将来展望について紹介する。
2.未培養微生物の大規模遺伝子配列解析を実現する技術 – bit-MAP
我々が開発したbit-MAP® (bitBiome Microbiome Analysis Platform)は、微生物を対象としたシングルセル解析とバイオインフォマティクスを含む微生物ゲノム解析全般を統合した基盤技術である。シングルセル解析は、未培養微生物叢から、微生物を1細胞ずつより分けて個別の遺伝子配列解析を行うものである1,2)。試料中の様々な微生物の全ゲノムを個別に解析するため、微生物と遺伝子の保有関係を明らかにできる。この紐づけは、バイオものづくりへの活用において大きな強みになる。例えば、特定の細菌種が保有する酵素遺伝子を探索することや、標的遺伝子の保有種を参照して遺伝子組換え発現に最適な宿主を選択することなどが可能である。未培養微生物における遺伝子保有関係は、従来のメタゲノム解析では特定が困難なことが多く、シングルセル解析を用いて初めて明らかになることが多い3)。
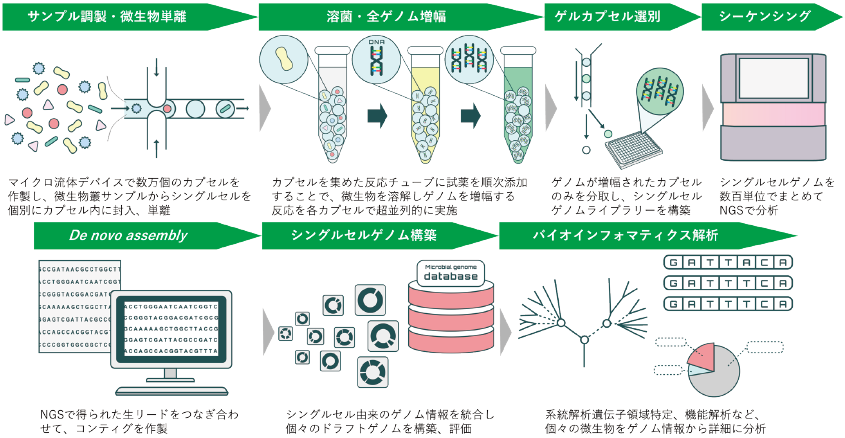
シングルセルゲノム解析のフローは図1に示した。解析実績として、共生細菌 (腸内細菌、口腔内細菌、皮膚細菌、動植物組織、昆虫など)、環境細菌 (水圏:温泉水、河川水、井戸水、海水、海泥、廃水など、土壌:圃場土、水田、土漠、その他:微生物マット、活性汚泥など)がある。
図1 微生物シングルセル解析bit-MAPによる未培養微生物遺伝子データ収集の流れ
3.酵素探索のリソースを広げる未培養微生物データベース - bit-GEM
ではbit-MAPは酵素工学にどのような貢献ができるのか?1つはこれまでに解析が困難だった微生物叢サンプルも効率的にゲノム解析が出来る、という点を挙げることができる。
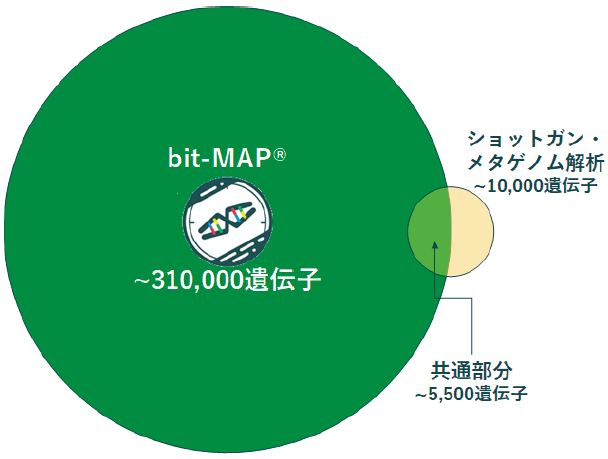
図2は土壌微生物サンプルを既存の手法 (ショットガンメタゲノム解析) と我々のシングルセル解析bit-MAPで解析した比較データである。同一サンプルを用い、かつ次世代シーケンサーの解析データ量も両者ともに40Gbpに揃えた時、600bpより長い遺伝子 (断片化した不完全な遺伝子は除くため) がどのぐらい取得できたかを数えたものであるが、ショットガンメタゲノム解析が約1万遺伝子獲得出来ているのに比べ、bit-MAPではその30倍以上の31万遺伝子が獲得されている。また、ショットガンメタゲノム解析で取得された遺伝子の50%以上はシングルセル解析においても取得できている。このことは、既存の手法が苦手とする非常に多様な微生物叢からなるサンプルからもbit-MAPは品質の高い遺伝子配列を取得できることを示している。このデータの中には、これまで誰も手にしたことのない酵素遺伝子配列が数多く含まれている。我々はbit-MAPを用い、日本中の様々な環境から採取した未培養微生物ゲノム解析を行ってきた。自社の微生物遺伝子データベースbit-GEM (bitBiome Gene Encyclopedia from Microbes)には、現在は約12億件超の遺伝子が集まっている。
図2 土壌微生物ゲノム解析による取得遺伝子数の比較
4.大規模データから目的の酵素を探索し改良する– bit-QED
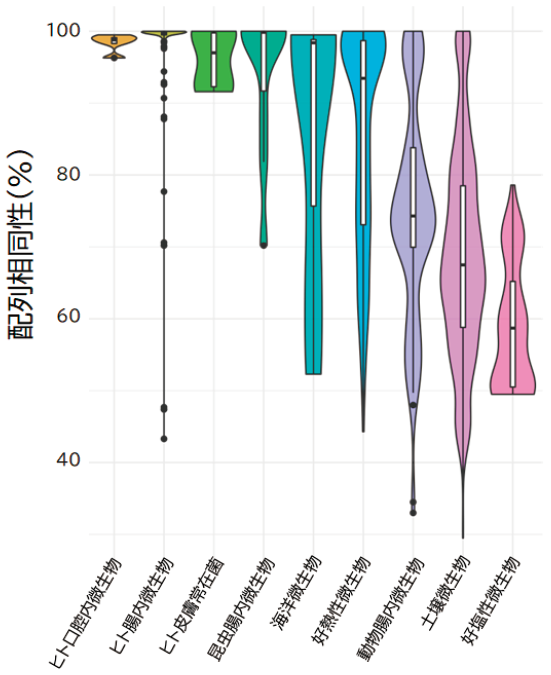
図3はある酵素をbit-GEMから探索し、公共データベースに登録されている遺伝子との配列相同性を検証したものである。口腔内や腸内などヒト由来微生物叢から取得された遺伝子は公共データベースとの高い相同性を示す一方、土壌等の環境微生物由来の遺伝子では配列相同性が低いことが見て取れる。他の様々な酵素を対象とした場合でも同様の傾向が見られる。これは、ヒト共生微生物に比べて環境微生物が多様であることを示しているだけでなく、既存のゲノム解析手法ではその配列多様性を未だ充分に捉えきれておらず、公共データベースに登録実績のない未知の遺伝子がまだまだ膨大に存在していることを示している。
図3 自社データベース由来遺伝子の公共データベース中の遺伝子との配列相同性
このようにして得られた未知の微生物ゲノム、遺伝子データはどのように活用できるだろうか。いくら大量にデータがあったとしても、必要とする遺伝子の中で機能を持つものを発見できなければ宝の持ち腐れである。BLASTなどの配列類似性ベースの探索を行うと、数万、 数十万の候補遺伝子がヒットするが、これらをすべて実験的に検証することは不可能である。酵素工学的観点からは「目的の基質に反応するのはどの酵素なのか?」が分かればよい。このため、我々は望ましい特性を持った酵素の絞り込みを行うためのin silico解析手法bit-QED(bitBiome Quality Enzyme Discovery)を開発した。
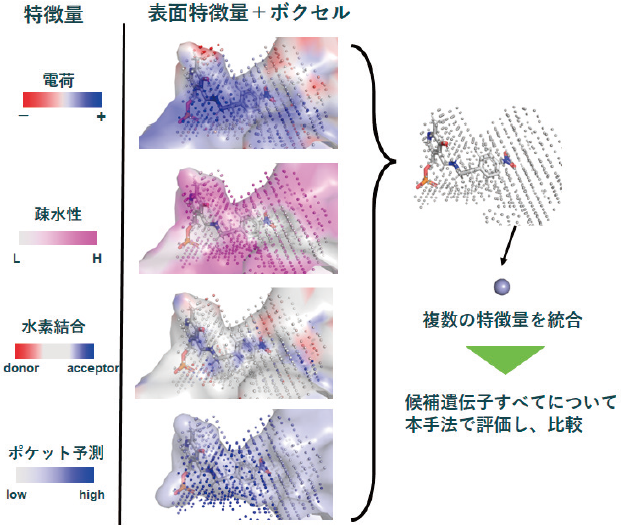
具体的には、これまでの配列ベースの探索手法に加え、近年AlphaFold2などで注目が集まっている3次元構造モデリングの手法を用いる (図4)。酵素反応の予測では、タンパク質の立体構造のうち特に活性部位の構造に注目する。活性部位のポケット構造表面を様々な特徴量を用いて定量化し、探索対象の酵素遺伝子のすべてに対してこれらの特徴量を解析し比較することで、配列情報だけでは分からない各酵素の物理化学的特徴を分類する。その後、目的の基質との反応性について、分子動力学シミュレーションを活用して絞り込む。産業ニーズが大きいトランスアミナーゼを未培養微生物遺伝子データから選抜し、bit-QEDで活性が見込まれた候補配列の活性を実験的に検証したところ、70%程度の確度で目的の活性を持つ酵素を取得できた。また、bit-QEDで得られる情報を元に、酵素配列の改変を行い、機能向上を行うことも可能である。
図4 表面特徴量を活用した酵素探索
bit-QEDは現在も機能拡張を継続し進化を続けている。タンパク質言語モデルなど最新の人工知能技術の統合、ロボティクス技術を活用した大規模な実験的検証などにより、今後さらに精緻化し、より高確度で酵素探索・絞り込みが行える技術に発展させることができると確信している。
5.おわりに
培養はおろか解析が難しい微生物は「微生物ダークマター」と呼ばれる。微生物ダークマターが環境にはまだまだ多く存在し、まだ見ぬ有用遺伝子が手つかずのまま残されている。未培養微生物ゲノム解析技術bit-MAP®、膨大な微生物酵素データベースbit-GEM、そしてin silico酵素探索・改良技術bit-QEDは、この微生物ダークマターに光を当て、微生物の潜在的な機能を解き放ち、産業応用の実現を支援する。これまでは、ゲノム解析や遺伝子合成にかかる高いコストが、こうした未培養微生物の産業応用可能性を探る上で大きなハードルとなっていた。しかし、コストは年々大幅に減少しデータ規模が増大しており、この傾向は今後も続くと予想される。その中で、唯一無二の膨大な遺伝子配列データを保有することは、微生物機能を生かしたバイオものづくりでの競争優位性を保ち、独自性の高い研究開発を実現するための基盤となると考えている。
世界中の多くの国がCO2削減、脱石油を志向し、バイオものづくりに注力しようという流れは、微生物のポテンシャルを最大限に活用しよう、という我々の目標にまさに合致するものである。微生物由来酵素そして微生物自体がこれまで以上に食品、医療、エネルギー、農業といった幅広い産業に応用されていくことを願ってやまない。
謝辞
本稿で紹介した1つであるシングルセル解析に関する研究は、早稲田大学にてスタートしたものであり、多くの共同研究者の方々にご支援頂いて進められたものである。早稲田大学の竹山春子教授をはじめ、関係する全ての皆さまにこの場を借りて心より感謝を申し上げる。また、本稿で紹介した技術開発にこれまで関わってきたbitBiome社研究開発部の全てのメンバーにも改めて感謝したい。
文献
1) Chijiiwa, R., Hosokawa, M., Kogawa, M., Nishikawa, Y., Ide, K., Sakanashi, C., Takahashi, K., Takeyama, H.: Microbiome, 8, 5 (2020).
2) Nishikawa, Y., Kogawa, M., Hosokawa, M., Wagatsuma, R., Mineta, K., Takahashi, K., Ide, K., Yura, K., Behzad, H., Gojobori, T., Takeyama, H.: ISME Commun., 2, 92 (2022).
3) 青柳秀紀: 未培養微生物研究の最新動向、シーエムシー出版 (2023).