【トピックス】
非リボソームペプチド合成酵素のエンジニアリング:アデニル化酵素の厳密な基質選択性の制御
石川文洋
近畿大・薬
1.はじめに
資源が枯渇しつつある現代においても、医薬品候補化合物として天然物の多様性をさらに拡充していくことが重要である。新たな天然物の多様性拡充戦略として生合成遺伝子や生合成酵素の応用が盛んに研究されている。それにより、微生物を宿主として生合成遺伝子の異種発現や物質生産に加えて、生合成系の反応機構が解明されてきており、生合成系の人工的な改変から天然物を超えた非天然物生合成へと研究がシフトしてきている。
ペプチド天然物の多くは、非リボソームペプチド合成酵素 (NRPS) により生合成される1)。NRPSによるペプチドの生合成では、タンパク質性アミノ酸以外に500種類以上の非タンパク質性アミノ酸、α-ケト酸、α-ヒドロキシ酸、安息香酸誘導体などが使用され、多様なペプチド骨格が構築される2)。
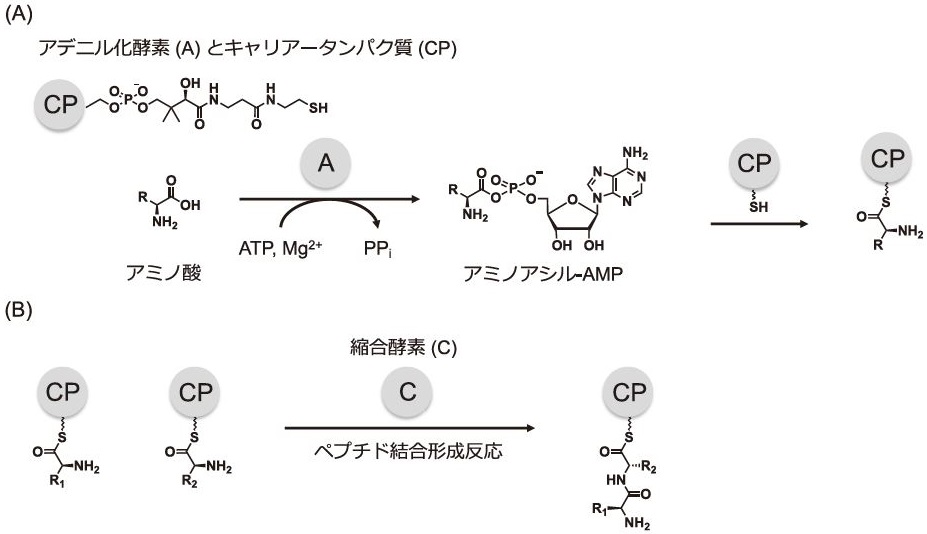
NRPSのアデニル化酵素 (Aドメイン) は、タンパク質性アミノ酸、非タンパク質性アミノ酸、安息香酸誘導体などを選択し、ATPとMg2+を利用してアミノアシル-AMPに活性化する (図1A)3)。次に、アミノアシル-AMPは、キャリアータンパク質 (CP) へチオエステル結合を介して受け渡される (図1A)3)。続いて、キャリアータンパク質に受け渡されたアミノ酸は、縮合酵素 (Cドメイン) が触媒するペプチド結合形成反応によりペプチド骨格へ導入される (図1B)3)。
図1 一般的なNRPSの触媒機構 (A) アデニル化酵素が触媒する反応 (B) 縮合酵素が触媒する反応
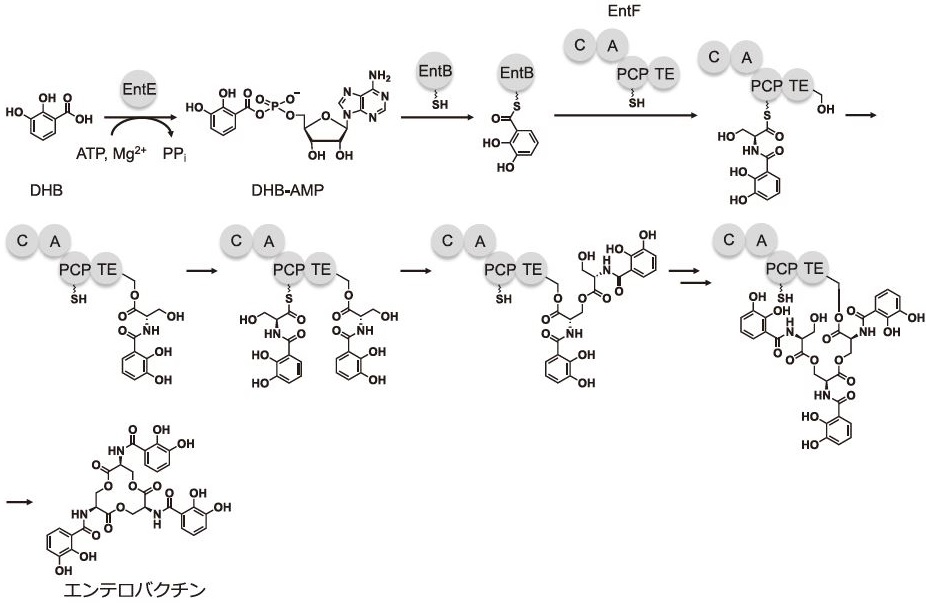
NRPS生合成システムの詳細に関して、世界的なモデル系であるエンテロバクチンの生合成を例に説明する4-6)。エンテロバクチンは、EntE、EntB、EntFにより生合成される (図2)。EntE (Aドメイン) が2,3-ジヒドロキシ安息香酸 (DHB) をアデニル化する。続いて、EntB (CPドメイン) にDHBが受け渡される。EntFはCドメイン、Aドメイン、ペプチジルキャリアータンパク質 (PCP) ドメイン、チオエステラーゼ (TE) ドメインから構成されるNRPSモジュールである。AドメインがL-Serをアデニル化後、L-SerがPCPドメインへ受け渡される。続いて、CドメインがEntBへ受け渡されたDHBチオエステルへのL-Serの一級アミンの求核攻撃を触媒し、アミド結合 (DHB-L-Ser) を形成する。その後、TEドメインが三量体化したDHB-L-Serの環化反応を触媒することで、エンテロバクチンは生合成される。
図2 NRPSによるエンテロバクチンの生合成機構
NRPSにおいてAドメインは、ペプチド骨格へ導入する基質を選択する“gatekeeper”として機能している。そのため、NRPS生合成系においてAドメインはエンジニアリングにおける標的タンパク質になっている。NRPSを利用して非天然型ペプチド骨格を生合成する手法のうち、最も汎用されるドメインスワッピング法 (Aドメインをアミノ酸選択性の異なる別のA′ドメインへと人工的に置換する) は、AドメインとPCPドメインとのタンパク質間相互作用を破壊してしまうため、望む化合物ができないことや収量が低下することが多い7-9)。その問題を回避するため、Aドメインの活性部位をエンジニアリングし、アミノ酸選択性を制御する手法 (合理的な変異導入や進化分子工学など) が多く報告されてきている10-13)。一方で、安息香酸誘導体を基質とするAドメインのエンジニアリングに関して、報告例は二例にとどまっている。また、いずれの場合もAドメインの基質特異性をDHBから2-アミノ安息香酸へ改変した例に限定されている14,15)。
筆者は、DHB選択的AドメインEntEへの合理的な変異導入により、AドメインEntEの基質特異性の改変に成功している16,17)。本稿では、AドメインEntEのエンジニアリングによる基質適応範囲拡張16)やNRPS Codeを利用するAドメインEntEのエンジニアリング17)について紹介する。
2.立体構造を基盤とするAドメインEntEのエンジニアリング17)
2-1 AドメインEntEの基質適応範囲拡張を目指した活性部位エンジニアリング
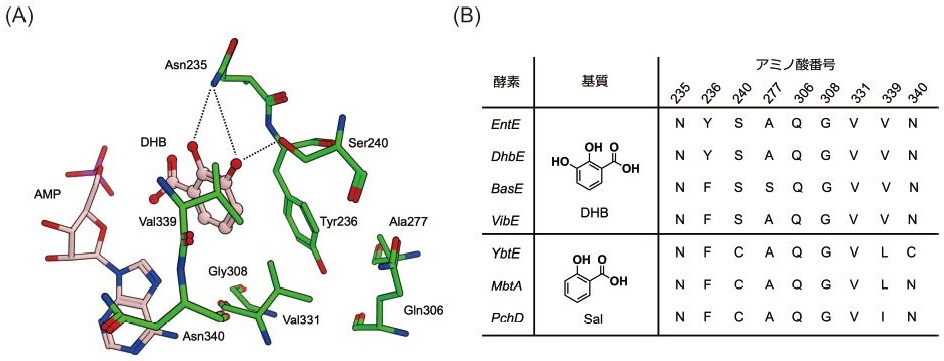
本研究では、AドメインEntEの厳密な基質特異性を緩和し、多種多様な安息香酸誘導体を基質として許容するAドメインEntE変異体の創製を検討した。まず、AドメインEntEと同様にDHBに基質特異性をもつAドメインDhbEとDHBとの複合体のX線結晶構造18)を利用して、AドメインEntE活性部位の立体構造予測を行った (図3AB)。EntEは、1) 235番目のAsnと240番目のSerと2つの水酸基との水素結合、2) 236番目のTyrとベンゼン環とのπ-πスタッキング相互作用、によりDHBを特異的に認識する。安息香酸誘導体を基質とするAドメインは、ベンゼン環部を共通の認識部位とし、水素結合などにより置換基を認識していると考えられる。そのため、AドメインEntEにおいてベンゼン環認識部位を維持し、置換基認識部位周辺を拡張できれば、多種多様な置換基や置換様式をもつ安息香酸誘導体を基質として許容する活性部位が創製できると期待した。そこで、235番目のAsnをGly、Ala、Ser、Thrに置換した変異体を作製した。続いて、DHBおよびサリチル酸 (Sal) を基質として、野生型AドメインEntEおよび4種類のAドメインEntE変異体に関して酵素速度論解析を行った。その結果、AドメインEntE N235G変異体は、その他変異体と比較してDHBおよびSalへの酵素活性を維持していることが判明した (野生型EntE: DHB kcat / Km = 1311 mM-1 min-1、Sal kcat / Km = 412 mM-1 min-1; EntE N235G: DHB kcat / Km = 318 mM-1 min-1、Sal kcat / Km = 157 mM-1 min-1)。これらの結果から、AドメインEntE N235G変異体は、DHBやSalのベンゼン環を共通の認識部位とし、置換基認識部位が拡張された活性部位をもつと考えられた。

2-2 非天然型安息香酸誘導体を基質としたAドメインEntE N235Gの機能解析
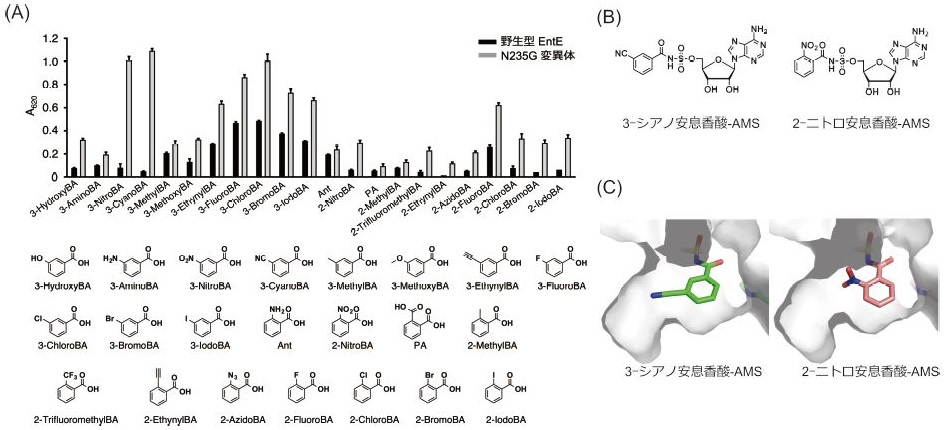
多種多様な置換基や置換様式をもつ22種類の非天然型安息香酸を基質として酵素活性の測定をマラカイトグリーン法 (マラカイトグリーン、モリブデン酸およびオルトリン酸が形成する緑色の複合体を分光光度計 (620 nm) により計測することで、ATPから遊離したリン酸を定量する方法) により行った (図4A)。その結果、AドメインEntE N235G変異体は16種類におよぶ多様多様な置換基や置換様式をもつ安息香酸誘導体を基質として許容することがわかった。また、野生型AドメインEntEおよびAドメインEntE N235G変異体の酵素反応速度論解析を行った。その結果、AドメインEntE N235G変異体は、野生型AドメインEntEと比較して、3-エチニル安息香酸、3-フルオロ安息香酸、3-クロロ安息香酸、3-ブロモ安息香酸、3-ヨード安息香酸、2-フルオロ安息香酸、2-クロロ安息香酸に対する基質特異性がそれぞれ30倍、10倍、3.8倍、14倍、72倍、8.3倍、203倍向上した。さらに、野生型AドメインEntEは5種類の安息香酸誘導体 (3-ニトロ安息香酸、3-シアノ安息香酸、2-ニトロ安息香酸、2-ブロモ安息香酸、2-ヨード安息香酸) をアデニル化することができないが、AドメインEntE N235G変異体はこれらの基質を許容することがわかった。また、MALDI-TOFMS解析によりAドメインEntE N235G変異体は、下流のCPドメインEntBへ3-エチニル安息香酸、3-フルオロ安息香酸、3-クロロ安息香酸、2-フルオロ安息香酸、2-クロロ安息香酸、2-ブロモ安息香酸、2-ヨード安息香酸、2-ニトロ安息香酸を定量的に受け渡すことが示された。一方、3-ニトロ安息香酸、3-シアノ安息香酸、3-ブロモ安息香酸では、EntBの50 - 70%にこれらの基質を受け渡すことができた。3位にニトロ基、シアノ基などの電子求引性基をもつ基質では、キャリアータンパク質のチオエステル結合が活性化され、加水分解反応を受けるため、受け渡しの効率が低下すると考えられた。

2-3 非天然型安息香酸誘導体を基質としたAドメインEntE N235Gの機能解析
最後に、AドメインEntE N235G変異体の寛容な基質特異性の構造的基盤を明らかにするため、アデニル化反応中間体アナログとのX線結晶構造解析を検討した。AドメインEntE N235G変異体の良い基質である3-ニトロ安息香酸、3-シアノ安息香酸、2-ニトロ安息香酸、2-クロロ安息香酸、2-ブロモ安息香酸とスルファモイルアデノシン (AMS) を縮合した化合物を合成した (図4B)。次に、各化合物を阻害剤として野生型AドメインEntEおよびEntE N235G変異体に対する阻害定数Kiを算出した。AドメインEntE N235G変異体に対して最も良いKi値を示した3-シアノ安息香酸-AMS (Ki = 0.18 ± 0.03µM) および2-ニトロ安息香酸-AMS (Ki = 0.050 ± 0.003µM) をリガンド分子として選択し、AドメインEntEN235G変異体との複合体のX線結晶構造解析を行った (図4BC)。その結果、AドメインEntE N235G変異体の基質結合部位は、ベンゼン環を共通の認識部位とし、2位および3位近傍に嵩高い官能基を許容する非常に大きな空間を有していることが明らかになった (図4C)。
3.NRPS codeを利用するAドメインEntEのエンジニアリング18)
3-1 NRPS codeを入れ替えた変異体の作製 (NRPS codeスワッピング)
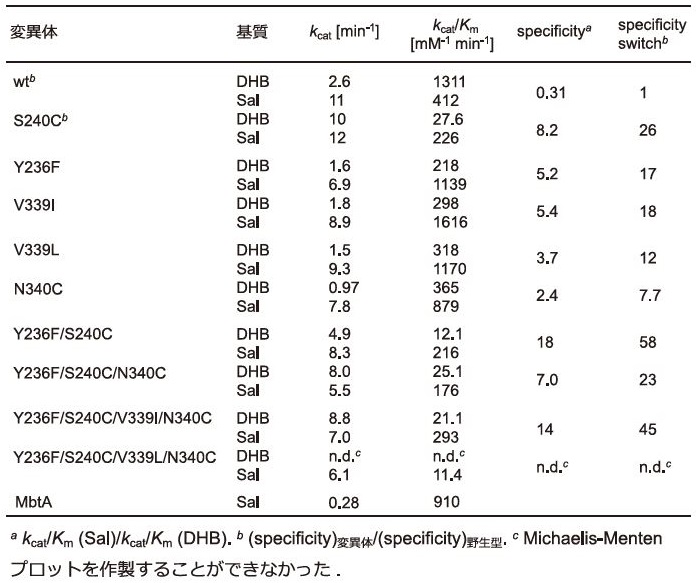
AドメインEntEのエンジニアリングにより基質適応範囲拡張は可能であった (図4A)16)。しかし、長い進化や淘汰の過程において生き残ったアデニル化酵素の基質特異性のエンジニアリングは難しいことが多い。実際、AドメインEntE N235G変異体の3-ニトロ安息香酸、3-シアノ安息香酸、2-クロロ安息香酸に対するkcat / Kmは、それぞれ12 mM-1 min-1、18 mM-1 min-1、15 mM-1 min-1であり、野生型AドメインEntEのDHBに対するkcat / Km= 1311 mM-1 min-1には遠く及ばない16)。Aドメインの構造と機能の体系的な研究により、アミノ酸選択性を決定する活性部位の9アミノ酸残基 (NRPS code) が明らかになった (図3B)19,20)。そのため、DHBに基質特異性をもつAドメインEntEのNRPS codeをSalに基質特異性をもつAドメインYbtEやPchDのNRPS codeに入れ替えることで、AドメインEntEの基質特異性を酵素活性を維持したままSalへ変換できると考えた (図3B)。そこで、AドメインEntEのNRPS codeをAドメインYbtEやPchDのNRPS codeに入れ替えた9種類のAドメインEntE変異体 (Y236F、S240C、V339I、V339L、N340C、Y236F/S240C、Y236F/S240C/N340C、Y236F/S240C/V339I/N340C、Y236F/S240C/V339L/N340C) を設計した。特に、EntE Y236F/S240C/V339L/N340C変異体はSalに基質特異性をもつAドメインYbtEのNRPS codeをAドメインEntEへ導入した変異体である。
3-2 NRPS codeを入れ替えた変異体の酵素反応速度論解析
9種類のAドメインEntE変異体を大腸菌組換えタンパク質として調製後、DHBおよびSalを基質として酵素反応速度論解析を行った (表1)。その結果、AドメインEntE Y236F/S240C/V339I/N340C 変異体 (DHB: kcat / Km = 21 mM-1 min-1; Sal: kcat / Km = 293 mM-1 min-1) およびEntE Y236F/S240C 変異体 (DHB: kcat / Km = 24 mM-1 min-1; Sal: kcat / Km = 216 mM-1 min-1) は、野生型AドメインEntE (DHB: kcat / Km = 1311 mM-1 min-1; Sal:kcat / Km = 412 mM-1 min-1) と比較して、Salに対する基質特異性がそれぞれ45倍および58倍向上したことがわかった (表1)。また、AドメインEntE Y236F/S240C/V339I/N340C変異体のSalに対する酵素活性 (kcat / Km) は、野生型AドメインEntEのSalに対する酵素活性 (kcat / Km) と比較して同程度であった (表1)。また、AドメインEntE V339I変異体 (DHB: kcat / Km = 298 mM-1 min-1; Sal: kcat / Km = 1616 mM-1 min-1) は、Salに対する基質特異性が18倍向上し、Salに対する酵素活性 (kcat / Km =1616 mM-1 min-1) は、野生型AドメインEntEのDHBに対する酵素活性と比較して同程度であった (表1)。さらに、マイコバクチンの生合成に関与するSalを基質とするAドメインMbtAの酵素活性 (Sal: kcat / Km = 913 mM-1 min-1) と比較して、1.8倍の酵素活性をもつことが判明した (表1)21)。一方、AドメインEntE Y236F/S240C/V339L/N340C変異体では、DHBおよびSalに対する酵素活性が消失した (表1)。これは、AドメインEntEY236F/S240C/N340C変異体に嵩高いV339L変異を導入することでAドメインEntE Y236F/S240C/V339L/N340C変異体の基質結合部位が狭くなり、DHBやSalが結合できなくなったためと考えられる。以上の結果から、NRPS Codeを利用することで、安息香酸誘導体に基質特異性をもつAドメインのエンジニアリングが可能であることがわかった。
表1 AドメインEntE変異体の速度論的パラメーター
4.おわりに
本稿では、非リボソームペプチド生合成においてアミノ酸や安息香酸誘導体を選択する“gatekeeper”としての役割を担っているAドメインのエンジニアリングに関する検討について紹介した。DHB選択的AドメインEntEの立体構造に基づく合理的な部位特異的変異導入によって、基質適応範囲を拡張することに成功した。AドメインEntE変異体は、天然にはない16種類におよぶ多種多様な置換様式の安息香酸誘導体を基質として許容するが、その酵素活性は野生型AドメインEntEと比較すると非常に低い。また、NRPS codeを利用することで、効率的にAドメインEntEをエンジニアリングするとともに、天然酵素を凌駕する酵素活性 (kcat / Km) の創製に成功した。しかし、NRPS codeの利用できない基質 (天然にはない基質) に対する酵素活性を創製することはできない。上述のような課題はあるが、本稿で紹介したAドメインEntE変異体を利用して酵素的に非天然型誘導体が生合成できるか検討が必要である。NRPSをエンジアニアリングするための汎用性の高い方法を開発することは難しいのが現状ではあるが、理論系の計算科学や情報科学をはじめとする多様な技術の融合を通して、NRPSのエンジニアリングの方法論を確立したいと考えている。
謝辞
本研究を遂行するにあたり、ご助言を賜りました近畿大学薬学部有機薬化学研究室田邉元三教授に感謝申し上げます。本研究を行うにあたり、東京工業大学理学院化学系江口正教授、工藤史貴准教授、宮永顕正助教 (現東京大学農学生命科学研究科准教授)、近畿大学薬学部仲西功教授、中村真也講師に多大なご助力を賜りました。この場を借りて厚く御礼申し上げます。また、JSPS科研費 (17H05438、19K05722、19H04664) と公益財団法人武田科学振興財団薬学系研究奨励の支援を受けて実施された研究です。
文献
1) Süssmuth, R. D., Mainz, A.: Angew. Chem. Int. Ed., 56, 3770 (2017).
2) Walsh, C. T., O’Brien, R. V., Kosla, C.: Angew. Chem. Int. Ed., 52, 7098 (2013).
3) Hur, G. H., Vickery, C. R., Burkart, M. D.: Nat. Prod. Rep., 29, 1074 (2012).
4) Gehring, A. M., Mori, I., Walsh, C. T.: Biochemistry, 37, 2648 (1998).
5) Sundlov, J. A., Shi, C., Wilson, D. J., Aldrich, C. C., Gulick, A. M.: Chem. Biol., 19, 188 (2012).
6) Drake, E. J., Miller, B. R., Shi, C., Tarrasch, J. T., Sundlov, J. A., Allen, C. L., SKiniotis, G., Aldrich, C. C., Gulick, A. M.: Nature, 529, 235 (2016).
7) Calcott, M. J., Owen, J. G., Lamont, I. L., Ackerley, D. F.: Appl. Environ. Microbiol., 80, 5723 (2014).
8) Yu, D., Xu, F., Zhang, S., Zhan, J.: Nat. Commun., 8, 15349 (2017).
9) Brown, A. S., Calcott, M. J., Owen, J. G., Ackery, D. F.: Nat. Prod. Rep., 35, 1210 (2018).
10) Kries, H., Wachtel, R., Pabst, A., Wanner, B., Niquille, D., Hilvert, D.: Angew. Chem. Int. Ed., 53, 10105 (2014).
11) Niquille, D. L., Hansen, D. A., Mori, T., Fercher, D., Kries, H., Hilvert, D.: Nat. Chem., 10, 282 (2018).
12) Niquille, D. L., Folger, I. B., Basler, S., Hilvert, D.: J. Am. Chem. Soc., 143, 2736 (2021).
13) Camus, A., Truong, G., Mittl, P. R. E., Markert, G., Hilvert, D.: J. Am. Chem. Soc., 144, 17567 (2022).
14) Zhang, K., Nelson, K. M., Bhuripanyo, K., Grime, K. D., Zhao, B., Aldrich, C. C., Yin, J.: Chem. Biol., 20, 92 (2013).
15) Wang, M., Zhao, H.: ACS Catal., 4, 1219 (2014).
16) Ishikawa, F., Miyanaga, A., Kitayama, H., Nakamura, S., Nakanishi, I., Kudo, F., Eguchi, T., Tanabe, G.: Angew. Chem. Int. Ed., 58, 6906 (2019).
17) Ishikawa, F., Nohara, M., Nakamura, S., Nakanishi, I., Tanabe, G.: Biochemistry, 59, 351 (2020).
18) May, J. J., Kessler, N., Marahiel, M. A., Stubbs, M. T.: Proc. Natl. Acad. Sci. USA, 99, 12120 (2002).
19) Stachelhaus, T., Mootz, H. D., Marahiel, M. A.: Chem. Biol., 6, 493 (1999).
20) Challis, G. L., Ravel, J., Townsend, C. A.: Chem. Biol., 7, 211 (2000).
21) Wilson, D. J., Aldrich, C. C.: Anal. Biochem., 404, 56 (2010).