【トピックス】
難培養微生物を可培養化する意義と可能性
青井議輝
広大院・統合生命/瀬戸内CN 国際センター
1.はじめに
環境中の微生物の多くは培養されていない、すなわち未培養である。実際にデータベース上の情報について培養されている種とされていない種 (環境から抽出したDNAに基づいた情報) を統合的に解析した場合、これまでに培養されている種は4%程度と見積もられている1)。しかしこの数値はデータベースに登録されている情報だけを基に解析した結果であり、データベースに登録されていない種 (情報) もまた膨大に存在することを想定すると未培養微生物の割合はもっと大きいと考えることもできる。また近年、培養された菌株がいっさい存在しない「門」が半数近く存在することが正確に明らかになったことで、それらを宇宙の暗黒物質になぞらえてMicrobial Dark Matter (微生物ダークマター) と魅力的なキーワードで呼称されるようにもなった2)。さらにCandidate phyla radiation (CPR)/Candidatus PatescibacteriaやDPANNという、巨大な未培養系統群が存在することも後に明らかになった (Rinkeら2)とBrownら3)によって提案されたバクテリアまたはアーキアの上門 (superphylum) で、発見当初は太古に分岐し進化系統学的に極めて独立していると考えられていたが、その部分については議論が残されている)。しかし、最近黒田らは、CPR微生物の純粋2者培養に成功し、ゲノム情報に基づいて分子系統解析を行い、CPRは全体として一つの「門」であることを示している4)。
2.資源としての可能性
未培養微生物は、膨大に眠る未利用バイオリソースとして捉えることができる。つまり、これまで培養可能な微生物から人類が得てきた恩恵を考慮すると、未培養微生物を可培養化することができれば産業応用という側面でも大きな波及効果が期待でき、例えば創薬資源の大幅拡大などが挙げられる。これまで多くの抗菌薬は天然物 (バクテリアやカビなどの微生物の代謝物) から発見されてきていることは周知の事実である。実際に、市販されている低分子医薬品は天然物 (天然物の誘導体含む)、あるいは天然物の模倣物質 (合成化合物だが薬理作用をもたらす部位の構造は天然物由来) が全体の5割以上を占めている。特に抗菌薬に限るとワクチンを除いて実に7割以上を占めている5)。しかし昨今は、天然物からの創薬スクリーニングに対しては「新規の生理活性物質の取得効率が悪い」という一般的なイメージがぬぐいきれない。なぜなら培養可能な有用微生物 (易培養性の放線菌など) の多くはすでに発見され、有用な化合物は取り尽くされたと考えられるからである。しかし逆に、従来法では培養不可能な難培養微生物を可培養化し利用可能にすることさえできれば、創薬を目的にしたスクリーニング資源としての微生物資源は魅力的かもしれない。実際に、難培養系統群として知られているVerrucomicrobiota 門、Acidobacteriota 門に属する微生物は、放線菌門と同等レベルの数の生理活性物質の生合成遺伝子クラスターを保持していることが近年判明している6)。さらに、後述するin situ培養 (i-chipという新規培養法) を通じて獲得した未培養微生物から耐性菌が原理的には出現しにくいという画期的な抗菌薬の候補物質「Teixobactin」が発見されている7)。また線虫の腸内細菌叢より分離した微生物よりグラム陰性細菌を選択的に殺菌可能な新規抗菌薬候補物質が発見されている8)。複数の抗菌薬に耐性を持つ多剤耐性菌の増大に対して新薬の開発が追いつかないという深刻な状況9)が好転する兆しとまでは言い難いが、革新的な分離培養法やスクリーニング方法が開発されれば、引き続き天然物創薬研究から新しい医薬品の誕生が期待できる。同様に新規酵素など新しい代謝活性を有する微生物を発見することも期待できると言える。
一方で、近年メタゲノム解析・バイオDXなど培養非依存的な解析アプローチの技術的な進展が著しい。培養株を得てそこから有用活性や機能を探索するというアプローチではなく、メタゲノム解析やシングルセルゲノムミクスで培養非依存的に得られた遺伝子配列情報からデータベース上に登録されている遺伝子・アミノ酸情報と照合してバイオインフォマティクスツールを駆使して機能を推定し、さらにそれを遺伝子資源として利用するというアプローチは、従来型の限界を突破できる方法論として期待されている。実際に培養非依存的に遺伝子配列ベースでスクリーニングをするハイスループット手法も開発されている10)。しかし原理的に、1) データベースの情報に立脚するため機能未知遺伝子へは対応困難であること、2) 取得した遺伝子は宿主微生物で異種発現させる必要があるが、それは容易ではないこと、などの制限が存在する。したがって「できるのであれば」培養ベースのアプローチの充実や進展も培養非依存的なアプローチ同様に必要である。また、遺伝子配列情報からでは、微生物の本質的に重要な生理・生態学的な情報は得られないことがある。しかし、培養手法におけるイノベーションは近年着実に進みつつあるものの11)進展は遅いと言わざるを得ない。一方、難培養微生物の可培養化には微生物間相互作用が重要であることが示唆されており、これは手法開発の有効な方向性と言える11-14)。ここで難培養微生物とは、従来の培養法では分離培養困難である微生物を指すが詳細は後述する。
3.難培養微生物とは何か?どうしたら培養できるのか
寒天平板上にコロニーを形成する微生物の割合 (植菌細胞数に対するコロニーの数の割合) は極めて低いという観察はコッホが寒天平板培養法を確立した直後の古い時代から報告されている15)。“The Great Plate Count Anomaly”と呼称されるなど16)、微生物学において最も大きな課題の一つと言える。さらに多くの微生物は「培養できない」と表現することは科学的に正しくはないが、寒天平板培養など従来の培養法では環境中の微生物は簡単には培養できないことは多くの微生物学者は肌感覚として感じている。このように従来法では容易に培養されない微生物のことを「難培養微生物」と本稿では呼称することとする。一方で、本稿の冒頭で述べた未培養微生物について、「存在は把握されているものの未だ分離株が得られていない (培養されていない) 微生物」と定義したが、それらの全てが従来法で培養できないことを意味してはいない。すなわち未培養微生物の多くは難培養微生物であると推察されるが、未培養微生物と難培養微生物は定義が異なることを明記しておく。
さて、多くの微生物が培養困難であることについて普遍的な要因が存在し、その理由やメカニズムを解明できれば、難培養微生物を可培養化するための有効な手段や画期的手法を導き出せると考えられる。しかし、「培養できない理由」はほとんど解明されていない。なぜなら、従来法で容易に分離される微生物を眺めていても「難培養微生物がなぜ培養できないか」という問いの答えにはたどりつかないというジレンマが存在するからである。そのジレンマを解消するためには、1) 新規分離培養手法を開発し、2) その手法を用いて従来法では培養困難な難培養微生物を獲得する、さらに3) それら難培養微生物を用いてなぜ従来法では培養できなかったのかという疑問を解消するための検証を行うというステップが合理的であると考えられる。
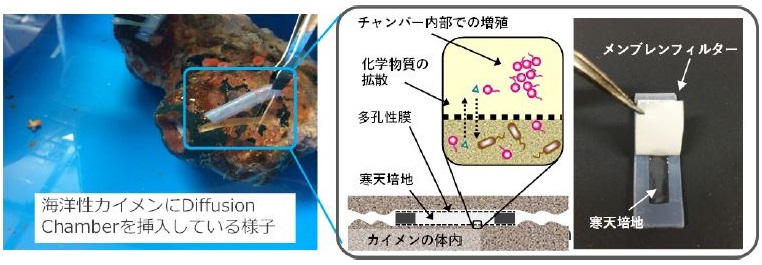
著者らは、多くの微生物が培養困難である理由の解明を目的の一つとして、海洋性カイメンから複数の培養手法 (新規培養手法および従来法) を用いて網羅的に分離株を獲得し、それらの新規性 (既存種に対する16S rRNA遺伝子配列の相同性) や生理学的性質を培養手法ごとに比較した。その際、新規培養手法としてin situ培養 (図1) および連続集積培養法を用いた。ここでin situ培養とは、孔径0.1µm以下の膜 (メンブレンフィルターなど) によって構成された培養チャンバーを用い、環境中の微生物を植菌して原位置 (実環境中) に投入し培養する培養法である。増殖に必要な未知因子が実際の環境中には存在するためそれらが供給され、難培養微生物が可培養化されるという期待に基づいている。これまでにいくつかの方法が提案されており、例えば1) 寒天培地の中に微生物を植菌しその両側をメンブレンフィルターで包むタイプの「Diffusion Chamber」17)、2) 多数の独立した小さなdiffusion chamberで構成される「iChip」18)、3) 中空糸膜をチャンバーとして用いる「Hollow Fiber Membrane Chamber (HFMC)」19)などが挙げられる。これまでにin situ培養は従来法と比較して多様な未培養微生物が獲得可能であることが判明している20,21)。しかし未培養・難培養微生物の可培養化に寄与する本質的に重要な因子は十分に明らかになっていなかった。連続集積培養法とは、環境サンプル (この場合は海洋性カイメンをすりつぶしたもの) を植菌源として何らかの担体に付着固定化させ、培地をリアクター内に連続的に供給して比較的長期間培養する方法である22,23)。基質負荷と消費速度のバランスをコントロールすることでリアクター内部の基質濃度を低く一定に保つことができる。一般的に微生物の性質として基質親和性と最大比増殖速度は反比例するため、リアクター内を低濃度に維持することができれば、リアクター内部では基質親和性が高く増殖速度の遅い微生物 (k-strategist) を優占化させることが期待できる。ここでk-strategistとは、基質親和性が高い (最大比増殖速度は低い) という性質に特化することで、基質濃度が低い場合においてはその条件下における比増殖速度を競合他者より高く保ち、同じ基質を競合する他者に対して優位に立つ戦略をとる微生物のことを指す。

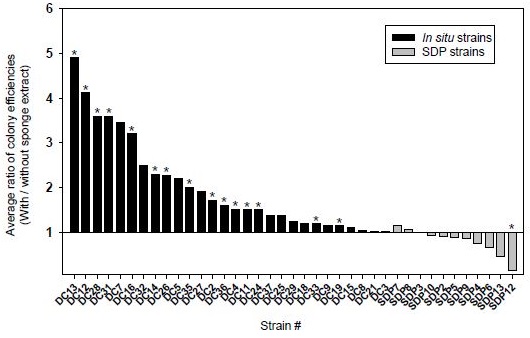
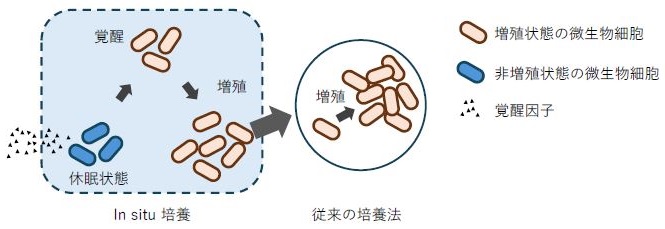
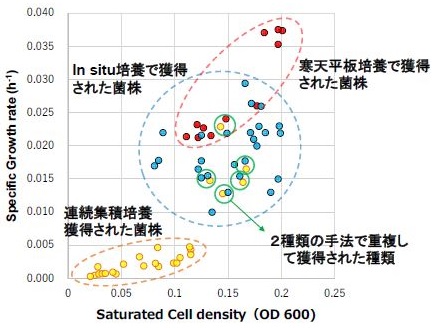
上記の新規培養手法および従来法 (寒天平板培養法) で得られた菌株について手法ごとに比較した結果、1) それぞれの手法ごとに獲得される種類が全く異なること、2) 新規分離培養手法を用いた場合、新規性の高い未培養微生物の獲得効率が格段に高まることが判明した13,23)。具体的には、例えばin situ培養で得られた菌株と従来法で得られた菌株をそれぞれ60株程度比較したところ、両手法で重複して検出された種類は1種だけであった。さらに得られた菌株の16S rRNA遺伝子配列を既存種のそれと比較したところ、in situ培養で得られた菌株の約半分程度は新種相当である一方で、従来法で得られた菌株のほぼ全ては既存種であることが判明した13)。さらに、in situ培養で得られた菌株は海洋性カイメンの抽出液に含まれる成分によって増殖の開始が誘導される (抽出液の添加は菌株の比増殖速度には影響しない)、という結果が得られた (図2)13)。この結果は、難培養微生物を可培養化するためには「外部環境に存在するなんらかの因子によって休眠状態の微生物の増殖の開始を誘導させる」必要があることを示唆している (図3)。外部環境から供給される化合物の多くまたは少なくとも一部は微生物によって生産されると考えられるため、多くの未培養微生物は微生物間相互作用によって増殖が制御されるという可能性がここから見出される。一方で、連続集積培養を通じて得られた菌株は、従来法およびin situ培養で得られた菌株と比較して、最高到達菌体密度および比増殖速度が低く (図4)、コロニーサイズが小さいことなどが判明した23)。これらの結果から、連続集積培養由来の菌株は、増殖速度が低いだけでなく自身の代謝産物または代謝副産物に感受性が高いことが示唆された。このように低菌体密度でないと増殖できないタイプはやはり従来法では分離培養困難 (未培養) であると考えられる。さらに著者らは、平板上においてコロニーを形成できない菌株を選択的に分離するスクリーニング手法を開発したところ、極めて新規性の高い未培養微生物の分離培養に成功し、連続集積培養で得られた菌株と同様に自身の代謝産物に高い感受性を持つ微生物が獲得されることを明らかにしている24)。



以上の結果を踏まえると、微生物の難培養性にはある一定の普遍的要因が存在していることが示唆される。すなわち、1) 環境中の微生物の多くは休眠状態 (増殖不能状態) であるが、栄養源の供給だけでは増殖を開始できない、2) 自身の代謝産物に高い感受性を持つために従来法では検出可能レベルまで増殖することが困難 (または時間がかかる)、といった性質を持つ微生物の多くは従来法では培養困難であり、上記二つの性質を併せ持つ微生物は、極めて培養することが難しいと推察できる。
4.おわりに
未培養・難培養微生物の存在は微生物学の黎明期から観察されてきた未解決な重要課題である。一方で、培養非依存的なアプローチの進展によって一見その課題は解決できるかのように感じられるかもしれない。しかし、「微生物を培養する」ことは学術面そして応用面において引き続き意義が高い。また、微生物の難培養性について「個々の微生物種の適合性」だけではなく、普遍的に環境微生物の難培養性をコントロールするメカニズムが存在することも示唆されるため、微生物の「難培養性」について本質的な理解を得ることは、増殖という微生物の活動の本来根幹となる部分を理解することに直結する。つまり、分離培養手法の革新や培養可能な微生物の拡充、そして環境微生物の「難培養性」を解明することにより、直接利用できるバイオリソースの大幅な拡大が見込まれることも期待できる。
謝辞
本稿にて紹介した成果は、科研費 (22H04887、20H05587、20K21279、19H02873、18K19181、26709063) の支援を受けて実施したものです。
文献
1) Steen, A. D., Crits-Christoph, A., Carini, P., DeAngelis, K. M., Fierer, N., Lloyd, K. G., Cameron Thrash, J.: ISME J., 13, 3126 (2019).
2) Rinke, C., Schwientek, P., Sczyrba, A., Ivanova, N. N., Anderson, I. J., Cheng, J.-F., Darling, A., Malfatti, S., Swan, B. K., Gies, E.A., Dodsworth, J.A., Hedlund, Tsiamis, G., Sievert, S.M., Liu, W.-T., Eisen, J.A., Hallam, S.J., Kyrpides, N.C., Stepanauskas, R., Rubin, E. M., Hugenholtz, P., Woyke, T.: Nature, 499, 431 (2013).
3) Brown, C. T., Hug, L. A., Thomas, B. C., Sharon, I., Castelle, C. J., Singh, A., Wilkins, M. J., Wrighton, K. C., Williams K. H., Banfield, J. F.: Nature, 523, 208 (2015).
4) Nakajima, M., Nakai , R., Hirakata , Y., Kubota, K. , Satoh, H. , Nobu, M. K. , Narihiro, T., Kuroda, K.: Int. J. Syst. Evol. Microbiol., 75, 006668 (2025).
5) Newman, D. J, Cragg, G. M.: J. Nat Prod., 83, 770 (2020).
6) Crits-Christoph, A., Diamond, S., Butterfield, C. N., Thomas, B. C., Banfield, J. F.: Nature, 558, 440 (2018).
7) Ling, L. L., Schneider, T., Peoples, A. J., Spoering, A. L., Engels, I., Conlon, B. P., Mueller, A., Schäberle, T. F., Hughes, D. E., Epstein, S., Jones, M., Lazarides, L., Steadman V. A., Cohen, D. R., Felix, C. R., Fetterman K. A., Millett, W. P., Nitti, A. G., Zullo, A. M., Chen, C., Lewis, K.: Nature, 517, 455 (2015).
8) Imai, Y., Meyer, K. J., Iinishi, A., Favre-Godal, Q., Green, R., Manuse, S., Caboni, M., Mori, M., Niles, S., Ghiglieri, M., Honrao, C., Ma, X., Guo, J., Makriyannis, A., Linares-Otoya, L., Böhringer, N., Wuisan, Z.G., Kaur, H., Wu, R., Mateus, A., Typas, A., Savitski, M. M., Espinoza, J. L., O’Rourke, A., Nelson, K. E., Hiller, S., Noinaj, N., Schäberle, T. F., D’Onofrio, A., Lewis, K.: Nature , 576, 459 (2019).
9) WHO news release: https://www.who.int/news/item/14-06-2024-who-releases-report-on-state-ofdevelopment-of-antibacterials (2024).
10) Hosokawa, M., Hoshino, Y., Nishikawa, Y., Hirose, T., Yoon, D. H., Mori, T., Sekiguchi, T., Shoji, S., Takeyama, H.: Biosens. Bioelectron., 67, 379 (2015).
11) Lewis, W. H., Tahon, G., Geesink, P., Sousa, D. Z., Ettema, T. J. G.: Nat. Rev. Microbiol., 19, 225 (2021).
12) Epstein, S.: Nature, 457, 1083 (2009).
13) Jung, D., Machida, K., Nakao, Y., Kindaichi, T., Ohashi, A., Aoi, Y.: Front. Microbiol., 12, 537194 (2021).
14) 鈴木陸太, 青井議輝: 日本微生物生態学会誌, 39, 3 (2024).
15) Winterberg, H.: Zeitschrift für Hygiene und Infektionskrankheiten, 29, 75 (1898).
16) Staley, J. T., Konopka, A.: Annu. Rev. Microbiol., 39, 321 (1985).
17) Kaeberlein, T., Lewis, K., Epstein, S. S.: Science, 296, 1127 (2002).
18) Berdy, B., Spoering, A. L., Ling, L. L., Epstein, S. S.: Nat. Protoc., 12, 2232 (2017).
19) Aoi, Y., Kinoshita, T., Hata, T., Ohta, H., Obokata, H., Tsuneda, S.: Appl. Environ. Microbiol., 75, 3826 (2009).
20) Nichols D., Cahoon N., Trakhtenberg E. M., Pham L., Mehta A., Belanger A., Kanigan T., Lewis K., Epstein S. S.: Appl. Environ. Microbiol., 76, 2445 (2010).
21) Jung, D., Liu, L., He, S.: Mar. Life Sci. Technol., 3, 148 (2021).
22) Fujitani, H., Aoi, Y., Tsuneda, S.: Microb. Environ., 28, 236 (2013).
23) Jung, D., Machida, K., Nakao, Y., Owen, J. S., He, S., Kindaichi, T., Ohashi, A., Aoi, Y.: Front. Mar. Sci., 9, 963277 (2022).
24) Seo, E-Y., Jung, D., Epstein, S. S., Zhang, W., Owen, J. S., Baba, H., Yamamoto, A., Harada, M., Nakashimada, Y., Kato, S., Aoi, Y., He, S.: Front. Microbiol., 14, 1194466 (2023).