【トピックス】
β-1,2- グルカンというユニークな領域からの新規な糖質関連酵素の探索
中島将博、元内 省
東京理科大・創域理工、東京理科大院・創域理工
1. はじめに
―糖鎖構造と糖鎖に作用する酵素の多様性―
糖鎖は、核酸やタンパク質とならんで生命3鎖ともよばれる生命現象に重要な鎖状の高分子化合物の一つであり、エネルギー源となるだけでなく、細胞骨格、貯蔵多糖、感染や免疫といった生物間相互作用に関わる重要な生理機能を有している。このように様々な役割を果たせるのは、糖鎖が多様で複雑な化学構造を生み出せるからである。その主要因は、糖鎖を構成する単糖どうしの結合のパターンが複数あること、枝分かれ構造がごく普通に存在することである。あくまで理論値であるが、単糖6個の結合の組み合わせは1兆以上とする論文もあり1)、この数は、同じ6個のヌクレオチドやアミノ酸の場合の単純な組み合わせの数と比べて圧倒的に大きい。したがって、まだ日の目を見ない糖鎖が多数埋もれているのでは、と筆者らは考えている。
糖鎖の合成や分解は生物においては酵素がそのほとんどを担っている。糖鎖構造の多様性に対応して酵素の側も様々な糖鎖に作用できるよう複雑な分子進化を遂げてきた。糖鎖に作用する酵素群はCAZy database (https://www.cazy.org/Home.html)2)において主にアミノ酸配列の類似性を基にファミリーに分類されている。その中でも糖質加水分解酵素(glycoside hydrolase, GH)では現在180以上ものファミリーが見いだされ、非常に多様性に富んでいる。また、この酵素群は立体構造の「見本市」ともよばれており、様々なドメイン構成の構造 (全体的な見た目の大きく異なる構造) を含んでいるのも大きな特徴の一つである3,4)。
糖鎖の有用機能性の探索や利用のためには実用的な調製法が必要であり、糖鎖の場合は主に酵素法が採用されている。筆者らは主にGH酵素群を研究しているが、この酵素群は分解酵素だけでなく、糖転移酵素などの糖鎖の合成に使用できる酵素も多数含まれている。そのため、応用の可能性のある酵素の探索の対象としてGH酵素群は有望である。しかし、新規な糖鎖の合成にはその糖鎖に作用する酵素が不可欠であるにもかかわらず、その酵素の探索のためには目的とする糖鎖が結局は必要となり、堂々巡りの状況とも言える。新たな糖鎖やその関連酵素を探索するという方向の研究にはそういった難しさがある。
2.β-1,2-グルカンとβ-1,2-グルカン関連酵素
2-1 β-1,2-グルカン
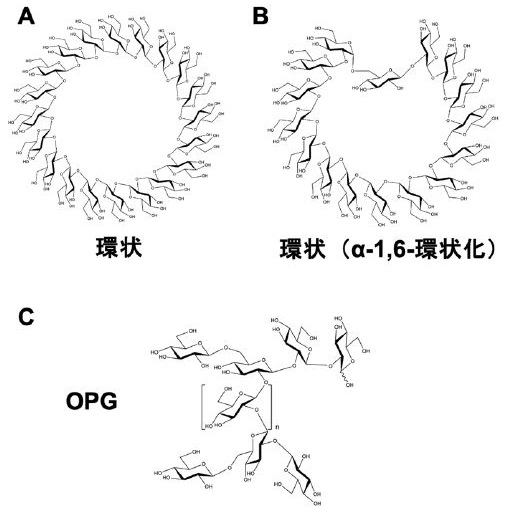
グルコースがβ-1,2-結合で連結したβ-1,2-グルカンは筆者らのグループで主に扱っている天然の糖鎖である。自然界での存在量としては希少なため聞き馴染みがないかもしれないが、セルロース (β-1,4-結合) や澱粉 (主にα-1,4-結合) と同じくグルコースポリマーである。最も主要な単糖のグルコースで構成される糖鎖ですらよく知らないものがある、ということは糖鎖の世界の奥深さの一端と言えるであろう。β-1,2-グルカンは主に環状糖として見いだされる糖鎖であり (図1A, 1B)、細菌において宿主への感染や宿主との共生のための因子、浸透圧調節物質などとしての役割が報告されている5)。結合の一つがα-1,6-結合となった環状糖は重合度13, 16, 18のものが微生物より見いだされており、重合度16の環状糖は植物への感染に必須とされるが、他の二つの生理的な役割については知られていない5) (図1B)。また、大腸菌などのグラム陰性細菌がペリプラズムに合成する側鎖を有する重合度5–12程度の直鎖状β-1,2-グルカンはosmoregulated periplasmic glucans (OPG)として知られており (図1C)、合成に関与する遺伝子の欠失は病原性の喪失、抗生物質抵抗性の上昇、運動性の低下などをもたらすことが報告されている5,6)。

2-2 β-1,2-グルカン関連酵素
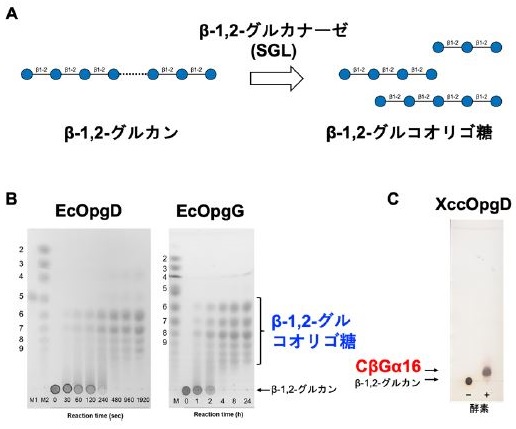
β-1,2-グルカンに作用する酵素としては、環状β-1,2-グルカン合成酵素が同定されていたのみであり、分解酵素については遺伝子同定されたものは皆無であった。2014年に筆者らのグループは、直鎖状β-1,2-グルカンに作用する反応が可逆的な酵素をGH酵素群から発見した。これを契機に、筆者らはこの酵素を用いたβ-1,2-グルカンの大量合成法も確立した7,8)。さらに、そのβ-1,2-グルカンを炭素源として微生物を培養し、細菌と糸状菌のそれぞれからβ-1,2-グルカンをエンド型で分解してβ-1,2-グルコオリゴ糖を生成するβ-1,2-グルカナーゼ (SGL) (図2A) を同定した。これらは系統的に新規であったため、これらのSGLの各ホモログ群に対して新規のファミリー (それぞれGH144とGH162) が創設された9,10)。この発見をベースにβ-1,2-グルカン関連の新規酵素がさらに見いだされている11)。以降では、最新のβ-1,2-グルカン関連の新規酵素の発見について紹介する。

3.大腸菌由来SGLの発見とその反応機構
3-1 系統的に新規な大腸菌由来SGL
大腸菌は低浸透圧条件においてペリプラズムにOPGを合成する5)。このOPGの合成には膜タンパク質であるOpgH、OpgGとOpgD (この2つはパラログ) が関与すると遺伝学的には推察されている。OpgHはUDP-グルコースを基質としてペリプラズムに直鎖状β-1,2-グルカンを合成し、OpgGとOpgDはOPGの最終的な糖鎖骨格 (図1C) への変換に関与するとされている5)。しかし、特にOpgGとOpgDの機能に関しては生化学的な証明はなかった。
筆者らが組換えOpgG、OpgD (以降それぞれEcOpgG、EcOpgD) を作成し、直鎖状β-1,2-グルカンに作用させたところ、いずれもβ-1,2-グルコオリゴ糖の生成が明確に検出され、他の多糖には作用しなかった。この結果から、これらはSGLであることが示された (図2B)12)。これらは上記のGH144やGH162のSGLを含む全てのGHファミリーとアミノ酸配列の相同性を示さない。そこで、EcOpgG、EcOpgDのホモログ群のための新たなファミリー(GH186)が創設された。GH186は近年創設されたGHファミリーの中では最大級であり、「大腸菌のようなモデル生物からでも新しいファミリーが見つかることを思い出させてくれる」とCAZy databaseでは紹介された。興味深いことに、EcOpgDとEcOpgGには活性に大きな差があり、前者は一般的なGH酵素といえる範囲であったが、後者は前者の100倍程度低い活性しか示さなかった。この理由については3-2項で説明する。
3-2 EcOpgGとEcOpgDのユニークな反応機構
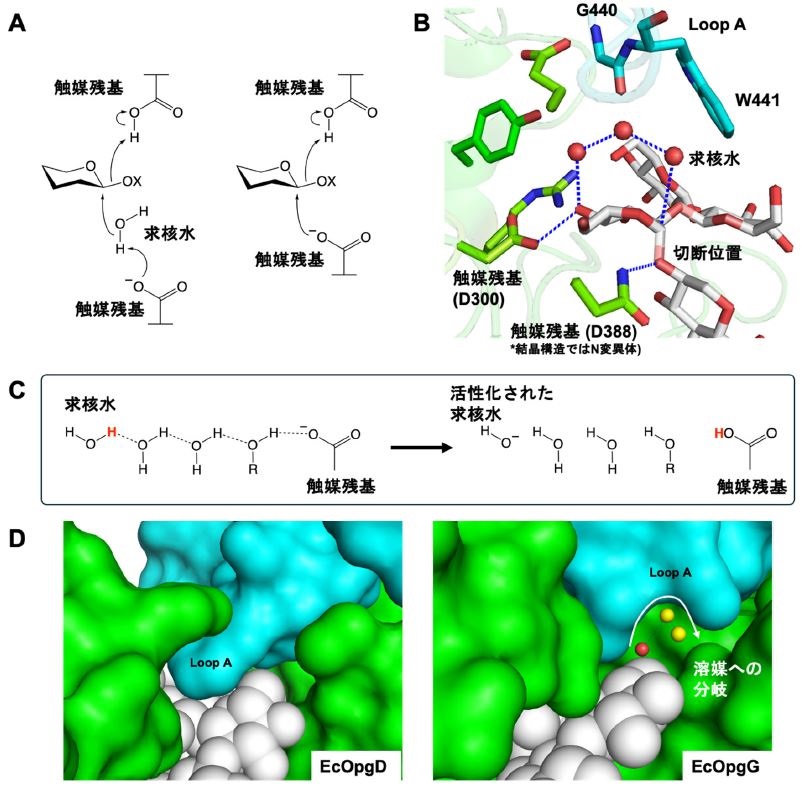
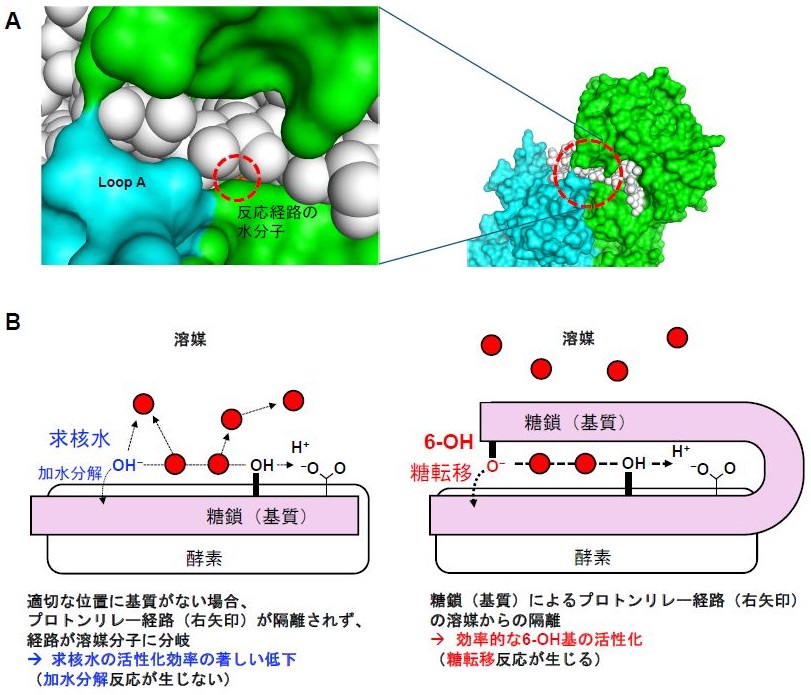
EcOpgG、EcOpgDとβ-1,2-グルカンとの複合体構造を解いたところ、反応機構 (経路) 自体とその反応機構を実現するための構造的な仕組みの2点に大きな特徴が見いだされた12)。前者に関しては、一般的にGH酵素群では2個の酸性アミノ酸残基が触媒残基となっており、それらが基質または求核水 (基質に直接作用する水分子) に直接相互作用することで触媒反応が生じる (図3A)。しかし、EcOpgDでは求核水側でそのような残基が見当たらず、求核水からさらに2個の水分子と基質のヒドロキシ基を介して求核水からプロトンを引き抜くという長い反応経路を有することが示唆された (図3BC)。水分子を3個介する例は初めてであり、GH酵素に新たな反応機構のパターンが加わったといえる。

後者に関しては、水分子は溶媒中に多量に存在する分子であり、単純に活性部位周辺に水分子を配するだけでは、プロトン授受経路は溶媒中に発散して酵素触媒としては成り立たない。そのため、水分子をプロトン授受経路として用いるためには、必要な水分子のみを適切な位置で溶媒から隔離する必要がある。EcOpgDにおいては、二量体のうち基質ポケットとなるサブユニットとは別のサブユニットから基質ポケットへと伸びているループ (以降Loop A) がこの隔離の役割を果たしている (図3D左)。特にW441とG440はこの隔離において中心的な役割を担う残基である (図3B)。これらの残基はEcOpgDに近いホモログでは高度に保存されている。一方で、EcOpgGでは基本的な反応経路はEcOpgDと同じであったものの、このLoop Aの形状がEcOpgDとは大きく異なっている。求核水の隣の水分子から溶媒に通じる経路が存在し、溶媒からの水分子の隔離が不完全である (図3D右)。これが、EcOpgGがEcOpgDよりも顕著に低い活性を示す構造的要因であり、Loop Aによる水分子の隔離の重要性を示していると言える。
4. 環状糖を合成する植物病原菌由来の新規酵素の機能と構造
4-1 Xanthomonas属細菌の新規環状糖合成酵素の発見
Loop Aは直接触媒反応に関わってはいないが、反応経路の溶媒からの隔離という点で極めて重要な領域であることから、GH186全体におけるEcOpgDのLoop Aの保存性を調べた。驚いたことに、これが保持されているのは系統的にEcOpgDの近傍のごく一部のグループだけであり、GH186ホモログの大半は全く異なるLoop Aの配列を有しており、Loop A自体が欠失したグループも存在していた。そのため、GH186には加水分解とは異なる反応を触媒する酵素が存在することが期待された。大腸菌の有するOPGとは構造が異なるβ-1,2-グルカンを有する菌としては、アブラナ科の植物の病原菌として知られているXanthomonas campestris pv. campestris (Xcc) が環状β-1,2-グルカンのうちの一つのβ-1,2-結合がα-1,6-結合に入れ替わったα-1,6-環状化β-1,2-グルカン (CβGα) を合成することが知られている5)。Xccが合成するのは重合度16のCβGα (CβGα16) であり (図1B)、この糖鎖を合成できないXccの変異株はモデル植物であるシロイヌナズナやタバコへの感染能を失うことから、この糖鎖が宿主への感染に必須であるとされている13)。しかし、この糖鎖を合成する酵素は未同定であったため、Xcc由来のGH186ホモログ(XccOpgD)を解析のターゲットとした。
XccOpgDを直鎖状β-1,2-グルカンに作用させたところ、重合度特異的な糖鎖が生成した (図2C)。ESI-MSやNMRによる生成物の解析からこの生成物がCβGα16と同定された14)。これにより、XccOpgDが基質のβ-1,2-結合を切断して、α-1,6-結合で繋ぎかえることにより分子内での糖転移反応を触媒する酵素であることが示された。この反応はβ-結合がα-結合に変わるアノマー反転型の糖転移反応で、GH酵素群の中ではユニークな反応であり、4-2項にそれを述べる。
4-2 XccOpgDのアノマー反転型糖転移反応
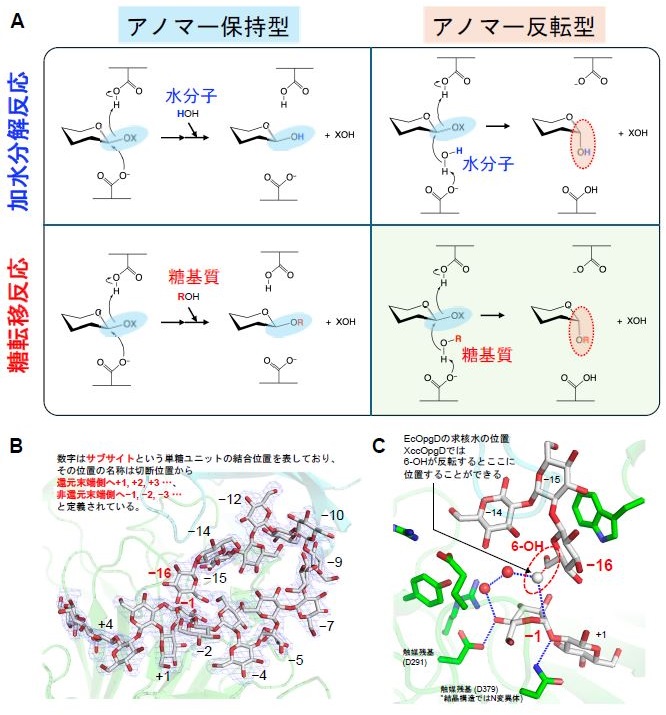
まず、前提となるGH酵素群の反応機構の分類について説明する。GH酵素群は基本的に2種類の分類方法により4種類に分類される (図4A)。一つは加水分解と転移反応による分類である。加水分解反応では水分子が基質に求核攻撃するステップがあるが、この水分子が糖のヒドロキシ基に置き換わると糖転移反応となる。反応機構におけるこれ以外の段階は加水分解反応と糖転移反応では共通しているため、加水分解反応と糖転移反応の両方を行う酵素は珍しくない。もう一つの分類は、基質と生成物で切断位置のアノマーが保持されるか、反転するかである (それぞれの反応機構はアノマー保持型、アノマー反転型とよばれる)。GH酵素群における反応ステップはSN2反応、すなわちアノマーが反転する反応であり、アノマー反転型の酵素ではこの反応ステップが1回で完了する。一方で、アノマー保持型の場合、アノマーの反転が2回生じることによって、基質と生成物の切断位置のアノマーの向きが同じになる。したがって、XccOpgDの反応はアノマー反転型の糖転移反応であると言える。

不思議なことに糖転移反応はほぼ全てがアノマー保持型の酵素で見いだされ、アノマー反転型の糖転移酵素とされているのは、例外的にGH91のDFA III合成酵素 (イヌリンから環状化したフルクト二糖を生成する酵素) のみである15)。ただし、これは基質と生成物のアノマーからそのように見えるにすぎず、反応メカニズムとしてアノマー反転型の反応が生じているのかは立体構造の面から証明されていない。
XccOpgDとβ-1,2-グルカンとの複合体構造の取得を行ったところ、非還元末端から16個のグルコースユニットで環状になり得る結合様式が観察された (図4B)14)。非還元末端のグルコースユニット (サブサイト −16、図4B) の6位ヒドロキシ基は求核基が基質に作用する位置には観察されなかったが、反転すればその位置に来ることが可能である (図4C)。これは、XccOpgDの酵素反応によってα-1,6-結合が形成されることが立体構造的に示唆されたことを意味している。したがって、XccOpgDの機能構造解析により、アノマー反転型の糖転移反応の存在が初めて明確に証明されたといえる。
4-3 GH186ファミリーにおける機能の多様性
XccOpgDではEcOpgDと基本的な反応経路は保存されており、違いはEcOpgDにおける求核水がグルコースユニットの6位ヒドロキシ基に替わったことだけである12,14)。しかし、XccOpgDは短いLoop Aしか有しておらず、このLoop Aが反応経路の水分子を溶媒から隔離することは不可能である。しかし、XccOpgDでは基質分子自体が代わりにその隔離の役割を担っている (図5A)。したがって、反応が生じるグルコースユニット (サブサイト −16) に基質が存在しない場合、反応に関わる水分子は溶媒から隔離されないため、加水分解反応が生じない (図5B左)。基質がその場所に配置された時のみそれらの水分子が溶媒から隔離されることとなる (図5B右)。これが、XccOpgDが加水分解反応を生じずに糖転移反応のみを触媒することができる構造的要因であり、EcOpgDとの明確な反応機構の違いを生んでいる。

XccOpgDによる生成物の重合度特異性については、EcOpgDにはみられないサブサイト−8から−16までのグルコースユニットの認識残基が重要な役割を果たすと考えられる。これらの残基は、変異体の活性が野生型と比べて顕著に低下することから基質認識に非常に重要である。しかし、これらの残基もGH186全体では保存性が低く、GH186ファミリーにおいて反応機構も含め機能が多様化していることが期待される。
5. おわりに
ー β-1,2-グルカン関連の2つのGHファミリーの比較を通してー
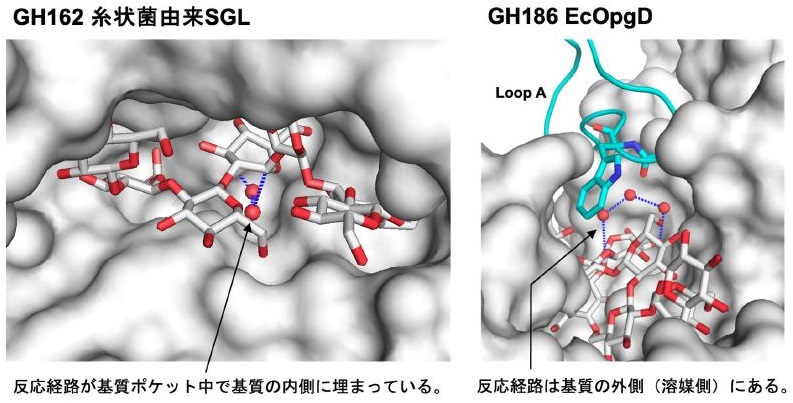
本稿で紹介したGH186酵素は同じくβ-1,2-グルカンに作用するGH144やGH162の酵素とは立体構造が全く異なり、基質の結合様式も大きく異なっている9,10,12,14)。反応機構が解明されている糸状菌由来GH162 SGLでは、結合した基質の内側 (基質ポケット側) がプロトン授受の反応経路となっている (図6左)。一方で、GH186 XccOpgDでは基質の外側 (溶媒側) が反応経路となっている。GH162の場合、基質ポケット形状がドメインを構成する二次構造にかなりの程度依存するため、分子進化による機能的な変化が生じづらいと推測される。一方でGH186の場合、基質ポケットの外側にあるLoop Aが機能に重要であり (図6右)、この限られた領域の違いによって求核種が水から糖に変わる大きな機能の違いが生じている。

2024年にタンパク質の構造予測や人工的なタンパク質の設計についての成果に対してノーベル化学賞が授与されたように、タンパク質構造の予測精度は近年急激に上昇した。人工タンパク質の設計はまだ高度な専門性が必要であるが、多くの研究者が新たな糖鎖を合成できる人工的な酵素の設計を行う時代は遠からず訪れるのではと期待している。その際、GH186のような構造は基質特異性や反応機構についての機能の多様性を生み出しやすいプラットフォームだと言えるだろう。
謝辞
本稿で紹介した筆者らの研究の一部は、日本学術振興会科学研究費助成事業 (グラント番号JP24KJ2021) の支援を受けて行われた。筆者の研究グループの研究に携わっていただいた多くの研究者、学生のみなさまに感謝申し上げる。
文献
1) Laine, R. A.: Glycobiology, 4, 759 (1994).
2) Drula, E., Garron, M. L., Dogan, S., Lombard, V., Henrissat, B., Terrapon, N.: Nucleic Acids Res., 50, 571 (2022).
3) Davies, G. J., Gloster, T. M., Henrissat, B.: Curr. Opin. Struct. Biol., 15, 637 (2005).
4) 伏信進矢: 化学と生物, 53, 45 (2015).
5) Bontemps-Gallo, S., Bohin, J. P., Lacroix, J. M.: EcoSal Plus, 7, 1 (2017).
6) Murakami, K., Nasu, H., Fujiwara, T., Takatsu, N., Yoshida, N., Furuta, K., Kaito, C.: J. Bacteriol, 203, e0051520 (2021).
7) Nakajima, M., Toyoizumi, H., Abe, K., Nakai, H., Taguchi, H., Kitaoka, M.: PLoS One, 9, e92353 (2014).
8) Abe, K., Nakajima, M., Kitaoka, M., Toyoizumi, H., Takahashi, Y., Sugimoto, N., Nakai, H., Taguchi, H.: J. Appl. Glycosci., 62, 47 (2015).
9) Abe, K., Nakajima, M., Yamashita, T., Matsunaga, H., Kamisuki, S., Nihira, T., Takahashi, Y., Sugimoto, N., Miyanaga, A., Nakai, H., Arakawa, T., Fushinobu, S., Taguchi, H.: J. Biol. Chem., 292, 7487 (2017).
10) Tanaka, N., Nakajima, M., Narukawa-Nara, M., Matsunaga, H., Kamisuki, S., Aramasa, H., Takahashi, Y., Sugimoto, N., Abe, K., Terada, T., Miyanaga, A., Yamashita, T., Sugawara, F., Kamakura, T., Komba, S., Nakai, H., Taguchi, H.: J. Biol. Chem., 294, 7942 (2019).
11) Nakajima, M.: Biologia, 78, 1741 (2023).
12) Motouchi, S., Kobayashi, K., Nakai, H., Nakajima, M.: Commun. Biol., 6, 961 (2023).
13) Rigano, L. A., Payette, C., Brouillard, G., Marano, M. R., Abramowicz, L., Torres, P. S., Yun, M., Castagnaro, A. P., Oirdi, M. E., Dufour, V., Malamud, F., Dow, J. M., Bouarab, K., Vojnov, A. A.: Plant Cell, 19, 2077 (2007).
14) Motouchi, S., Komba, S., Nakai, H., Nakajima, M.: J. Am. Chem. Soc., 146, 17738 (2024).
15) Jung, W. S., Hong, C. K., Lee, S., Kim, C. S., Kim, S. J., Kim, S. I., Rhee, S.: J. Biol. Chem., 282, 8414 (2007).
16) Neelamegham, S., Aoki-Kinoshita, K., Bolton, E., Frank, M., Lisacek, F., Lütteke, T., O’Boyle, N., Packer, N. H., Stanley, P., Toukach, P., Varki, A., Woods, R. J., Darvill, A., Dell, A., Henrissat, B., Bertozzi, C., Hart, G., Narimatsu, H., Freeze, H., Yamada, I., Paulson, J., Prestegard, J., Marth, J., Vliegenthart, J. F. G., Etzler, M., Aebi, M., Kanehisa, M., Taniguchi, N., Edwards, N., Rudd, P., Seeberger, P., Mazumder, R., Ranzinger, R., Cummings, R., Schnaar, R., Perez, S., Kornfeld, S., Kinoshita, T., York, W., Knirel, Y.: Glycobiology, 29, 620 (2019).
17) Mehta, A. Y., Cummings, R. D.: Bioinformatics, 36, 3613 (2020).